Schaar-steen-papier met bacteriën
- JanBaetens
Een groep wetenschappers houdt een toernooi schaar-steen-papier. Het klinkt als het begin van een mop, maar is de realiteit. Dit doen ze echter niet op de normale manier, met handgebaren tegen elkaar. Dit doen ze op hun supercomputer, miljoenen keren opnieuw. Maar waarom houden ze zich bezig met dit kinderspel?
De paradox van de plankton
De klassieke ecologie volgt het principe van wederzijdse uitsluiting. Dit principe zegt dat soorten die in volledige competitie zijn, die met andere woorden strijden voor exact dezelfde hulpbronnen, niet kunnen samenleven. Eén van de twee soorten zal een klein voordeel hebben ten opzichte van de andere. Zo kan één soort zich bijvoorbeeld iets sneller voortplanten of efficiënter gebruikmaken van het aanwezige voedsel. Deze soort zal de andere wegconcurreren en als enige overleven. Op basis van dit principe verwachten we dus geen rijkelijke hoeveelheid aanwezige soorten.
In de praktijk blijkt dit echter niet zo te zijn. Zo vindt men in planktonkolonies veel meer soorten dan men zou verwachten. Deze tegenstrijdigheid tussen theorie en de praktijk staat daarom bekend als de ‘paradox van de plankton’, en vindt men in vele ecosystemen, van graslanden tot bacteriepopulaties. Het blijft een mysterie dat tot op vandaag slechts deels is verklaard.
Het principe van wederzijdse uitsluiting gaat ervan uit dat iedereen even vaak kan interageren met iedereen. Dit is echter niet altijd het geval. In een ruimtelijk ingedeeld ecosysteem, dat dus niet perfect gemengd is, zal je eerder met je buur in interactie treden dan met iemand ver weg. Dit blijkt een belangrijk effect te hebben op biodiversiteit. Experimenten tonen aan dat meerdere competitieve soorten kunnen samenleven als het ecosysteem niet perfect gemengd is. Dit fenomeen verklaart dus deels de paradox van de plankton en verdient daarom verder onderzoek. Mijn onderzoek richtte zich hierbij op bacteriekolonies.
Model
Ik onderzocht deze dynamiek met behulp van een zogenaamd individu-gebaseerd model. Hierbij laat men virtuele bacteriën los in een gesimuleerde ruimte. In mijn model wordt elke bacterie voorgesteld als een cirkel op een bepaalde positie in de kleur van één van de drie soorten: rood, geel en blauw. Doorheen de tijd kunnen deze bacteriën drie verschillende interacties uitvoeren. Allereerst kunnen ze kiezen om te migreren, waarbij ze een stap zetten in willekeurige richting. De bacteriën kunnen er ook voor kiezen om zich voort te planten. Dit kan als er voldoende vrije ruimte is rondom hen. De bacterie splitst zich dan in twee dochterbacteriën.
Als laatste kunnen de bacteriën ervoor kiezen om met elkaar in gevecht te gaan. Een bacterie van de blauwe soort overwint een gele bacterie. Een gele bacterie zal een rode overwinnen, en deze laatste overwint dan weer een blauwe bacterie. Dit competitienetwerk is gelijkaardig aan het klassieke schaar-steen-papier spel. Het is bovendien het kleinst mogelijke netwerk waarbij geen enkele soort een absoluut voordeel heeft. Daarom is het een goede vereenvoudiging om competitie in echte ecosystemen voor te stellen.
Nieuwe inzichten
Door nu een groot aantal individuele bacteriën te simuleren kan men kijken hoe het ecosysteem evolueert. Zo kan je als het ware uitzoomen en kijken wat er gebeurt op macroscopische schaal. Dit simpel model kan je nu gebruiken om theoretische vragen te beantwoorden over biodiversiteit. Zo kunnen we nagaan hoe het komt dat soorten kunnen samenleven in een ruimtelijk ecosysteem, ondanks het principe van wederzijdse uitsluiting.
Het blijkt dat de soorten zichzelf organiseren in ruimtelijke structuren. Een gebalanceerde achtervolging ontstaat, waarbij elke soort terrein wint aan de kant van zijn prooi en tegelijkertijd terrein verliest aan de kant van zijn aanvaller. Door deze ruimtelijke ordening is het mogelijk dat meerdere competitieve soorten samenleven. Door verder te experimenteren met het model kunnen we nauwkeurig bepalen wat de belangrijkste effecten zijn op biodiversiteit. Dit model maakt het dus mogelijk om te kijken hoe een ecosysteem theoretisch zal evolueren. Zo kunnen we nieuwe inzichten krijgen in allerhande fenomenen. Als voorbeeld geef ik het effect van achtervolgen en vluchten.
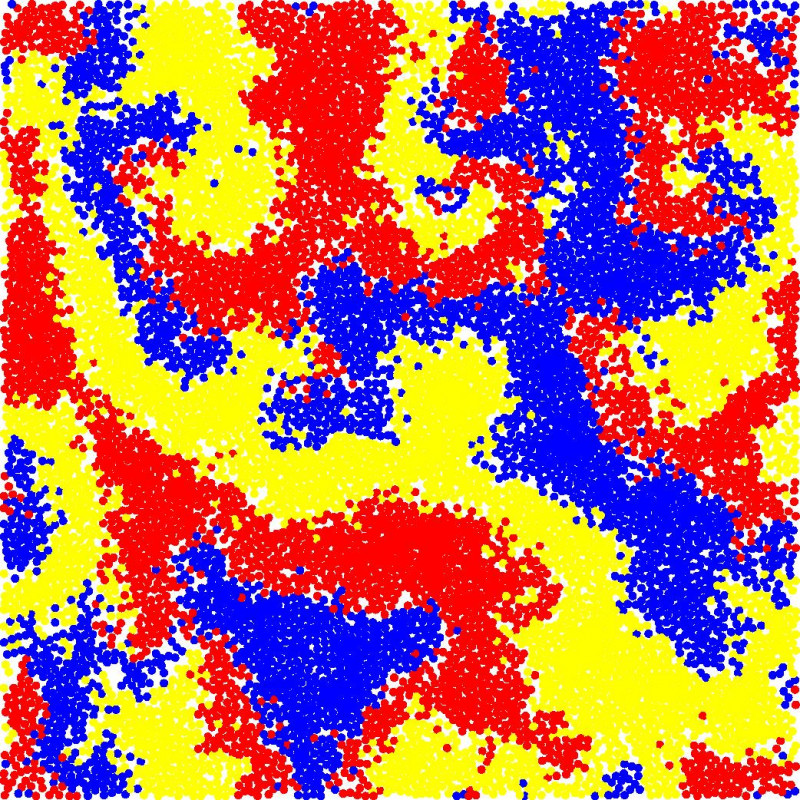
Vluchten en achtervolgen
Het is bekend dat sommige bacteriën hun prooi actief achtervolgen, terwijl anderen wegvluchten van hun aanvaller. Door een kleine aanpassing aan het model kunnen we het effect hiervan onderzoeken. Wanneer alle soorten gemengd zijn blijkt achtervolgen een slechte strategie te zijn. Door te achtervolgen verplaats je jezelf en vergroot de kans dat je één van jouw aanvallers tegenkomt. De kans dat je zelf aangevallen wordt en vervolgens sterft is daardoor groter. Wanneer alle soorten in ruimtelijke structuren zijn geordend is aanvallen wel een goede strategie. Door de ruimtelijke scheiding zal je prooi zich immers niet vlak naast jouw aanvaller bevinden. Je kan je prooi doden en ruimte vrijmaken om in voor te planten, zonder dat je het gevaar loopt zelf aangevallen te worden.
Op gelijkaardige manier kunnen we wegvluchten onderzoeken. Net zoals bij aanvallen blijkt dat wegvluchten een slechte strategie is als alle soorten gemengd zijn. Tijdens je vlucht verhoogt de kans om een andere aanvaller tegen te komen, en dus de kans dat je wordt vermoord. Wanneer ruimtelijke structuren aanwezig zijn is het beeld echter genuanceerder. Wegvluchten wanneer je wordt achtervolgd is een goede strategie. Je verlaat immers terrein waar je aanvaller naartoe beweegt. Zo vermijd je dat je wordt vermoord. Wegvluchten van een aanvaller die je niet achtervolgt is echter geen goede tactiek. Je verlaat terrein waar je aanvaller niet actief naar onderweg is. Hierdoor creëer je open ruimte waar je vijand in kan reproduceren. Je aanvaller groeit in aantal, wat een nadeel is voor jou.
Algemeen model
Gewapend met dit en andere individu-gebaseerde modellen kunnen wetenschappers nu een hele reeks vragen beantwoorden. Of het nu gaat over grote zoogdieren of kleine bacteriën, deze klasse van modellen geeft ons de mogelijkheid om complexe ecosystemen te onderzoeken, ermee te experimenteren en waardevolle inzichten te verwerven. Zo gebruikte ik dit model om onder andere na te gaan wanneer achtervolgen of wegvluchten een goede strategie is. Om een bekende dichter verkeerd te citeren: ‘to flee or not to flee, that’s no longer the question’.
** Een animatie is beschikbaar op: https://filesender.belnet.be/?vid=08c502ff-c4a0-1bc8-5384-00003eea3d6a **
Bibliografie
Abrudan, M., You, L., St, K., and Thuijsman, F. (2016). A Game Theoretical Approach to Microbial Coexistence. In Thuijsman, F. and Wagener, F., editors, Advances in Dynamic and Evolutionary Games, chapter 13, pages 267–282. Springer International Publishing, Switzerland.
Achtman, M., Mercer, A., Kusecek, B., Pohl, A., Aaronson, W., Sutton, A., and Silver, P. (1983). Six widespread bacterial clones among Escherichia coli K1 isolates. Infection and Immunity, 39(1):315–335.
Adamson, M. W. and Morozov, A. Y. (2012). Revising the Role of Species Mobility in Maintaining Biodiversity in Communities with Cyclic Competition. Bulletin of Mathematical Biology, 74(9):2004–2031.
Allesina, S. and Levine, J. M. (2011). A competitive network theory of species diversity. Proceedings of the National Academy of Sciences of the United States of America, 108(14):5638–5642.
An, D., Danhorn, T., Fuqua, C., and Parsek, M. R. (2006). Quorum sensing and motility mediate interactions between Pseudomonas aeruginosa and Agrobacterium tumefaciens in biofilm cocultures. Proceedings of the National Academy of Sciences, USA, 103(10):3828– 3833.
Arnoldi, M., Kacher, C. M., B¨auerlein, E., Radmacher, M., and Fritz, M. (1998). Elastic properties of the cell wall of Magnetospirillum gryphiswaldense investigated by atomic forcemicroscopy. Applied Physics A: Materials Science and Processing, 66(SUPPL. 1):613– 617.
Ashby, M. N., Rine, J., Mongodin, E. F., Nelson, K. E., and Dimster-Denk, D. (2007). Serial analysis of rRNA genes and the unexpected dominance of rare members of microbial communities. Applied and Environmental Microbiology, 73(14):4532–4542.
Avelino, P. P., Bazeia, D., Losano, L., Menezes, J., and Oliveira, B. F. (2012). Junctions and spiral patterns in generalized rock-paper-scissors models. Physical Review E - Statistical, Nonlinear, and Soft Matter Physics, 86(3):2–6.
Beaumont, M. A. (2010). Approximate Bayesian Computation in Evolution and Ecology. Annual Review of Ecology, Evolution, and Systematics, 41(1):379–406.
Bell, T., Newman, J. A., Silverman, B. W., Turner, S. L., and Lilley, A. K. (2005). The contribution of species richness and composition to bacterial services. Nature, 436(7054):1157– 1160.
Ben-Jacob, E., Schochet, O., Tenenbaum, A., Cohen, I., Czir´ok, A., and Vicsek, T. (1994). Generic modelling of cooperative growth patterns in bacterial colonies. Nature, 368(6466):46– 49.
Berr, M., Reichenbach, T., Schottenloher, M., and Frey, E. (2009). Zero-one survival behavior of cyclically competing species. Physical Review Letters, 102(4):1–4.
Buss, L. W. (1979). Competitive Networks : Nontransitive Competitive Relationships in Cryptic Coral Reef Environments. The American Naturalist, 113(2):223–234.
Buss, L. W. (1990). Competition within and between encrusting clonal invertebrates. Trends in Ecology and Evolution, 5(11):352–356.
Cabeen, M. T. and Jacobs-Wagner, C. (2005). Bacterial cell shape. Nature Reviews Microbiology, 3(8):601–610.
Cardinale, B. J., Palmer, M. A., and Collins, S. L. (2002). Species diversity enhances ecosystem functioning through interspecific facilitation. Nature, 415(6870):426–429.
Chao, L. and Levin, B. R. (1981). Structured habitats and the evolution of anticompetitor toxins in bacteria. Proceedings of the National Academy of Sciences of the United States of America, 78(10):6324–6328.
Cheng, H., Yao, N., Huang, Z.-G., Park, J., Do, Y., and Lai, Y.-C. (2014). Mesoscopic Interactions and Species Coexistence in Evolutionary Game Dynamics of Cyclic Competitions. Scientific Reports, 4:7486.
Chesson, P. (2000). Mechanisms of maintentance of species diversity. Annual Review of Ecology and Systematics, 31(1):343–366.
Csill´ery, K., Blum, M. G. B., Gaggiotti, O. E., and Francois, O. (2010). Approximate Bayesian Computation (ABC) in practice. Trends in Ecology and Evolution, 25(7):410–418.
Cz´ar´an, T. L., Hoekstra, R. F., and Pagie, L. (2002). Chemical warfare between microbes promotes biodiversity. Proceedings of the National Academy of Sciences of the United States of America, 99(2):786–790. 68
Daly, A. J., Baetens, J. M., and De Baets, B. (2015). The impact of initial evenness on biodiversity maintenance for a four-species in silico bacterial community. Journal of Theoretical Biology, 387:189–205.
Daly, A. J., Baetens, J. M., and De Baets, B. (2016). In silico substrate dependence increases community productivity but threatens biodiversity. Physical Review E, 93(4):042414.
De Roy, K., Marzorati, M., Negroni, A., Thas, O., Balloi, A., Fava, F., Verstraete, W., Daffonchio, D., and Boon, N. (2013). Environmental conditions and community evenness determine the outcome of biological invasion. Nature Communications, 4:1383.
De Roy, K., Marzorati, M., Van den Abbeele, P., Van de Wiele, T., and Boon, N. (2014). Synthetic microbial ecosystems: an exciting tool to understand and apply microbial communities. Environmental Microbiology, 16(6):1472–1481.
Descamps-Julien, B. and Gonzalez, A. (2005). Stable coexistence in a fluctuating environment: An experimental demonstration. Ecology, 86(10):2815–2824.
Diggle, S. P., Griffin, A. S., Campbell, G. S., and West, S. A. (2007). Cooperation and conflict in quorum-sensing bacterial populations. Nature, 450(7168):411–414.
Domach, M. M., Leung, S. K., Cahn, R. E., Cocks, G. G., and Shuler, M. L. (1984). Computer model for Glucose-limited growth of a single cell of Escherichia Coli. Biotechnology and Bioengineering, 26(3):203–216.
Durrett, R. and Levin, S. A. (1997). Allelopathy in Spatially Distributed Populations. Journal of Theoretical Biology, 185(2):165–171.
Durrett, R. and Levin, S. A. (1998). Spatial aspects of interspecific competition. Theoretical population biology, 53(1):30–43.
Dusenbery, D. B. (2009). Living at Micro Scale: The Unexpected Physics of Being Small. Harvard University Press.
Ermentrout, G. and Edelstein-Keshet, L. (1993). Cellular Automata Approaches to Biological Modeling. Journal of Theoretical Biology, 160:97–133.
Evans, M. R., Grimm, V., Johst, K., Knuuttila, T., de Langhe, R., Lessells, C. M., Merz, M., O’Malley, M. A., Orzack, S. H., Weisberg, M., Wilkinson, D. J., Wolkenhauer, O., and Benton, T. G. (2013). Do simple models lead to generality in ecology? Trends in Ecology and Evolution, 28(10):578–583.
Ferrer, J., Prats, C., and L´opez, D. (2008). Individual-based modelling: An essential tool for microbiology. Journal of Biological Physics, 34:19–37. 69
Fink, K. A. and Wilson, S. D. (2011). Invasion of a Native Grassland: Diversity and Resource Reduction. Botany, 89(3):157–164.
Flannagan, R. S., Valvano, M. A., and Koval, S. F. (2004). Downregulation of the motA gene delays the escape of the obligate predator Bdellovibrio bacteriovirus 109J from bdelloplasts of bacterial prey cells. Microbiology, 150(3):649–656.
Frean, M. and Abraham, E. R. (2001). Rock-scissors-paper and the survival of the weakest. Proceedings. Biological sciences / The Royal Society, 268(1474):1323–7.
Frey, E. and Reichenbach, T. (2011). Bacterial games. Springer, Berlin Heidelberg.
Gause, F. (1934). The Struggle for Existence. Hafner Publishing Company, New York.
Gillespie, D. T. (1976). A general method for numerically simulating the stochastic time evolution of coupled chemical reactions. Journal of Computational Physics, 22(4):403–434.
Ginovart, M. (2002). INDISIM, An Individual-based Discrete Simulation Model to Study Bacterial Cultures. Journal Of Theoretical Biology, 214(2):305–319.
Grimm, V. (1999). Ten years of individual-based modelling in ecology: What have we learned and what could we learn in the future? Ecological Modelling, 115(2-3):129–148.
Grimm, V., Berger, U., Bastiansen, F., Eliassen, S., Ginot, V., Giske, J., Goss-Custard, J., Grand, T., Heinz, S. K., Huse, G., Huth, A., Jepsen, J. U., Jørgensen, C., Mooij, W. M., M¨uller, B., Pe’er, G., Piou, C., Railsback, S. F., Robbins, A. M., Robbins, M. M., Rossmanith, E., R¨uger, N., Strand, E., Souissi, S., Stillman, R. A., Vabø, R., Visser, U., and DeAngelis, D. L. (2006). A standard protocol for describing individual-based and agent-based models. Ecological Modelling, 198(1-2):115–126.
Grimm, V., Berger, U., DeAngelis, D. L., Polhill, J. G., Giske, J., and Railsback, S. F. (2010). The ODD protocol: A review and first update. Ecological Modelling, 221(23):2760–2768.
Grman, E., Lau, J. A., Schoolmaster, D. R., and Gross, K. L. (2010). Mechanisms contributing to stability in ecosystem function depend on the environmental context. Ecology Letters, 13(11):1400–1410.
Hales, D., Rouchier, J., and Edmonds, B. (2003). Model-To-Model Analysis. Journal of Artificial Societies and Social Simulation, 6(4):5.
Hardin, G. (1960). The competitive exclusion principle. Science, 131:1292–1297.
Harpole, W. S. and Suding, K. N. (2007). Frequency-dependence stabilizes competitive interactions among four annual plants. Ecology Letters, 10(12):1164–1169. 70
Hassell, M. P., Comins, H. N., and May, R. M. (1994). Species coexitence and self-organizing spatial dynamics. Nature, 370(6487):290–292.
Hawick, K. A. (2011). Cycles, diversity and competition in rock-paper-scissors-lizard-spock spatial game agent simulations. Proceedings of the 2011 International Conference on Artificial Intelligence, 1:115–121.
Heinz, T. N. and Hunenberger, P. H. (2004). A fast pairlist-construction algorithm for molecular simulations under periodic boundary conditions. Journal of Computational Chemistry, 25(12):1474–1486.
Hellweger, F. L. and Bucci, V. (2009). A bunch of tiny individuals-Individual-based modeling for microbes. Ecological Modelling, 220(1):8–22.
Hellweger, F. L., Clegg, R. J., Clark, J. R., Plugge, C. M., and Kreft, J.-U. (2016). Advancing microbial sciences by individual-based modelling. Nature Reviews Microbiology, 14(7):461– 471.
Henke, J. M. and Bassler, B. L. (2004). Bacterial social engagements. Trends in Cell Biology, 14(11):648–656.
Hibbing, M. E., Fuqua, C., Parsek, M. R., and Peterson, S. B. (2010). Bacterial competition: surviving and thriving in the microbial jungle. Nature Reviews. Microbiology, 8(1):15–25.
Hillebrand, H., Bennett, D. M., and Cadotte, M. W. (2008). Consequences of dominance: a review of evenness effect on local and regional ecosystem processes. Ecology, 89(6):1510–1520.
Holcombe, M., Adra, S., Bicak, M., Chin, S., Coakley, S., Graham, A. I., Green, J., Greenough, C., Jackson, D., Kiran, M., MacNeil, S., Maleki-Dizaji, A., McMinn, P., Pogson, M., Poole, R., Qwarnstrom, E., Ratnieks, F., Rolfe, M. D., Smallwood, R., Sun, T., and Worth, D. (2012). Modelling complex biological systems using an agent-based approach. Integrative Biology, 4(November):53–64.
Hooper, D. U., Chapin, F. S., Ewel, J. J., Hextor, A., Inchausti, P., Lavorel, S., Lawton, J. H., Lodge, D. M., Loreau, M., Naeem, S., Schmid, B., Setala, H., Symstad, J., Vandermeer, J., and Wardle, D. A. (2005). Effects of biodiversity on ecosystem functioning: a concensus of current knowledge. America, 75(July 2004):3–35.
Huisman, J., Johansson, A. M., Folmer, E. O., and Weissing, F. J. (2001). Towards a solution of the plankton paradox: The importance of physiology and life history. Ecology Letters, 4(5):408–411.
Huisman, J. and Weissing, F. J. (1999). Biodiversity of plankton by species oscillations and chaos. Nature, 402(6760):407–410. 71
Hutchinson, G. . E. . (1961). The Paradox of the Plankton. The American Naturalist, 95(882):137–145.
Isbell, F. I., Polley, H. W., and Wilsey, B. J. (2009). Species interaction mechanisms maintain grassland plant species diversity. Ecology, 90(7):1821–1830.
Jackson, J. B. C. (1983). Biological determinants of present and past sessile animal distributions. In Tevesz, M. J. S. and McCall, P. L., editors, Biotic interactions in recent and fossil benthic communities., chapter 2, pages 39–120. Springer US.
James, R., Lazdunski, C., and Pattus, F. (2013). Bacteriocins, microcins and lantibiotics, volume 65. Springer Science & Business Media.
Jeong, J. W., Snay, J., and Ataai, M. M. (1990). A mathematical model for examining growth and sporulation processes of Bacillus subtilis. Biotechnology and Bioengineering, 35(2):160–184.
Kang, Y., Pan, Q., Wang, X., and He, M. (2013). A golden point rule in rock-paper-scissorslizard-spock game. Physica A: Statistical Mechanics and its Applications, 392(11):2652–2659.
Karolyi, G., Neufeld, Z., and Scheuring, I. (2005). Rock-scissors-paper game in a chaotic flow: The effect of dispersion on the cyclic competition of microorganisms. Journal of Theoretical Biology, 236(1):12–20.
Keddy, P. (2001). Competition (2nd edition). Kluwer, Dordrecht.
K´efi, S., Rietkerk, M., Alados, C. L., Pueyo, Y., Papanastasis, V. P., ElAich, A., and de Ruiter, P. C. (2007). Spatial vegetation patterns and imminent desertification in Mediterranean arid ecosystems. Nature, 449(7159):213–7.
Kennedy, T. A., Naeem, S., Howe, K. M., Knops, J. M. H., Tilman, D., and Reich, P. (2002). Biodiversity as a barrier to ecological invasion. Nature, 417(6889):636–638.
Kerr, B., Riley, M. A., Feldman, M. W., and Bohannan, B. J. M. (2002). Local dispersal promotes biodiversity in a real-life game of rock-paper-scissors. Nature, 418(6894):171–174.
Kinkel, L. L., Schlatter, D. C., Xiao, K., and Baines, A. D. (2014). Sympatric inhibition and niche differentiation suggest alternative coevolutionary trajectories among Streptomycetes. The ISME Journal, 8(2):249–56.
Kirkup, B. C. and Riley, M. A. (2004). Antibiotic-mediated antagonism leads to a bacterial game of rock-paper-scissors in vivo. Nature, 428(6981):412–4.
Klapper, I. and Dockery, J. (2010). Mathematical Description of Microbial Biofilm. SIAM Review, 52(2):221–265. 72
Kreft, J. U., Booth, G., and Wimpenny, J. W. T. (1998). BacSim, a simulator for individualbased modelling of bacterial colony growth. Microbiology, 144(12):3275–3287.
Kreft, J. U., Plugge, C. M., Grimm, V., Prats, C., Leveau, J. H. J., Banitz, T., Baines, S., Clark, J., Ros, A., Klapper, I., Topping, C. J., Field, A. J., Schuler, A., Litchman, E., and Hellweger, F. L. (2013). Mighty small: Observing and modeling individual microbes becomes big science. Proceedings of the National Academy of Sciences of the United States of America, 110(45):18027–18028.
Laird, R. A. (2014). Population interaction structure and the coexistence of bacterial strains playing ’rock-paper-scissors’. Oikos, 123(4):472–480.
Laird, R. A. and Schamp, B. S. (2006). Competitive intransitivity promotes species coexistence. The American Naturalist, 168(2):182–193.
Laird, R. A. and Schamp, B. S. (2008). Does local competition increase the coexistence of species in intransitive networks? Ecology, 89(1):237–247.
Laird, R. A. and Schamp, B. S. (2009). Species coexistence, intransitivity, and topological variation in competitive tournaments. Journal of Theoretical Biology, 256(1):90–95.
Laird, R. A. and Schamp, B. S. (2015). Competitive intransitivity, population interaction structure, and strategy coexistence. Journal of Theoretical Biology, 365:149–158.
Landau, L. and Lifshitz, E. (1986). EM Lifshitz Theory of elasticity. Pergamon Press, 3 edition. Lankau, R. A. and Strauss, S. Y. (2007). Genetic and Species Diversity in a Plant Community. Science, 317(5844):1561–1563.
Lemieux, J. and Cusson, M. (2014). Effects of habitat-forming species richness, evenness, identity, and abundance on benthic intertidal community establishment and productivity. PLoS ONE, 9(10).
Levin, B. R., Antonovics, J., and Sharma, H. (1988). Frequency-dependent selection in bacterial populations [and discussion]. Philosophical Transactions of the Royal Society B: Biological Sciences, 319(1196):459–472.
Levin, S. A. (1974). Dispersion and Population Interactions. The American Naturalist, 108(960):207–228.
Levine, J. M. and D’Antonio, C. M. (1999). Elton Revisited : A Review of Evidence Linking Diversity and Invasibility. Nordic Society Oikos, 87(1):15–26.
Matsushita, M. and Fujikawa, H. (1990). Diffusion-limited growth in bacterial colony formation. Physica A: Statistical Mechanics and its Applications, 168(1):498–506. 73
Mattson, W. and Rice, B. M. (1999). Near-neighbor calculations using a modified cell-linked list method. Computer Physics Communications, 119(2):135–148.
May, R. M. and Leonard, W. J. (1975). Nonlinear Aspects of Competition Between Three Species Author. ociety for Industrial and Applied Mathematics, 29(2):243–253.
Michaelis, L., Menten, M. L., Goody, R. S., and Johnson, K. A. (1913). Die Kinetik der Invertinwirkung The Kinetics of Invertase Action translated by. Biochem., 49:333–369.
Miller, M. B. and Bassler, B. L. (2001). Quorum sensing in bacteria. Annual Review of Microbiology, 55(1):165–199.
Mimura, M. and Tohma, M. (2015). Dynamic coexistence in a three-species competitiondiffusion system. Ecological Complexity, 21:215–232.
Mirams, G. R., Arthurs, C. J., Bernabeu, M. O., Bordas, R., Cooper, J., Corrias, A., Davit, Y., Dunn, S. J., Fletcher, A. G., Harvey, D. G., Marsh, M. E., Osborne, J. M., Pathmanathan, P., Pitt-Francis, J., Southern, J., Zemzemi, N., and Gavaghan, D. J. (2013). Chaste: An Open Source C++ Library for Computational Physiology and Biology. PLoS Computational Biology, 9(3).
Miyazaki, T., Tainaka, K. I., Togashi, T., Suzuki, T., and Yoshimura, J. (2006). Spatial coexistence of phytoplankton species in ecological timescale. Population Ecology, 48(2):107– 112.
Monod, J. (1949). The Growth of Bacterial Cultures. Annual Review of Microbiology, 3(1):371–394.
Naeem, S. (2009). Ecology: Gini in the bottle. Nature, 458(7238):579–580.
Naeem, S. and Li, S. (1997). Biodiversity enhances ecosystem reliability. Nature, 390(6659):507– 509.
Narisawa, N., Haruta, S., Arai, H., Ishii, M., and Igarashi, Y. (2008). Coexistence of antibioticproducing and antibiotic-sensitive bacteria in biofilms is mediated by resistant bacteria. Applied and Environmental Microbiology, 74(12):3887–3894.
Nisbet, R. M., Martin, B. T., and de Roos, A. M. (2016). Integrating ecological insight derived from individual-based simulations and physiologically structured population models. Ecological Modelling, 326:101–112.
Perc, M., Szolnoki, A., and Szab´o, G. (2007). Cyclical interactions with alliance-specific heterogeneous invasion rates. Physical Review E - Statistical, Nonlinear, and Soft Matter Physics, 75(5):3–6. 74
Petraitis, P. S. (1979). Competitive networks and measures of intransitivity. The American Naturalist, 114(6):921–925.
Picioreanu, C., Loosdrecht, M., and Heijnen, J. J. (1998a). Mathematical Modeling of Biofilm Strucutre with a Hybrid Differential-Discrete Cellular Automata Approach. Biotechnology and Bioengineering, 58(November):101–116.
Picioreanu, C., van Loosdrecht, M. C. M., and Heijnen, J. J. (1998b). A new combined differential discrete cellular automaton approach for biofilm modelling Application for growth in gel beads. Biotechnology and Bioengineering, 58(March):101–116.
Polley, H. W., Wilsey, B. J., and Derner, J. D. (2003). Do species evenness and plant density influence the magnitude of selection and complementarity effects in annual plant species mixtures? Ecology Letters, 6(3):248–256.
Pop lawski, N. J., Shirinifard, A., Swat, M., and Glazier, J. A. (2008). Simulation of singlespecies bacterial-biofilm growth using the Glazier-Graner-Hogeweg model and the CompuCell3D modeling environment. Mathematical Biosciences and Engineering, 5(2):355–88.
Power, K. and Marshall, K. C. (1988). Cellular growth and reproduction of marine bacteria on surfacebound substrate. Biofouling, 1(2):163–174. Proulx, R., Wirth, C., Voigt, W., Weigelt, A., Roscher, C., Attinger, S., Baade, J., Barnard, R. L., Buchmann, N., Buscot, F., Eisenhauer, N., Fischer, M., Gleixner, G., Halle, S., Hildebrandt, A., Kowalski, E., Kuu, A., Lange, M., Milcu, A., Niklaus, P. A., Oelmann, Y., Rosenkranz, S., Sabais, A., Scherber, C., Scherer-Lorenzen, M., Scheu, S., Schulze, E. D., Schumacher, J., Schwichtenberg, G., Soussana, J. F., Temperton, V. M., Weisser, W. W., Wilcke, W., and Schmid, B. (2010). Diversity promotes temporal stability across levels of ecosystem organization in experimental grasslands. PLoS ONE, 5(10):1–8.
Reichenbach, T., Mobilia, M., and Frey, E. (2007). Mobility promotes and jeopardizes biodiversity in rock-paper-scissors games. Nature, 448(7157):1046–1049.
Reichenbach, T., Mobilia, M., and Frey, E. (2008). Self-organization of mobile populations in cyclic competition. Journal of Theoretical Biology, 254(2):368–383.
Reusch, T. B. H., Ehlers, A., Hammerli, A., and Worm, B. (2005). Ecosystem recovery after climatic extremes enhanced by genotypic diversity. Proceedings of the National Academy of Sciences, 102(8):2826–2831.
Richerson, P., Armstrong, R., and Goldman, C. R. (1970). Contemporaneous disequilibrium, a new hypothesis to explain the ”paradox of the plankton”. Proceedings of the National Academy of Sciences of the United States of America, 67(4):1710–1714. 75
Riley, M. A. and Gordon, D. M. (1992). A survey of Col plasmids in natural isolates of Escherichia coli and an investigation into the stability of Col-plasmid lineages. Journal of General Microbiology, 138(7):1345–1352.
Riley, M. A. and Gordon, D. M. (1999). The ecological role of bacteriocins in bacterial competition. Trends in Microbiology, 7(3):129–133.
Rohani, P., Lewis, T. J., Gr¨unbaum, D., and Ruxton, G. D. (1997). Spatial self-organization in ecology: Pretty patterns or robust reality? Trends in Ecology and Evolution, 12(2):70–74.
Rojas-Echenique, J. and Allesina, S. (2011). Interaction rules affect species coexistence in intransitive networks. Ecology, 92(5):1174–1180.
Roman, A., Dasgupta, D., and Pleimling, M. (2016). A theoretical approach to understand spatial organization in complex ecologies. ArXiv, 403:10–16.
Rosenzweig, M. L. and MacArthur, R. H. (1963). Graphical Representation and Stability Conditions of Predator-Prey. The American Naturalist, 97(895):209–223.
Rousseau, R. and Van Hecke, P. (1999). Measuring Biodiversity. Acta Biotheoretica, 47:1–5.
Roy, S. and Chattopadhyay, J. (2007). Towards a resolution of ’the paradox of the plankton’: A brief overview of the proposed mechanisms. Ecological Complexity, 4(1-2):26–33.
Rudge, T. J., Steiner, P. J., Phillips, A., and Haseloff, J. (2012). Computational modeling of synthetic microbial biofilms. ACS Synthetic Biology, 1(8):345–352.
Sale, A. U. (2007). Getting the measure of well-being. Community Care, 405(1691):16–17.
Sankaran, M. and McNaughton, S. J. (1999). Determinants of biodiversity regulate compositional stability of communities. Nature, 401(6754):691–693.
Scanlon, T. M., Caylor, K. K., Levin, S. A., and Rodriguez-Iturbe, I. (2007). Positive feedbacks promote power-law clustering of Kalahari vegetation. Nature, 449(7159):209–212.
Scheffer, M., Baveco, J. M., Deangelis, D. L., Rose, K. A., and Vannes, E. H. (1995). SuperIndividuals A Simple Solution For Modeling Large Populations On An Individual Basis. Ecological Modelling, 80(1995):161–170.
Scheffer, M., Rinaldi, S., Huisman, J., and Weissing, F. J. (2003). Why plankton communities have no equilibrium: Solutions to the paradox. Hydrobiologia, 491:9–18.
Shapiro, J. A. (1998). Thinking about bacterial populations as multicellular organisms. Annual Review of Microbiology, 52:81–104. 76
Sinervo, B. R., Heulin, B., Surget-Groba, Y., Clobert, J., Miles, D. B., Corl, A., Chaine, A. S., and Davis, A. R. (2007). Models of Density-Dependent Genic Selection and a New Rock-Paper-Scissors Social System. The American Naturalist, 170(5):663–680.
Smith, V. H. (2002). Effects of resource supplies on the stucture and function of microbial communities. Antonie van Leeuwenhoek, 81:99–106.
Standaert, A. R., Poschet, F., Geeraerd, A. H., Uylbak, F. V., Kreft, J. U., and Van Impe, J. F. (2004). A novel class of predictive microbial growth models: implementation in an individual-based framework. Computer Applications in Biotechnology, pages 183–188.
Szab´o, G. and F´ath, G. (2007). Evolutionary games on graphs. Physics Reports, 446(4-6):97– 216.
Szolnoki, A., Mobilia, M., Jiang, L. L., Szczesny, B., Rucklidge, A. M., and Perc, M. (2014). Cyclic dominance in evolutionary games: a review. Journal of the Royal Society, Interface / the Royal Society, 11(100):20140735.
Taylor, D. R. and Aarssen, L. W. (1990). Complex Competitive Relationships Among Genotypes of Three Perennial Grasses : Implications for Species Coexistence. The American Society of Naturalist, 136(3):305–327.
Tilman, D. (1996). Biodiversity: population versus ecosystem stability. Ecological Society of America, 77(2):350–363.
Tilman, D., Knops, J. M. H., Wedin, D., Reich, P., Ritchie, M., and Siemann, E. (1997). The Influence of Functional Diversity and Composition on Ecosystem Processes. Science, 277(5330):1300–1302.
Touhami, A., Nysten, B., and Dufrene, Y. (2003). Nanoscale Mapping of the Elasticity of Microbial Cells by Atomic Force Microscopy Nanoscale Mapping of the Elasticity of Microbial Cells by Atomic Force Microscopy. Langmuir, (19):4539–4543.
Tracy, B. F. and Sanderson, M. A. (2004). Forage productivity, species evenness and weed invasion in pasture communities. Agriculture, Ecosystems and Environment, 102(2):175–183.
van der Vaart, E., Beaumont, M. A., Johnston, A. S., and Sibly, R. M. (2015). Calibration and evaluation of individual-based models using Approximate Bayesian Computation. Ecological Modelling, 312:182–190.
Verlet, L. (1967). Computer ”Experiments” on Classical Fluids. Physical Review, 159(1):98– 103.
Vetsigian, K., Jajoo, R., and Kishony, R. (2011). Structure and evolution of streptomyces interaction networks in soil and in silico. PLoS Biology, 9(10). 77
Vukov, J., Szolnoki, A., and Szab´o, G. (2013). Diverging fluctuations in a spatial five-species cyclic dominance game. Physical Review E - Statistical, Nonlinear, and Soft Matter Physics, 88(2):1–8.
Waters, C. M. and Bassler, B. L. (2005). Quorum Sensing : Communication in Bacteria. Annual Reviews in Cell Development Biology, 21(1):319–346.
Wessel, A. K., Hmelo, L., Parsek, M. R., and Whiteley, M. (2013). Going local: technologies for exploring bacterial microenvironments. Nature reviews. Microbiology, 11(5):337–48.
Wiggert, J. D., Haskell, A. G. E., Paffenh¨ofer, G. A., Hofmann, E. E., and Klinck, J. M. (2005). The role of feeding behavior in sustaining copepod populations in the tropical ocean. Journal of Plankton Research, 27(10):1013–1031.
Wilensky, U. (1999). Netlogo. Wills, C., Harms, K. E., Condit, R., King, D., Thompson, J., He, F., Muller-Landau, H. C., Ashton, P., Losos, E., Comita, L., Hubbell, S., LaFrankie, J., Bunyavejchewin, S., Dattaraja, H. S., Davies, S., Esufali, S., Foster, R., Gunatilleke, N., Gunatilleke, S., Hall, P., Itoh, A., John, R., Kiratiprayoon, S., de Lao, S. L., Massa, M., Nath, C., Noor, M. N. S., and Kassim, A. R. (2006). Diversity in tropical forests. Science, 311(5766):527.
Wilsey, B. J. (2004). Realistically Low Species Evenness Does Not Alter Grassland SpeciesRichness Productivity Relationships. Ecology, 85(10):2693–2700.
Wilsey, B. J. and Polley, H. W. (2002). Reductions in grassland species evenness increase dicot seedling invasion and spittle bug infestation. Ecology Letters, 5(5):676–684.
Wilsey, B. J. and Potvin, C. (2000). Biodiversity and ecosystem functioning: Importance of species evenness in an old field. Ecology, 81(4):887–892.
Wilsey, B. J., Teaschner, T. B., Daneshgar, P. P., Isbell, F. I., and Polley, H. W. (2009). Biodiversity maintenance mechanisms differ between native and novel exotic-dominated communities. Ecology Letters, 12(5):432–442.
Wimpenny, J. W. T. (1992). Microbial Systems, pages 469–522. Springer US, Boston, MA.
Wimpenny, J. W. T. and Colasanti, R. (1997). A unifying hypothesis for the structure of microbial biofilms based on cellular automaton models. FEMS Microbiology Ecology, 22:1–16.
Wittebolle, L., Marzorati, M., Clement, L., Balloi, A., Daffonchio, D., De Vos, P., Heylen, K., Verstraete, W., and Boon, N. (2009). Initial community evenness favours functionality under selective stress. Nature, 458(7238):623–626.










