Artificiële intelligentie doorgrondt ons immuunsysteem
Het immuunsysteem beschermt ons tegen allerhande pathogenen – bacteriën, virussen, etc. Het is echter niet opgewassen tegen elke indringer. Er is bijvoorbeeld nog steeds geen geneesmiddel voor het HIV-virus dat in 2016 nog 1 miljoen levens eiste (WHO, 2017). Blijvend onderzoek naar het immuunsysteem is dus van belang. Een nieuwe techniek die ons gedetailleerde inzichten kan verschaffen in dit systeem is afbeeldingsflowcytometrie. In mijn scriptie werd deze techniek met artificiële intelligentie gecombineerd.
Het immuunsysteem is een erg complex geheel van cellen en mechanismen dat een organisme beschermt tegen allerlei indringers of pathogenen waaronder virussen en bacteriën. Om dit te bereiken beschikt het immuunsysteem over een groot arsenaal van cellen die zich voornamelijk in het bloed bevinden. Elke cel in dit systeem is van een bepaald type, en elk type cel heeft een specifieke functie. Zo zijn er bijvoorbeeld de neutrofielen die snel reageren na een ontsteking, de T-helpercellen die vechten tegen infecties, en nog tientallen andere types.
Er zijn echter nog heel wat pathogenen waar het immuunsysteem niet tegen is opgewassen. Denk bijvoorbeeld aan het eerder vermelde HIV-virus. Dit virus verzwakt het immuunsysteem zodat het ons niet langer kan beschermen tegen een griepje of verkoudheid. We noemen dit immunodeficiëntie. Het immuunsysteem reageert ook zonder infecties niet altijd foutloos. Het kan bijvoorbeeld een allergische reactie teweeg brengen wanneer het te heftig reageert op bepaalde onschadelijke prikkels, zoals een wespensteek of pollen bij iemand met hooikoorts.
Immuuncellen onder de loep
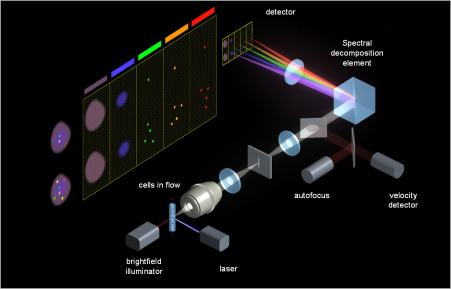 Om deze ziektes en kwalen de wereld uit te helpen, moeten we het immuunsysteem volledig doorgronden en karakteriseren. Dit wordt onderzocht in het domein van de immunologie waarin mijn onderzoek zich ook bevindt. In mijn scriptie onderzocht ik een techniek om met artificiële intelligentie immuuncellen te classificeren op basis van afbeeldingen ervan, gemaakt door een afbeeldingsflowcytometer (Figuur 1). Zo’n instrument fotografeert duizenden cellen per seconde met hoge precisie en verzamelt zo veel informatie over de cellen in een bloedstaal van een patiënt, bijvoorbeeld. Door de afbeeldingen te verwerken met artificiële intelligentie kunnen we uit die hoop informatie ook heel wat afleiden over het immuunsysteem van een patiënt.
Om deze ziektes en kwalen de wereld uit te helpen, moeten we het immuunsysteem volledig doorgronden en karakteriseren. Dit wordt onderzocht in het domein van de immunologie waarin mijn onderzoek zich ook bevindt. In mijn scriptie onderzocht ik een techniek om met artificiële intelligentie immuuncellen te classificeren op basis van afbeeldingen ervan, gemaakt door een afbeeldingsflowcytometer (Figuur 1). Zo’n instrument fotografeert duizenden cellen per seconde met hoge precisie en verzamelt zo veel informatie over de cellen in een bloedstaal van een patiënt, bijvoorbeeld. Door de afbeeldingen te verwerken met artificiële intelligentie kunnen we uit die hoop informatie ook heel wat afleiden over het immuunsysteem van een patiënt.
Een afbeeldingsflowcytometer kan twee types afbeeldingen maken van een cel. Enerzijds zijn er de label-vrije afbeeldingen (eerste tot derde afbeelding in Figuur 2), en anderzijds de fluorescentie afbeeldingen (vierde tot laatste afbeelding in Figuur 2). In het eerste type afbeeldingen wordt de vorm en het uitzicht van de cel vastgelegd met een soort microscoop. Voor het tweede type afbeeldingen moeten er eerst fluorescente labels aangebracht worden op de cellen. Die labels geven fluorescent licht af van een specifieke kleur wanneer de cellen gefotografeerd worden. Aan de hand van de specifieke kleur kunnen we dan vaststellen van welk type de cel is. We geven een cel als het ware een fluorescerend hesje om ze sneller en makkelijker te kunnen herkennen en classificeren. Deze labels zijn echter duur en ze aanbrengen op de cellen is een complex en arbeidsintensief proces.

Efficiënter (ver)werken met artificiële intelligentie
In het beste geval gebruiken we dus enkel de label-vrije afbeeldingen om de cel-classificatie uit te voeren. Wanneer we dit doen, beschikken we echter over heel wat minder informatie om de cellen te herkennen, wat een manuele classificatie onmogelijk maakt. In mijn scriptie heb ik onderzocht of een algoritme er toch nog in kan slagen om de cellen juist te sorteren. Hiervoor werden een aantal technieken uit de artificiële intelligentie toegepast.
De gebruikte technieken vallen onder de noemer van machine learning. Bij dit soort technieken leert een algoritme zelf regels om bijvoorbeeld een onderscheid te kunnen maken tussen cellen op basis van subtiele karakteristieken van deze cellen. Deze karakteristieken kunnen bijvoorbeeld de diameter van een cel of dikte van de celwand zijn. Om de regels te leren moet het algoritme een groot aantal voorbeelden te zien krijgen. Deze voorbeelden moeten ook gelabeld zijn – niet te verwarren met de fluorescente labels van eerder –, wat betekent dat we het algoritme moeten vertellen dat het naar een T-helpercel kijkt bijvoorbeeld.
Genoeg voorbeelden van elke celtype verzamelen bleek in dit onderzoek een grote uitdaging te zijn: de meting van de cellen is duur en tijdrovend, en sommige celtypes zijn simpelweg minder frequent aanwezig in het bloed. Om toch genoeg afbeeldingen te kunnen voorleggen aan het algoritme werd data augmentatie toegepast. Dit houdt in dat originele metingen van cellen willekeurig aangepast worden, bijvoorbeeld door de diameter van de cel iets te verhogen of verlagen. Op die manier worden zogezegd nieuwe cellen gecreëerd die als input aan het algoritme getoond kunnen worden.
Deze resultaten zijn zonder twijfel beter dan wat een wetenschapper manueel zou kunnen behalen
In dit onderzoek werden miljoenen label-vrije afbeeldingen van cellen aan een aantal verschillende machine learning algoritmen getoond. De geteste algoritmen presteren heel goed voor bepaalde celtypes. Wanneer we een algoritme cellen tonen van een nieuw bloedstaal is het in staat om minstens 95% van deze cellen juist te classificeren. Deze resultaten zijn zonder twijfel beter dan wat een wetenschapper manueel zou kunnen behalen, gezien de label-vrije afbeeldingen van verschillende celtypes onmogelijk met het blote oog te onderscheiden zijn.
Samengevat toonde dit onderzoek aan dat door artificiële intelligentie toe te passen in bepaalde immunologie-onderzoeken, het toevoegen van fluorescente labels niet langer nodig is. Dit is een interessante vondst die immunologie toegankelijker en efficiënter maakt. Dit onderzoek wordt ook verder gezet in mijn doctoraat waar ik nog meer technieken uit de artificiële intelligentie zal toepassen om nog meer voordeel te halen uit de veelbelovende afbeeldingsflowcytometrie.
Bibliografie
[1] Acharya, U. Rajendra, Oh, Shu Lih, Hagiwara, Yuki, Tan, Jen Hong, Adam, Muhammad, Gertych, Arkadiusz, and Tan, Ru San, "A Deep Convolutional Neural Network Model to Classify Heartbeats", Computers in Biology and Medicine 89 (2017), pp. 389-396. WOS:000413376600038.
[2] Adan, Aysun, Alizada, Günel, Kiraz, Yağmur, Baran, Yusuf, and Nalbant, Ayten, "Flow Cytometry: Basic Principles and Applications", Critical Reviews in Biotechnology 37, 2 (2017), pp. 163-176.
[3] Alpaydin, Ethem, Introduction to Machine Learning 2nd ed (Cambridge, Mass: MIT Press, 2010). OCLC: ocn317698631.
[4] Amnis, "IDEAS - Image Data Exploration and Analysis Software", Amnis (2015).
[5] Barteneva, Natasha S., Fasler-Kan, Elizaveta, and Vorobjev, Ivan A., "Imaging Flow Cytometry", Journal of Histochemistry and Cytochemistry 60, 10 (2012), pp. 723-733.
[6] Basiji, David A., Ortyn, William E., Liang, Luchuan, Venkatachalam, Vidya, and Morrissey, Philip, "Cellular Image Analysis and Imaging by Flow Cytometry", Clinics in laboratory medicine 27, 3 (2007), pp. 653-viii.
[7] Bengio, Yoshua, Simard, Patrice, and Frasconi, Paolo, "Learning Long-Term Dependencies with Gradient Descent Is Difficult", IEEE transactions on neural networks 5, 2 (1994), pp. 157-166.
[8] Bishop, Christopher M., Pattern Recognition and Machine Learning (New York: Springer, 2006).
[9] Blasi, Thomas, Hennig, Holger, Summers, Huw D., Theis, Fabian J., Cerveira, Joana, Patterson, James O., Davies, Derek, Filby, A…, "Label-Free Cell Cycle Analysis for High-Throughput Imaging Flow Cytometry", Nature Communications 7 (2016), pp. 10256.
[10] Breiman, Leo, "Random Forests" (2001).
[11] Bushnell, Tim, "Spectral Profile And Spectral Viewer | Expert Cytometry | Flow Cytometry Training", Expert Cytometry (2013).
[12] Cauchy, Augustin, "Comptes Rendus Hebdomadaires Des Séances de l'Académie Des Sciences / Publiés... Par MM. Les Secrétaires Perṕ…", Gallica (1847).
[13] Chattopadhyay, Pratip K., Hogerkorp, Carl-Magnus, and Roederer, Mario, "A Chromatic Explosion: The Development and Future of Multiparameter Flow Cytometry", Immunology 125, 4 (2008), pp. 441-449.
[14] Chattopadhyay, Pratip K., Gaylord, Brent, Palmer, Adrian, Jiang, Nan, Raven, Mary A., Lewis, Geoff, Reuter, Morgan A., Rahman, …, "Brilliant Violet Fluorophores: A New Class of Ultrabright Fluorescent Compounds for Immunofluorescence Experiments", Cytometry Part A 81A, 6 (2012), pp. 456-466.
[15] Chawla, N. V., Bowyer, K. W., Hall, L. O., and Kegelmeyer, W. P., "SMOTE: Synthetic Minority Over-Sampling Technique", Journal of Artificial Intelligence Research 16 (2002), pp. 321-357.
[16] Chen, A. Y., Yu, C., Gatto, B., and Liu, L. F., "DNA Minor Groove-Binding Ligands: A Different Class of Mammalian DNA Topoisomerase I Inhibitors", Proceedings of the National Academy of Sciences 90, 17 (1993), pp. 8131-8135.
[17] Chollet, François and others, "Keras", (2015).
[18] Crick, Francis, "Central Dogma Of Molecular Biology", Nature 227 (1970).
[19] Cubuk, Ekin D., Zoph, Barret, Mane, Dandelion, Vasudevan, Vijay, and Le, Quoc V., "AutoAugment: Learning Augmentation Policies from Data", arXiv:1805.09501 [cs, stat] (2018).
[20] Ding, Mei, Kaspersson, Karin, Murray, David, and Bardelle, Catherine, "High-Throughput Flow Cytometry for Drug Discovery: Principles, Applications, and Case Studies", Drug Discovery Today 22, 12 (2017), pp. 1844-1850.
[21] Doan, Minh, Vorobjev, Ivan, Rees, Paul, Filby, Andrew, Wolkenhauer, Olaf, Goldfeld, Anne E., Lieberman, Judy, Barteneva, Natash…, "Diagnostic Potential of Imaging Flow Cytometry", Trends in Biotechnology 0, 0 (2018).
[22] Doan, Minh, Sebastian, Joseph A., Pinto, Ruben N., McQuin, Claire, Goodman, Allen, Wolkenhauer, Olaf, Parsons, Michael J., Acke…, "Label-Free Assessment of Red Blood Cell Storage Lesions by Deep Learning", bioRxiv (2018), pp. 256180.
[23] Eulenberg, Philipp, Köhler, Niklas, Blasi, Thomas, Filby, Andrew, Carpenter, Anne E., Rees, Paul, Theis, Fabian J., and Wolf, F…, "Reconstructing Cell Cycle and Disease Progression Using Deep Learning", Nature Communications 8, 1 (2017).
[24] Fidler, Isaiah J., "Biological Heterogeneity of Cancer", Human Vaccines & Immunotherapeutics 8, 8 (2012), pp. 1141-1142.
[25] Filby, Andrew and Houston, Jessica P., "Imaging Cytometry: Automated Morphology and Feature Extraction", Cytometry Part A 91, 9 (2017), pp. 851-853.
[26] Fisher, R. A., "The Use of Multiple Measurements in Taxonomic Problems", Annals of Eugenics 7, 2 (1936), pp. 179-188.
[27] George, Thaddeus C., Basiji, David A., Hall, Brian E., Lynch, David H., Ortyn, William E., Perry, David J., Seo, Michael J., Zi…, "Distinguishing Modes of Cell Death Using the ImageStream® Multispectral Imaging Flow Cytometer", Cytometry Part A 59A, 2 (2003), pp. 237-245.
[28] Glorot, Xavier and Bengio, Yoshua, "Understanding the Difficulty of Training Deep Feedforward Neural Networks", (2010), pp. 8.
[29] Glorot, Xavier, Bordes, Antoine, and Bengio, Yoshua, "Deep Sparse Rectifier Neural Networks", in Proceedings of the Fourteenth International Conference on Artificial Intelligence and Statistics (, 2011), pp. 315-323.
[30] Goodfellow, Ian, Bengio, Yoshua, and Courville, Aaron, Deep Learning (MIT Press, 2016).
[31] Ha, T. M. and Bunke, H., "Off-Line, Handwritten Numeral Recognition by Perturbation Method", IEEE Transactions on Pattern Analysis and Machine Intelligence 19, 5 (1997), pp. 535-539.
[32] Hanson, Stephen Jose and Pratt, Lorien Y, "Comparing Biases for Minimal Network Construction with Back-Propagation", (1989), pp. 9.
[33] He, Kaiming, "Deep Learning Gets Way Deeper", (2016), pp. 89.
[34] Hennig, Holger, Rees, Paul, Blasi, Thomas, Kamentsky, Lee, Hung, Jane, Dao, David, Carpenter, Anne E., and Filby, Andrew, "An Open-Source Solution for Advanced Imaging Flow Cytometry Data Analysis Using Machine Learning", Methods (San Diego, Calif.) 112 (2017), pp. 201-210.
[35] Heo, Young Jin, Lee, Donghyeon, Kang, Junsu, Lee, Keondo, and Chung, Wan Kyun, "Real-Time Image Processing for Microscopy-Based Label-Free Imaging Flow Cytometry in a Microfluidic Chip", Scientific Reports 7, 1 (2017), pp. 11651.
[36] Ibrahim, Sherrif F. and van den Engh, Ger, "Flow Cytometry and Cell Sorting", in Cell Separation (Springer, Berlin, Heidelberg, 2007), pp. 19-39.
[37] Japkowicz, Nathalie, "The Class Imbalance Problem: Signi Cance and Strategies", (2000), pp. 7.
[38] Jones, Eric, Oliphant, Travis, Peterson, Pearu, and others, "SciPy: Open Source Scientific Tools for Python", (2001).
[39] Karpathy, Andrej, "CS231n Convolutional Neural Networks for Visual Recognition" (2016).
[40] Kiefer, J. and Wolfowitz, J., "Stochastic Estimation of the Maximum of a Regression Function", The Annals of Mathematical Statistics 23, 3 (1952), pp. 462-466.
[41] Kikly, Kristine K., Bochner, Bruce S., Freeman, Sylvie D., Tan, K.B., Gallagher, Kathleen T., D'Alessio, Karla J., Holmes, Step…, "Identification of SAF-2, a Novel Siglec Expressed on Eosinophils, Mast Cells, and Basophils", Journal of Allergy and Clinical Immunology 105, 6 (2000), pp. 1093-1100.
[42] Klambauer, Günter, Unterthiner, Thomas, Mayr, Andreas, and Hochreiter, Sepp, "Self-Normalizing Neural Networks", in Advances in Neural Information Processing Systems 30 (Curran Associates, Inc., 2017), pp. 971--980.
[43] Krizhevsky, Alex, Sutskever, Ilya, and Hinton, Geoffrey E, "ImageNet Classification with Deep Convolutional Neural Networks", in Advances in Neural Information Processing Systems 25 (Curran Associates, Inc., 2012), pp. 1097--1105.
[44] Köhler, Niklas, "Deepflow: The Neural Network Implementation", Theis Lab (2018).
[45] Landgrebe, Thomas C. W. and Duin, Robert P. W., "Approximating the Multiclass ROC by Pairwise Analysis", Pattern Recognition Letters 28, 13 (2007), pp. 1747-1758.
[46] Lemâitre, Guillaume, Nogueira, Fernando, and Aridas, Christos K., "Imbalanced-Learn: A Python Toolbox to Tackle the Curse of Imbalanced Datasets in Machine Learning", Journal of Machine Learning Research 18, 17 (2017), pp. 1-5.
[47] Liu, X. Y., Wu, J., and Zhou, Z. H., "Exploratory Undersampling for Class-Imbalance Learning", IEEE Transactions on Systems, Man, and Cybernetics, Part B (Cybernetics) 39, 2 (2009), pp. 539-550.
[48] Macey, Marion G., ed., Flow Cytometry: Principles and Applications (Humana Press, 2007).
[49] Malecki, A., Garrido, R., Mattson, M. P., Hennig, B., and Toborek, M., "4-Hydroxynonenal Induces Oxidative Stress and Death of Cultured Spinal Cord Neurons", Journal of Neurochemistry 74, 6 (2000), pp. 2278-2287.
[50] Martín Abadi, Ashish Agarwal, Paul Barham, Eugene Brevdo, Zhifeng Chen, Craig Citro, Greg S. Corrado, Andy Davis, Jeffrey Dean…, TensorFlow: Large-Scale Machine Learning on Heterogeneous Systems (, 2015).
[51] McGrath, Kathleen E., Bushnell, Timothy P., and Palis, James, "Multispectral Imaging of Hematopoietic Cells: Where Flow Meets Morphology", Journal of immunological methods 336, 2 (2008), pp. 91-97.
[52] Merck, "Amnis® Imaging Flow Cytometers" (2016).
[53] Merck, "How Amnis® Imaging Flow Cytometry Works" (2018).
[54] Min, Seonwoo, Lee, Byunghan, and Yoon, Sungroh, "Deep Learning in Bioinformatics", Briefings in Bioinformatics 18, 5 (2017), pp. 851-869.
[55] Murphy, Kevin Patrick, Machine Learning: A Probabilistic Perspective (, 2013).
[56] Nair, Vinod and Hinton, Geoffrey E, "Rectified Linear Units Improve Restricted Boltzmann Machines", (2010), pp. 8.
[57] Nguyen, Quang, Valizadegan, Hamed, and Hauskrecht, Milos, "Learning Classification Models with Soft-Label Information", Journal of the American Medical Informatics Association : JAMIA 21, 3 (2014), pp. 501-508.
[58] Ortyn, William E., Hall, Brian E., George, Thaddeus C., Frost, Keith, Basiji, David A., Perry, David J., Zimmerman, Cathleen A.…, "Sensitivity Measurement and Compensation in Spectral Imaging", Cytometry. Part A: The Journal of the International Society for Analytical Cytology 69, 8 (2006), pp. 852-862.
[59] P.Bradley, Andrew, "The Use of the Area under the ROC Curve in the Evaluation of Machine Learning Algorithms - ScienceDirect" (1997).
[60] Pedregosa, Fabian, Varoquaux, Gaël, Gramfort, Alexandre, Michel, Vincent, Thirion, Bertrand, Grisel, Olivier, Blondel, Mathieu,…, "Scikit-Learn: Machine Learning in Python", Journal of Machine Learning Research 12, Oct (2011), pp. 2825-2830.
[61] Pereira, S., Pinto, A., Alves, V., and Silva, C. A., "Brain Tumor Segmentation Using Convolutional Neural Networks in MRI Images", IEEE Transactions on Medical Imaging 35, 5 (2016), pp. 1240-1251.
[62] Radbruch, A., "Immunofluorescence: Basic Considerations", in Flow Cytometry and Cell Sorting (Springer, Berlin, Heidelberg, 1992), pp. 34-46.
[63] Roederer, Mario, "Spectral Compensation for Flow Cytometry: Visualization Artifacts, Limitations, and Caveats", Cytometry 45, 3 (2001), pp. 194-205.
[64] Rumelhart, David E., Hinton, Geoffrey E., and Williams, Ronald J., "Learning Representations by Back-Propagating Errors", Nature 323, 6088 (1986), pp. 533-536.
[65] Sanderson, Michael J., Smith, Ian, Parker, Ian, and Bootman, Martin D., "Fluorescence Microscopy", Cold Spring Harbor protocols 2014, 10 (2014), pp. pdb.top071795.
[66] Schmidhuber, Jürgen, "Deep Learning in Neural Networks: An Overview", Neural Networks 61 (2015), pp. 85-117.
[67] Smith, Paul J, Wiltshire, Marie, Davies, Sharon, Patterson, Laurence H, and Hoy, Terence, "A Novel Cell Permeant and Far Red-Fluorescing DNA Probe, DRAQ5, for Blood Cell Discrimination by Flow Cytometry", Journal of Immunological Methods 229, 1 (1999), pp. 131-139.
[68] Smolewski, P., Bedner, E., Gorczyca, W., and Darzynkiewicz, Z., ""Liquidless" Cell Staining by Dye Diffusion from Gels and Analysis by Laser Scanning Cytometry: Potential Application at Mi…", Cytometry 44, 4 (2001), pp. 355-360.
[69] Sobel, Irwin, "An Isotropic 3x3 Image Gradient Operator", Presentation at Stanford A.I. Project 1968 (2014).
[70] Sugiyama, Masashi, Lawrence, Neil D, Schwaighofer, Anton, and others, Dataset Shift in Machine Learning (The MIT Press, 2017).
[71] Szegedy, Christian, Wei Liu, Yangqing Jia, Sermanet, Pierre, Reed, Scott, Anguelov, Dragomir, Erhan, Dumitru, Vanhoucke, Vincen…, "Going Deeper with Convolutions", in (IEEE, 2015), pp. 1-9.
[72] Taylor, Ian, "Getting Started with FlowJo | FlowJo, LLC" (2016).
[73] Uhm, Tae Gi, Kim, Byung Soo, and Chung, Il Yup, "Eosinophil Development, Regulation of Eosinophil-Specific Genes, and Role of Eosinophils in the Patho…", Allergy, Asthma & Immunology Research 4, 2 (2012), pp. 68-79.
[74] Vranic, Sandra, Boggetto, Nicole, Contremoulins, Vincent, Mornet, Stéphane, Reinhardt, Nora, Marano, Francelyne, Baeza-Squiban,…, "Deciphering the Mechanisms of Cellular Uptake of Engineered Nanoparticles by Accurate Evaluation of Internalization Using Imagi…", Particle and Fibre Toxicology 10, 1 (2013), pp. 2.
[75] Wojcik, Krzysztof and Dobrucki, Jurek W., "Interaction of a DNA Intercalator DRAQ5, and a Minor Groove Binder SYTO17, with Chromatin in Live Cells--Influence …", Cytometry. Part A: The Journal of the International Society for Analytical Cytology 73, 6 (2008), pp. 555-562.
[76] Zeiler, Matthew D. and Fergus, Rob, "Visualizing and Understanding Convolutional Networks", in Computer Vision – ECCV 2014 vol. 8689, (Cham: Springer International Publishing, 2014), pp. 818-833.
[77] Zuba-Surma, Ewa K, Kucia, Magdalena, Abdel-Latif, Ahmed, Jr, James W Lillard, and Ratajczak, Mariusz Z, "The ImageStream System: A Key Step to a New Era in Imaging", (2007), pp. 12.
[78] van der Walt, Stéfan, Schönberger, Johannes L., Nunez-Iglesias, Juan, Boulogne, François, Warner, Joshua D., Yager, Neil, Gouil…, "Scikit-Image: Image Processing in Python", PeerJ 2 (2014), pp. e453.









