Computervisie in precisielandbouw: een combinatie van twee werelden?
“They can have the world. We’ll create our own.”
– The Lion King, 1994

Hier wordt door sommigen een belangrijke rol toebedeeld aan precisielandbouw, een sector die tracht om landbouwsystemen te ontwikkelen en te optimaliseren. Hierbij streeft men naar het toepassen van de juiste teeltmaatregel op de juiste plaats op het juiste moment. In deze sector werd de laatste jaren sterk geïnvesteerd in de verzameling van data om onderzoek te kunnen doen naar systemen die aan dat globale probleem tegemoet kunnen komen.
Kortom, er wacht een enorme hoeveelheid onontgonnen data in de landbouw. Om deze datastromen de baas te kunnen, zal er een versnelling hoger geschakeld moeten worden door het gebruik van innovatieve analysetechnieken zoals artificiële intelligentie. Er is nood aan een combinatie van twee werelden: die van de computerwetenschappen en die van de landbouw.
Artificiële intelligentie: kan het de wereld helpen?

Artificiële intelligentie is niet meer weg te denken uit onze leefwereld (spraakassistentie, zoekmachines…). In vele gevallen gaat het echter nog om relatief eenvoudige applicaties die simpele taken uitvoeren, waarbij veel en duidelijke data wereldwijd beschikbaar is. In de landbouwsector moet de data voornamelijk zelf verzameld worden en kent deze bovendien een sterke natuurlijke variabiliteit, eigen aan de biologische aard ervan. De enorme variatie in onder meer gewassen en omgevingsfactoren is bijgevolg een belangrijk obstakel in het ontwerp van robuuste systemen.
In deep learning, een recente tak van artificiële intelligentie, wordt er automatisch gezocht naar patronen op basis van een ‘bijleer’ mechanisme. De meest voorkomende vorm hierbij is het gesuperviseerd leren, waarbij tijdens het trainingsproces voorbeelden aan de machine getoond worden om de computer correcte voorspellingen te leren maken. Hierbij is het weliswaar vereist om grote hoeveelheden data te labelen om als voorbeeld te kunnen dienen, een proces dat dan wel weer een knelpunt creëert op vlak van specifieke domeinkennis: een kost- en tijdrovende taak.
Veel taken in de precisielandbouw maken gebruik van deze gesuperviseerde methodes, zo kan bijvoorbeeld bepaald worden of de bloemkool juist of omgekeerd ligt op de transportband of kunnen kevers in planten gedetecteerd worden op basis van fotomateriaal en de intelligentie van de computer.
Semi-gesuperviseerd werken: tijd- en kostenbesparend?
Het grote nadeel is uiteraard dat het tijd kost om telkens alles een label toe te kennen. Onderzoek naar methoden die dit kan reduceren staat in de precisielandbouwsector nog in de kinderschoenen. De methode die hier wordt voorgesteld tracht planten te detecteren in grote overzichtsfoto’s van landbouwvelden, gemaakt door drones, op een semi-gesuperviseerde manier: enkel en alleen de noodzakelijk stukjes afbeelding worden gelabeld. Op die manier werd getracht een zo algemeen bruikbaar en overdraagbaar mogelijk basissysteem te ontwikkelen.
Het vooropgestelde probleem werd bekeken vanuit de hoek van anomaliedetectie. Bij anomaliedetectie beoogt men de afwijkende elementen op te sporen en is er sprake van een sterk ongebalanceerde dataset. In dit opzicht worden de planten gezien als afwijkingen ten opzichte van de gevarieerde achtergrond. Door de detectie van planten te bekijken vanuit deze invalshoek, wordt in de methode gebruik gemaakt van een autoencoder als netwerk, een techniek uit de deep learning.
In een eerste stap, de training van de autoencoder, wordt de overzichtsfoto in kleine deeltjes (monsters) als input gegeven aan het netwerk. Deze worden vervolgens gereduceerd tot een eigenschappen-vector door het netwerk om daarna op basis hiervan de afbeelding terug te reconstrueren. Het voordeel hierbij is dat het netwerk dus autonoom de interessante kenmerken selecteert (niet-gesuperviseerd).
In een tweede stap worden enkele monsters uit de afbeelding geselecteerd voor labeling, dit in tegenstelling tot een complete labeling zoals bij het gesuperviseerd werken. De geselecteerde monsters bevatten zowel planten als achtergrond, met als bedoeling de variatie in de afbeelding zoveel mogelijk te dekken. Deze gelabelde monsters worden door de autoencoder gestuurd, waaruit hun bijhorende eigenschappen-vector gehaald wordt. Op die manier wordt een representatieve set van positieve en negatieve eigenschappen-vectoren opgebouwd. Deze eigenschappen-vectoren worden gebruikt om correlaties te ontdekken.
Om vervolgens te bepalen welke monsters al dan niet planten bevatten, wordt tenslotte een eigenschappen-vector bepaald door de volledige afbeelding (per monster) opnieuw door de autoencoder gestuurd. Met behulp van de Canberra afstandsformule wordt de mate van (on)gelijkheid tussen de eigenschappen-vector en alle eigenschappen-vectoren uit de representatieve set berekend. De minimale afstand en dus grootste gelijkheid tot één van deze representatieve samples bepaalt tot welke categorie dat monster gerekend wordt. Op basis hiervan wordt elk monster dus al dan niet geclassificeerd als plant.
Het idee van deze stap is om een applicatie te bouwen waarbij de landbouwer zijn expertise kan gebruiken om kleine stukjes van het veld te labelen. Vervolgens zal het systeem een volledige classificatie suggereren, waarbij de landbouwer indien gewenst kan bijsturen op eenvoudige wijze.
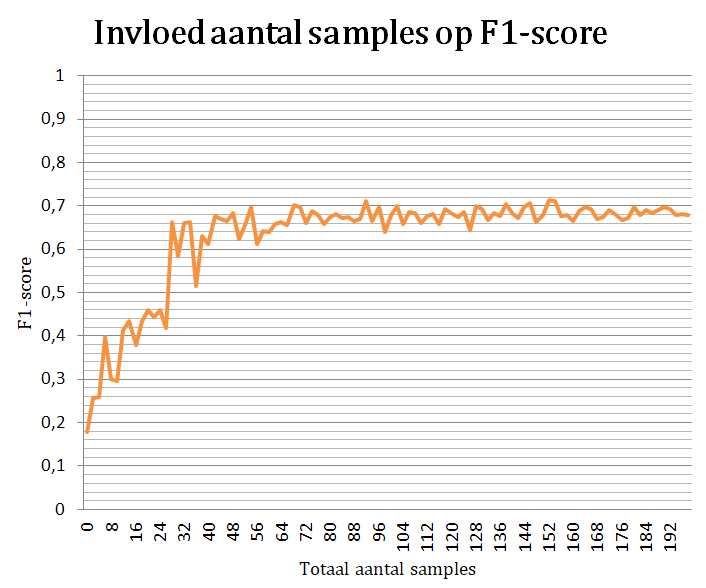
Aangezien deze methode steunt op het idee van minimalisatie van de menselijke interventie, werd ook onderzocht wat het effect is van het aantal gelabelde monsters op de accuraatheid. Hieruit bleek dat de invloed van het aantal stagneert bij zo’n 35 monsters per categorie. Deze grens suggereert dat een extra labeling in deze methode niet resulteert in betere resultaten.
De toekomst?
De procedure zoals voorgesteld in dit werk tracht dus planten te detecteren met een beperkte menselijke interventie. Dit kan in een volgende fase gebruikt worden om onkruiden of zieke planten te detecteren. In de toekomst kunnen intelligente landbouwsystemen ontwikkeld worden die dit automatiseren tot bijvoorbeeld mechanisch wieden in plaats van milieubelastende gewasbeschermingsmiddelen te gebruiken. Want, wees nu eerlijk, een nieuwe fysieke wereld maken is onmogelijk. Wordt het niet tijd om in deze wereld te investeren?
Bibliografie
[1] S. University, “Cs231n: Convolutional neural networks for visual recognition,”
http://cs231n.stanford.edu/.
[2] E. Pekel. (2017) Feed forward and recurrent ann architecture. [Online].
Available: https://www.researchgate.net/figure/Feed-forward-and-recurrent-ANNarchi…
fig1_315111480
[3] U. Udofia. (2018) Basic overview of convolutional neural network (cnn). [Online].
Available: https://medium.com/@udemeudofia01/basic-overview-of-convolutional-neura…-
cnn-4fcc7dbb4f17
[4] A. Deshpande. (2016) A beginner’s guide to understanding convolutional neural networks
- part 2. [Online]. Available: https://adeshpande3.github.io/adeshpande3.github.io/ABeginner’s-
Guide-To-Understanding-Convolutional-Neural-Networks-Part-2/
[5] A. Deshpande. (2016) A beginner’s guide to understanding convolutional neural networks
- part 1. [Online]. Available: https://adeshpande3.github.io/adeshpande3.github.io/ABeginner’s-
Guide-To-Understanding-Convolutional-Neural-Networks/
[6] Y. Saeys, “Machine learning introductie,” 2018, slides available for students at University
Ghent concerning the course ’Artificiële intelligentie’.
[7] S. Cool, “Precisielandbouw: een samenvatting,” Presentatie, 2019.
[8] H. Yalcin, “Plant phenology recognition using deep learning: Deep-pheno,” pp. 1–5, Aug
2017.
[9] A. Kamilaris and F. X. Prenafeta-Boldú, “Deep learning in agriculture: A survey,”
Computers and Electronics in Agriculture, vol. 147, pp. 70 – 90, 2018. [Online]. Available:
http://www.sciencedirect.com/science/article/pii/S0168169917308803
[10] S. Banu, “Precision agriculture: Tomorrow’s technology for today’s farmer,” Journal of Food
Processing & Technology, vol. 6: 468, 2015.
[11] J. McCarthy, “What is artificial intelligence,” 2007. [Online]. Available: http:
//jmc.stanford.edu/articles/whatisai/whatisai.pdf
[12] Y. Saeys, “Artificiële intelligentie,” 2018, slides available for students at University Ghent
concerning the course ’Artificiële intelligentie’.
[13] M. Molina, “Introduction to deep learning,” 2018, slides available for students EIT master
in DataScience at UPM concerning the course ’Deep Learning’.
[14] S. Shalev-Shwartz and S. Ben-David, Understanding Machine Learning: From Theory to
Algorithms. New York, NY, USA: Cambridge University Press, 2014.
[15] D. I. Patrício and R. Rieder, “Computer vision and artificial intelligence in precision
agriculture for grain crops: A systematic review,” Computers and Electronics in Agriculture,
vol. 153, pp. 69 – 81, 2018. [Online]. Available: http://www.sciencedirect.com/science/
article/pii/S0168169918305829
[16] D. Manrique Gamo, “Neural networks and deep learning: Foundations,” 2018, slides available
for students EIT master in DataScience at UPM concerning the course ’Deep Learning’.
[17] V. Vanhoucke, A. Senior, and M. Z. Mao, “Improving the speed of neural networks on
cpus,” 2011.
[18] M. Ponti, L. Ribeiro, T. Santana Nazare, T. Bui, and J. Collomosse, “Everything you wanted
to know about deep learning for computer vision but were afraid to ask,” pp. 17–41, 10
2017.
[19] L. Baumela, “Introduction to computer vision,” 2018, slides available for students EIT
master in DataScience at UPM concerning the course ’Deep Learning’.
[20] P. Veelaert, “Basic problems computer vision,” 2019, slides available for students at University
Ghent concerning the course ’Computervisie’.
[21] A. Golda. (2005) Introduction to neural networks. [Online]. Available: http://home.agh.
edu.pl/~vlsi/AI/intro/
[22] M. A. Nielsen, Neural networks and deep learning. Determination Press, 2015.
[23] A. M. Tekalp and O. Kirmemis. (2018) Deep learning for image and video processing.
University Lecture.
[24] D. Manrique Gamo, “Neural networks and deep learning: Learning. logistic regression,”
2018, slides available for students EIT master in DataScience at UPM concerning the course
[25] D. Manrique Gamo., “Neural networks and deep learning: Scaling up. deep learning,” 2018,
slides available for students EIT master in DataScience at UPM concerning the course ’Deep
Learning’.
[26] L. Baumela, “Convolutional neural networks (i): Fundamentals,” 2018, slides available for
students EIT master in DataScience at UPM concerning the course ’Deep Learning’.
[27] Y. LeCun, L. Bottou, G. B. Orr, and K.-R. Müller, “Efficient backprop,” pp. 9–50, 1998.
[Online]. Available: http://dl.acm.org/citation.cfm?id=645754.668382
[28] S. Russell and P. Norvig, Artificial Intelligence: A Modern Approach, 3rd ed. Upper Saddle
River, NJ, USA: Prentice Hall Press, 2009.
[29] A. Pizurica, “Neural networks,” 2018, slides available for students at University Ghent
concerning the course ’Artificial intelligence’.
[30] M. Molina, “Performance evaluation,” 2018, slides available for students EIT master in
DataScience at UPM concerning the course ’Deep Learning’.
[31] S. university. Autoencoders. [Online]. Available: http://ufldl.stanford.edu/tutorial/
unsupervised/Autoencoders/
[32] D. Erhan, P.-A. Manzagol, Y. Bengio, S. Bengio, and P. Vincent, “The difficulty of training
deep architectures and the effect of unsupervised pre-training,” vol. 5, pp. 153–160, 16–18
Apr 2009. [Online]. Available: http://proceedings.mlr.press/v5/erhan09a.html
[33] A. Pizurica, “Supervised learning,” 2018, slides available for students at University Ghent
concerning the course ’Artificial intelligence’.
[34] D. Soni. (2018) Supervised vs. unsupervised learning. [Online]. Available: https:
//towardsdatascience.com/supervised-vs-unsupervised-learning-14f68e32ea8d
[35] R. Shwartz-Ziv and N. Tishby, “Opening the black box of deep neural networks via
information,” CoRR, vol. abs/1703.00810, 2017. [Online]. Available: http://arxiv.org/abs/
1703.00810
[36] W. H. Maes and K. Steppe, “Perspectives for remote sensing with unmanned aerial vehicles
in precision agriculture,” Trends in Plant Science, vol. 24, no. 2, pp. 152 – 164, 2019.
[Online]. Available: http://www.sciencedirect.com/science/article/pii/S1360138518302693
[37] W. Nugteren, “Precisielandbouw en gewasbescherming: hoe precies?” 2009. [Online].
Available: https://www.knpv.org/db/upload/documents/Precisielandbouw/Nugteren.pdf
[38] A. Chlingaryan, S. Sukkarieh, and B. Whelan, “Machine learning approaches for crop yield
prediction and nitrogen status estimation in precision agriculture: A review,” Computers
and Electronics in Agriculture, vol. 151, pp. 61–69, 08 2018.
[39] J. V. Stafford, “Implementing precision agriculture in the 21st century,” Journal of
Agricultural Engineering Research, vol. 76, no. 3, pp. 267 – 275, 2000. [Online]. Available:
http://www.sciencedirect.com/science/article/pii/S0021863400905778
[40] N. Zhang, M. Wang, and N. Wang, “Precision agriculture-a worldwide overview,” Computers
and Electronics in Agriculture, vol. 36, no. 2-3, pp. 113 – 132, 2002.
[41] D. Slaughter, D. Giles, and D. Downey, “Autonomous robotic weed control systems: A
review,” Computers and Electronics in Agriculture, vol. 61, no. 1, pp. 63 – 78, 2008,
emerging Technologies For Real-time and Integrated Agriculture Decisions. [Online].
Available: http://www.sciencedirect.com/science/article/pii/S0168169907001688
[42] P. Lottes, M. Hoeferlin, S. Sander, M. Müter, P. Schulze, and L. C. Stachniss, “An effective
classification system for separating sugar beets and weeds for precision farming applications,”
pp. 5157–5163, May 2016.
[43] P. Lottes, J. Behley, A. Milioto, and C. Stachniss, “Fully convolutional networks with
sequential information for robust crop and weed detection in precision farming,” CoRR,
vol. abs/1806.03412, 2018. [Online]. Available: http://arxiv.org/abs/1806.03412
[44] C. Potena, D. Nardi, and A. Pretto, “Fast and accurate crop and weed identification with
summarized train sets for precision agriculture,” Advances in Intelligent Systems and Computing,
vol. 531, pp. 105–121, 02 2017.
[45] A. Milioto, P. Lottes, and C. Stachniss, “Real-time semantic segmentation of crop and
weed for precision agriculture robots leveraging background knowledge in cnns,” CoRR,
vol. abs/1709.06764, 2017. [Online]. Available: http://arxiv.org/abs/1709.06764
[46] M. Fawakherji, A. Youssef, D. Bloisi, A. Pretto, and D. Nardi, “Crop and weeds classification
for precision agriculture using context-independent pixel-wise segmentation,” pp. 146–152,
Feb 2019.
[47] C. Hung, Z. Xu, and S. Sukkarieh, “Feature learning based approach for weed classification
using high resolution aerial images from a digital camera mounted on a uav,” Remote
Sensing, vol. 6, pp. 12 037–12 054, 12 2014.
[48] P. Lottes, R. Khanna, J. Pfeifer, R. Siegwart, and C. Stachniss, “Uav-based crop and weed
classification for smart farming,” pp. 3024–3031, May 2017.
[49] A. Milioto, P. Lottes, and C. Stachniss, “Real-time blob-wise sugar beets vs weeds
classification for monitoring fields using convolutional neural networks,” ISPRS Annals
of Photogrammetry, Remote Sensing and Spatial Information Sciences, vol. IV-2/W3, pp.
41–48, 2017. [Online]. Available: https://www.isprs-ann-photogramm-remote-sens-spatialinf-
sci.net/IV-2-W3/41/2017/
[50] M. D. Bah, A. Hafiane, and R. Canals, “Deep learning with unsupervised data labeling for
weeds detection on UAV images,” CoRR, vol. abs/1805.12395, 2018. [Online]. Available:
http://arxiv.org/abs/1805.12395
[51] M. Pérez-Ortiz, P. A. Gutiérrez, J. M. Peña-Barragán, J. Torres-Sánchez, C. Martínez,
and F. López-Granados, “An experimental comparison for the identification of weeds in
sunflower crops via unmanned aerial vehicles and object-based analysis,” vol. 9094, 06 2015.
[52] M. Pérez-Ortiz, J. M. Peña, P. A. Gutiérrez, J. Torres-Sánchez, C. Hervás-Martínez, and
F. López-Granados, “Selecting patterns and features for between- and within- crop-row weed
mapping using uav-imagery,” Expert Systems with Applications, vol. 47, pp. 85 – 94, 2016.
[Online]. Available: http://www.sciencedirect.com/science/article/pii/S0957417415007472
[53] E. Hamuda, M. Glavin, and E. Jones, “A survey of image processing techniques for
plant extraction and segmentation in the field,” Computers and Electronics in Agriculture,
vol. 125, pp. 184 – 199, 2016. [Online]. Available: http://www.sciencedirect.com/science/
article/pii/S0168169916301557
[54] J. Gao, W. Liao, D. Nuyttens, P. Lootens, J. Vangeyte, A. Pižurica, Y. He, and
J. G. Pieters, “Fusion of pixel and object-based features for weed mapping using
unmanned aerial vehicle imagery,” International Journal of Applied Earth Observation and
Geoinformation, vol. 67, pp. 43 – 53, 2018. [Online]. Available: http://www.sciencedirect.
com/science/article/pii/S0303243417303252
[55] “Color indices for weed identification under various soil, residue, and lighting conditions,”
Transactions of the ASABE, vol. 38, no. 1, pp. 259–269, 1 1995.
[56] Y. Kyosuke, W. Guo, Y. Yoshioka, and S. Ninomiya, “On plant detection of intact tomato
fruits using image analysis and machine learning methods,” Sensors (Basel, Switzerland),
vol. 14, pp. 12 191–12 206, 07 2014.
[57] S. W. Chen, S. S. Shivakumar, S. Dcunha, J. Das, E. Okon, C. Qu, C. J. Taylor, and
V. Kumar, “Counting apples and oranges with deep learning: A data-driven approach,”
IEEE Robotics and Automation Letters, vol. 2, no. 2, pp. 781–788, April 2017.
[58] O. Csillik, “Superpixels: the end of pixels in obia. a comparison of state-of-the-art superpixel
methods for remote sensing data.”
[59] K. P. Ferentinos, “Deep learning models for plant disease detection and diagnosis,”
Computers and Electronics in Agriculture, vol. 145, pp. 311 – 318, 2018. [Online]. Available:
http://www.sciencedirect.com/science/article/pii/S0168169917311742
[60] X. Pantazi, D. Moshou, T. Alexandridis, R. Whetton, and A. Mouazen, “Wheat
yield prediction using machine learning and advanced sensing techniques,” Computers
and Electronics in Agriculture, vol. 121, pp. 57 – 65, 2016. [Online]. Available:
http://www.sciencedirect.com/science/article/pii/S0168169915003671
[61] J. Cnops and R. Stoop, “Algoritmen ii,” 2018, available for students at University Ghentconcerning
the course ’Gevorderde algoritmen’.
[62] M. D. Cicco, C. Potena, G. Grisetti, and A. Pretto, “Automatic model based dataset
generation for fast and accurate crop and weeds detection,” CoRR, vol. abs/1612.03019,
2016. [Online]. Available: http://arxiv.org/abs/1612.03019
[63] I. Goodfellow, Y. Bengio, and A. Courville, Deep Learning. MIT Press, 2016, http://www.
deeplearningbook.org.
[64] TensorFlow, “Tensorflow.” [Online]. Available: https://www.tensorflow.org/
[65] Keras, “Keras.” [Online]. Available: https://keras.io/
[66] C. Van Ceulebroeck, L. Vanhaeren, F. Tememrman, V. De Blauwer, N. Rutten, J. Witters,
L. Taning, N. Berckvens, and H. Casteels, “Onderzoek naar ritnaalden uit de startblokken,”
pp. 10–13, 2 2016.
[67] M. P. Paing and S. Choomchuay, “Improved random forest (rf) classifier for imbalanced
classification of lung nodules,” pp. 1–4, July 2018.
[68] R. G. Devi and P. Sumanjani, “Improved classification techniques by combining knn and
random forest with naive bayesian classifier,” pp. 1–4, March 2015.
[69] D. Manrique Gamo, “Neural networks and deep learning: Learning. linear regression,” 2018,
slides available for students EIT master in DataScience at UPM concerning the course ’Deep
Learning’.
[70] L. Baumela, “Convolutional neural networks (ii): Regularization,” 2018, slides avaible for
students EIT master in DataScience at UPM concerning the course ’Deep Learning’.
[71] M. D. Zeiler and R. Fergus, “Visualizing and understanding convolutional networks,”
CoRR, vol. abs/1311.2901, 2013. [Online]. Available: http://arxiv.org/abs/1311.2901
[72] K. Sabanci, A. Kayabasi, and A. Toktas, “Computer vision-based method for classification
of the wheat grains using artificial neural network,” Journal of the Science of Food and
Agriculture, vol. 97, p. 2588–2593, 06 2017.
[73] Agisoft LLC, “Agisoft metashape.” [Online]. Available: https://www.agisoft.com/about/
[74] PyTorch, “Torchvision.transforms package.” [Online]. Available: https://pytorch.org/docs/
stable/torchvision/transforms.html
[75] R. Chalapathy and S. Chawla, “Deep learning for anomaly detection: A survey,” CoRR,
vol. abs/1901.03407, 2019. [Online]. Available: http://arxiv.org/abs/1901.03407
[76] A. Borghesi, A. Bartolini, M. Lombardi, M. Milano, and L. Benini, “Anomaly detection
using autoencoders in high performance computing systems,” CoRR, vol. abs/1811.05269,
2018. [Online]. Available: http://arxiv.org/abs/1811.05269
[77] A. Bhattad, J. Rock, and D. A. Forsyth, “Detecting anomalous faces with
’no peeking’ autoencoders,” CoRR, vol. abs/1802.05798, 2018. [Online]. Available:
http://arxiv.org/abs/1802.05798
[78] C. Baur, B. Wiestler, S. Albarqouni, and N. Navab, “Deep autoencoding models for
unsupervised anomaly segmentation in brain MR images,” CoRR, vol. abs/1804.04488,
2018. [Online]. Available: http://arxiv.org/abs/1804.04488
[79] J. Jelínek. (2007) Metrics in similarity search. [Online]. Available: https://is.muni.cz/th/
cxyzo/SimSearch_Metrics.pdf
[80] E. S. M. and Y. N., “Robustness of canberra metric in computer intrusion detection,”
Proceedings of the IEEE Workshop on Information Assurance and Security, 01 2001.
[81] A. Kumar, A. Abdelhadi, and C. Clancy, “Novel anomaly detection and classification schemes
for machine-to-machine uplink,” pp. 1284–1289, Dec 2018.
[82] L. Derksen. (2016) Visualising high-dimensional datasets using pca and t-sne in
python. [Online]. Available: https://towardsdatascience.com/visualising-high-dimensionaldatasets-
using-pca-and-t-sne-in-python-8ef87e7915b
[83] PyTorch, “Pytorch.” [Online]. Available: https://pytorch.org/
[84] B. Zhang, “Computer vision vs. human vision,” pp. 3–3, July 2010.
[85] S. Hosein. (2018) Active learning: Curious ai algorithms. [Online]. Available: https:
//www.datacamp.com/community/tutorials/active-learning
[86] M. J. Garbade. (2018) Understanding k-means clustering in machine learning. [Online].
Available: https://towardsdatascience.com/understanding-k-means-clustering-in-mach…-
6a6e67336aa1









