Blindheid genezen met kanker
Kankercellen vermenigvuldigen zich snel en zijn soms onsterfelijk. Daardoor kunnen ze gebruikt worden om een ongelimiteerde hoeveelheid cellen te produceren voor de ontwikkeling van transplanteerbare weefsels.
Dat de beperkte hoeveelheid orgaandonoren voor lange wachtlijsten zorgt is algemeen gekend, maar ook wetenschappelijk onderzoek is sterk afhankelijk van humane cellen afkomstig van deze donoren. Decennia geleden heeft de wetenschap een oplossing bedacht voor dit tekort door “normale” cellen om te zetten in kankercellen. Kanker ontstaat door een opstapeling van mutaties in het DNA van een cel. Het is moeilijk om deze natuurlijke strategie op een gecontroleerde manier na te bootsen in het laboratorium. Daarom worden de cellen genetisch gemanipuleerd door speciale kankergenen in het DNA van de cellen te brengen. Dankzij deze genen krijgen de cellen eigenschappen die met kanker geassocieerd worden, zoals een snelle en onbeperkte vermenigvuldiging.
Het omzetten van “normale” cellen naar kankercellen in het laboratorium wordt ook wel immortalisatie genoemd. Op deze manier kan men van een kleine hoeveelheid cellen van een orgaandonor, oneindig veel cellen produceren en vervolgens gebruiken voor experimenten. Nagenoeg elk celtype in het menselijk lichaam (levercel, spiercel, witte bloedcel, …) kan geïmmortaliseerd worden. Maar deze geïmmortaliseerde cellen gebruiken om transplanteerbare weefsels te maken is gevaarlijk voor de patiënt… toch?
Knutselen met DNA
Geïmmortaliseerde cellen kunnen zich snel en ongelimiteerd vermenigvuldigen in het laboratorium. Maar doordat de omstandigheden in het lichaam minder optimaal zijn, leiden ze niet altijd tot de vorming van een tumor na transplantatie. Toch is de aanwezigheid van kankergenen een veiligheidsrisico, waardoor deze cellen niet geschikt zijn voor het ontwikkelen van transplanteerbare weefsels.
Als oplossing voor dit probleem kunnen de geïmmortaliseerde cellen opnieuw omgezet worden in “normale” cellen voordat ze in het lichaam terechtkomen. Dit gebeurt door de kankergenen, die eerder in het DNA van de cellen geplakt werden, weg te knippen. Hierdoor verliezen de geïmmortaliseerde cellen de eigenschap om zich snel en onbeperkt te vermenigvuldigen. Extra veiligheidsmechanismen verzekeren dat resterende cellen, waarbij het kankergen nog aanwezig is, verwijderd worden voordat ze in de patiënt terechtkomen.
Wat overblijft is een ongelimiteerde voorraad aan “normale” cellen. Doordat organen vaak uit verschillende celtypes bestaan, maar ook bloedvaten en zenuwen bevatten, zijn deze cellen op zichzelf onvoldoende om volledige organen na te maken zoals de lever of de longen. Wel kunnen de verkregen cellen gebruikt worden om eenvoudige weefsels te ontwikkelen die bestaan uit één celtype en waarin bloedvaten en zenuwen overbodig zijn. Een voorbeeld van zo’n weefsel is het endotheel. Het bevindt zich aan de achterzijde van het hoornvlies, de doorzichtige laag aan de voorzijde van het oog (Figuur 1).
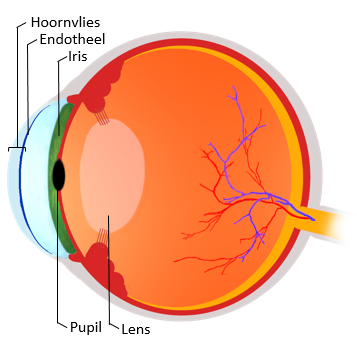
Figuur 1: Schematische voorstelling van het oog.
Ik zie, ik zie wat jij niet ziet
Vertroebelingen van het hoornvlies hebben een enorme invloed op onze samenleving. De visuele beperking die hiermee gepaard gaat, tast de zelfstandigheid van de patiënten aan. Ook de kosten voor de maatschappij kunnen stevig oplopen. Wereldwijd wachten 13 miljoen mensen op een hoornvliestransplantatie om van hun visuele beperking af te raken. Meer dan de helft van deze patiënten kan geholpen worden door enkel het hoornvlies endotheel te vervangen.
Ook nu al transplanteert men alleen het endotheel in plaats van het volledige hoornvlies indien dit mogelijk is. Dit heeft als voordeel dat de patiënt sneller herstelt na de operatie. Voor een transplantatie van het endotheel is de patiënt op dit moment nog afhankelijk van een orgaandonor, maar de geïmmortaliseerde cellen kunnen hier verandering in brengen.
Door de endotheelcellen van het hoornvlies van één orgaandonor te immortaliseren, kunnen grote hoeveelheden van dit celtype aangemaakt worden. Eenmaal genoeg cellen zijn verkregen, worden de kankergenen weggeknipt. Wat overblijft zijn “normale” endotheelcellen zonder de eigenschappen van een kankercel. Deze kunnen dan gebruikt worden om patiënten te behandelen die op de wachtlijst staan voor een hoornvliestransplantatie. Normaal is het hoornvlies van één donor geschikt voor de behandeling van één patiënt. Maar met behulp van bovenstaande techniek kan men met één donor, duizenden patiënten van een transplantaat voorzien.
Rome is niet op een dag gebouwd
Ondertussen zijn de experimenten om de kankergenen in het DNA te plakken bijna beëindigd. Hiervoor werd gebruik gemaakt van virussen die van nature expert zijn in het knippen en plakken van DNA. Om na te gaan hoe effectief virussen hierin zijn, hebben we het GFP (groen fluorescerend proteïne) gen in het DNA van de cellen geplakt in plaats van een kankergen. Zoals de naam het al zegt, krijgen de cellen die het GFP gen bevatten een groen fluorescerende kleur. Op deze manier kan eenvoudig worden nagegaan hoeveel cellen het GFP gen bevatten. Hoe meer groen fluorescerende cellen, hoe effectiever het virus. Daarnaast zijn er ook nog andere factoren die een belangrijke rol spelen, zoals de hoeveelheid van en de blootstellingstijd aan deze virussen. Bij het gebruik van grote hoeveelheden virussen bevatten tot 100% van de cellen het GFP gen. In Figuur 2 is een foto te zien waarbij een lagere hoeveelheid virussen gebruikt werd en daardoor maar 60-70% van de cellen het GFP gen in hun DNA hebben opgenomen. Als we het experiment herhalen onder dezelfde omstandigheden, maar met een kankergen in plaats van het GFP gen, dan weten we dat ook hier 60-70% van de cellen het gen zal bevatten.
Het volledige project vraagt natuurlijk jaren van onderzoek en optimalisatie. Voorlopig ziet het er dus naar uit dat we afhankelijk zullen blijven van orgaandonoren om patiënten met een vertroebeling van het hoornvlies vooruit te helpen. Wel zijn we ondertussen een stap dichter gekomen om met kanker blindheid te genezen.
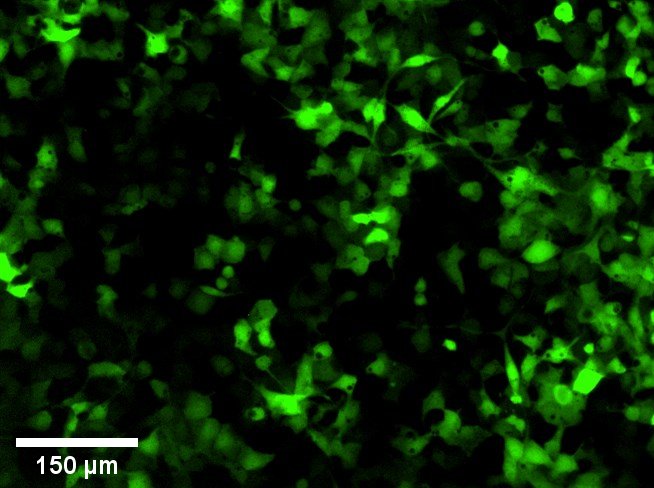
Figuur 2: Foto van genetisch gemanipuleerde hoornvlies endotheelcellen gemaakt met behulp van een microscoop. De cellen hebben een groen fluorescerende kleur dankzij het GFP gen dat met behulp van virussen in hun DNA werd gebracht.
Bibliografie
1. Meek KM, Knupp C. Corneal structure and transparency. Prog Retin Eye Res. 2015;49:1–16.
2. Eghrari AO, Riazuddin SA, Gottsch JD. Overview of the Cornea: Structure, Function, and Development. 1st ed. Vol. 134, Progress in Molecular Biology and Translational Science. Elsevier Inc.; 2015. 7–23 p.
3. Feizi S, Jafarinasab MR, Karimian F, Hasanpour H, Masudi A. Central and peripheral corneal thickness measurement in normal and keratoconic eyes using three corneal pachymeters. J Ophthalmic Vis Res. 2014;9(3):296.
4. Mochizuki H, Fukui M, Hatou S, Yamada M, Tsubota K. Evaluation of ocular surface glycocalyx using lectin-conjugated fluorescein. Clin Ophthalmol. 2010;4(1):925–30.
5. Schermer A, Galvin S, Sun TT. Differentiation-related expression of a major 64K corneal keratin in vivo and in culture suggests limbal location of corneal epithelial stem cells. J Cell Biol. 1986;103(1):49–62.
6. Thoft RA, Friend J. The X, Y, Z hypothesis of corneal epithelial maintenance. Invest Ophthalmol Vis Sci. 1983;24:1442–3.
7. Hayashi S, Osawa T, Tohyama K. Comparative observations on corneas, with special reference to Bowman’s layer and Descemet’s membrane in mammals and amphibians. J Morphol. 2002;254(3):247–58.
8. Peh GSL, Beuerman RW, Colman A, Tan DT, Mehta JS. Human corneal endothelial cell expansion for corneal endothelium transplantation: An overview. Transplantation. 2011;91(8):811–9.
9. Torricelli AAM, Wilson SE. Cellular and extracellular matrix modulation of corneal stromal opacity. Exp Eye Res. 2014;129(October):151–60.
10. DelMonte DW, Kim T. Anatomy and physiology of the cornea. J Cataract Refract Surg. 2011;37(3):588–98.
11. Matthyssen S, Van den Bogerd B, Dhubhghaill SN, Koppen C, Zakaria N. Corneal regeneration: A review of stromal replacements. Acta Biomater. 2018;
12. Bonanno JA. Molecular mechanisms underlying the corneal endothelial pump. Exp Eye Res. 2012;95(1):2–7.
13. Ali M, Raghunathan VK, Li JY, Murphy CJ, Thomasy SM. Biomechanical relationships between the corneal endothelium and Descemet’s membrane. Exp Eye Res. 2016;152:57–70.
14. Joyce NC. Proliferative capacity of the corneal endothelium. Prog Retin Eye Res. 2003;22(3):359–89.
15. Waring GO, Bourne WM, Edelhauser HF, Kenyon KR. The Corneal Endothelium: Normal and Pathologic Structure and Function. Ophthalmology. 1982;89(6):531–90.
16. Bonanno JA. Identity and regulation of ion transport mechanisms in the corneal endothelium. Prog Retin Eye Res. 2003;22(1):69–94.
17. Elbaz U, Mireskandari K, Tehrani N, Shen C, Khan MS, Williams S, et al. Corneal endothelial cell density in children: normative data from birth to 5 years old. Am J Ophthalmol. 2017;173:134–8.
18. Abdellah MM, Ammar HG, Anbar M, Mostafa EM, Farouk MM, Sayed K, et al. Corneal Endothelial Cell Density and Morphology in Healthy Egyptian Eyes. J Ophthalmol. 2019;2019(Cd).
19. Bourne WM, Nelson LR, Hodge DO. Central corneal endothelial cell changes over a ten-year period. Invest Ophthalmol Vis Sci. 1997;38(3):779–82.
20. Van den Bogerd B, Dhubhghaill SN, Koppen C, Tassignon MJ, Zakaria N. A review of the evidence for in vivo corneal endothelial regeneration. Surv Ophthalmol. 2018;63(2):149–65.
21. Joyce NC, Meklir B, Joyce SJ, Zieske JD. Cell cycle protein expression and proliferative status in human corneal cells. Investig Ophthalmol Vis Sci. 1996;37(4):645–55.
22. Joyce NC. Proliferative capacity of corneal endothelial cells. Exp Eye Res. 2012;95(1):16–23.
23. McAlister JC, Joyce NC, Harris DL, Ali RR, Larkin DFP. Induction of replication in human corneal endothelial cells by E2F2 transcription factor cDNA transfer. Investig Ophthalmol Vis Sci. 2005;46(10):3597–603.
24. Wilson SE, Weng J, Blair S, He YG, Lloyd S. Expression of E6/E7 or SV40 large T antigen-coding oncogenes in human corneal endothelial cells indicates regulated high-proliferative capacity. Investig Ophthalmol Vis Sci. 1995;36(1):32–40.
25. Wilson SE, Lloyd SA, He YG, McCash CS. Extended life of human corneal endothelial cells transfected with the SV40 large T antigen. Investig Ophthalmol Vis Sci. 1993;34(6):2112–23.
26. Feizi S. Corneal endothelial cell dysfunction: etiologies and management. Ther Adv Ophthalmol. 2018;10:2515841418815802.
27. Gain P, Jullienne R, He Z, Aldossary M, Acquart S, Cognasse F, et al. Global survey of corneal transplantation and eye banking. JAMA Ophthalmol. 2016;134(2):167–73.
28. Bourne WM. Biology of the corneal endothelium in health and disease. Eye. 2003;17(8):912–8.
29. Elhalis H, Azizi B, Jurkunas U V. Fuchs endothelial corneal dystrophy. Ocul Surf. 2010;8(4):173–84.
30. Jurkunas U V, Bitar M, Rawe I. Colocalization of Increased Transforming Growth Factor- Beta – Induced Protein ( TGFBIp ) and Clusterin in Fuchs Endothelial Corneal Dystrophy. 2009;50(3):1–8.
31. Ramboer E, De Craene B, De Kock J, Vanhaecke T, Berx G, Rogiers V, et al. Strategies for immortalization of primary hepatocytes. J Hepatol. 2014;61(4):925–43.
32. Wang Y, Chen S, Yan Z, Pei M. A prospect of cell immortalization combined with matrix microenvironmental optimization strategy for tissue engineering and regeneration 06 Biological Sciences 0601 Biochemistry and Cell Biology. Cell Biosci. 2019;9(1):1–21.
33. Schmedt T, Chen Y, Nguyen TT, Li S, Bonanno JA, Jurkunas U V. Telomerase Immortalization of Human Corneal Endothelial Cells Yields Functional Hexagonal Monolayers. PLoS One. 2012;7(12):1–11.
34. Salmon P, Oberholzer J, Occhiodoro T, Morel P, Lou J, Trono D. Reversible immortalization of human primary cells by lentivector-mediated transfer of specific genes. Mol Ther. 2000;2(4):404–14.
35. Chira S, Jackson CS, Oprea I, Ozturk F, Pepper MS, Diaconu I, et al. Progresses towards safe and efficient gene therapy vectors. Oncotarget. 2015;6(31):30675–703.
36. Ginn SL, Amaya AK, Alexander IE, Edelstein M, Abedi MR. Gene therapy clinical trials worldwide to 2017: An update. J Gene Med. 2018;20(5).
37. Sakuma T, Barry MA, Ikeda Y. Lentiviral vectors: Basic to translational. Biochem J. 2012;443(3):603–18.
38. Naldini L, Blömer U, Gallay P, Ory D, Mulligan R, Gage FH, et al. In vivo gene delivery and stable transduction of nondividing cells by a lentiviral vector. Science (80- ). 1996;272(5259):263–7.
39. Zufferey R, Nagy D, Mandel RJ, Naldini L, Trono D. Multiply attenuated lentiviral vector achieves efficient gene delivery in vivo. Nat Biotechnol. 1997;15(9):871–5.
40. Dull T, Zufferey R, Kelly M, Mandel RJ, Nguyen M, Trono D, et al. A third-generation lentivirus vector with a conditional packaging system. J Virol. 1998;72(11):8463–71.
41. Yu SF, von Ruden T, Kantoff PW, Garber C, Seiberg M, Rüther U, et al. Self-inactivating retroviral vectors designed for transfer of whole genes into mammalian cells. Proc Natl Acad Sci U S A. 1986;83(10):3194–8.
42. Schlimgen R, Howard J, Wooley D, Thompson M, Baden LR, Yang OO, et al. Risks associated with lentiviral vector exposures and prevention strategies. J Occup Environ Med. 2016;58(12):1159–66.
43. Hoess RH, Wierzbicki A, Abremski K. The role of the loxP spacer region in PI site-specific recombination. Nucleic Acids Res. 1986;14(5):2287–300.
44. Abremski K, Hoess R, Sternberg N. Studies on the properties of P1 site-specific recombination: Evidence for topologically unlinked products following recombination. Cell. 1983;32(4):1301–11.
45. abm. Cre-Lox Recombination [Internet]. [cited 2019 Dec 19]. Available from: https://www.abmgood.com/marketing/knowledge_base/Cre-Lox_Recombination…
46. Izsvák Z, Ivics Z. Sleeping Beauty transposition: Biology and applications for molecular therapy. Mol Ther. 2004;9(2):147–56.
47. Fraser MJ, Ciszczon T, Elick T, Bauser C. Precise excision of TTAA-specific lepidopteran transposons piggyBac (IFP2) and tagalong (TFP3) from the baculovirus genome in cell lines from two species of Lepidoptera. Insect Mol Biol. 1996;5(2):141–51.
48. Yusa K. piggyBac transposon. In: Mobile DNA III. American Society of Microbiology; 2015. p. 875–92.
49. Vargas JE, Chicaybam L, Stein RT, Tanuri A, Delgado-Cañedo A, Bonamino MH. Retroviral vectors and transposons for stable gene therapy: Advances, current challenges and perspectives. J Transl Med. 2016;14(1):1–15.
50. Li X, Burnight ER, Cooney AL, Malani N, Brady T, Sander JD, et al. PiggyBac transposase tools for genome engineering. Proc Natl Acad Sci U S A. 2013;110(25).
51. Ding S, Wu X, Li G, Han M, Zhuang Y, Xu T. Efficient transposition of the piggyBac (PB) transposon in mammalian cells and mice. Cell. 2005;122(3):473–83.
52. Li MA, Turner DJ, Ning Z, Yusa K, Liang Q, Eckert S, et al. Mobilization of giant piggyBac transposons in the mouse genome. Nucleic Acids Res. 2011;39(22).
53. Li MA, Pettitt SJ, Eckert S, Ning Z, Rice S, Cadinanos J, et al. The piggyBac Transposon Displays Local and Distant Reintegration Preferences and Can Cause Mutations at Noncanonical Integration Sites. Mol Cell Biol. 2013;33(7):1317–30.
54. Gogol-Doring A, Ammar I, Gupta S, Bunse M, Miskey C, Chen W, et al. Genome-wide profiling reveals remarkable parallels between insertion site selection properties of the MLV retrovirus and the piggyBac transposon in primary human CD4+ T cells. Mol Ther. 2016;24(3):592–606.
55. Fang SY, Hu CQ, Liu MN, Tao L, Wang Y, Gong MJ, et al. Reversibly immortalized hepatic progenitor cell line containing double suicide genes. Int J Mol Med. 2018;42(4):1977–86.
56. Jones BS, Lamb LS, Goldman F, Di Stasi A. Improving the safety of cell therapy products by suicide gene transfer. Front Pharmacol. 2014;5(November):1–8.
57. Vassaux G. Use of suicide genes for cancer gene therapy : study of the. Expert Opin Biol Ther. 2004;4(4):519–30.
58. Yu S, Yi M, Qin S, Wu K. Next generation chimeric antigen receptor T cells: Safety strategies to overcome toxicity. Mol Cancer. 2019;18(1).
59. Springer CJ, Niculescu-Duvaz I. Gene-directed enzyme prodrug therapy (GDEPT): choice of prodrugs. Adv Drug Deliv Rev. 1996;22(3):351–64.
60. Zhang J, Kale V, Chen M. Gene-Directed Enzyme Prodrug Therapy. AAPS J. 2015;17(1):102–10.
61. Karjoo Z, Chen X, Hatefi A. Progress and problems with the use of suicide genes for targeted cancer therapy. Adv Drug Deliv Rev. 2016;99:113–28.
62. MolMed SpA. Assessment report Zalmoxis [Internet]. 2016. Available from: https://www.ema.europa.eu/en/documents/assessment-report/zalmoxis-epar-…
63. MolMed SpA. MolMed Press Release [Internet]. 2019 [cited 2019 Dec 9]. Available from: https://www.molmed.com/sites/default/files/2019-10/PR_Zalmoxis update WITHDRAWAL_101019_DEF.pdf
64. European Commission. Union Register of not active medicinal products for human use [Internet]. 2019 [cited 2019 Dec 9]. Available from: https://ec.europa.eu/health/documents/community-register/html/h1121.htm
65. Fan L, Freeman KW, Khan T, Pham E, Spencer DM. Improved artificial death switches based on caspases and FADD. Hum Gene Ther. 1999;10(14):2273–85.
66. Gargett T, Brown MP. The inducible caspase-9 suicide gene system as a “safety switch” to limit on-target, off-tumor toxicities of chimeric antigen receptor T-cells. Front Pharmacol. 2014;5(OCT):1–7.
67. Kemper K, Rodermond H, Colak S, Grandela C, Medema JP. Targeting colorectal cancer stem cells with inducible caspase-9. Apoptosis. 2012;17(5):528–37.
68. Philip B, Kokalaki E, Mekkaoui L, Thomas S, Straathof K, Flutter B, et al. A highly compact epitope-based marker/suicide gene for easier and safer T-cell therapy. Blood. 2014;124(8):1277–87.
69. Wang X, Chang WC, Wong CLW, Colcher D, Sherman M, Ostberg JR, et al. A transgene-encoded cell surface polypeptide for selection, in vivo tracking, and ablation of engineered cells. Blood. 2011;118(5):1255–63.
70. Introna M, Barbui AM, Bambacioni F, Casati C, Gaipa G, Borleri G, et al. Genetic modification of human T cells with CD20: A strategy to purify and lyse transduced cells with anti-CD20 antibodies. Hum Gene Ther. 2000;11(4):611–20.
71. Rönkkö S, Vellonen KS, Järvinen K, Toropainen E, Urtti A. Human corneal cell culture models for drug toxicity studies. Drug Deliv Transl Res. 2016;6(6):660–75.
72. Peh GSL, Toh KP, Wu FY, Tan DT, Mehta JS. Cultivation of human corneal endothelial cells isolated from paired donor corneas. PLoS One. 2011;6(12).
73. Picot J, Guerin CL, Le Van Kim C, Boulanger CM. Flow cytometry: Retrospective, fundamentals and recent instrumentation. Cytotechnology. 2012;64(2):109–30.
74. Horizon Discovery. Dose response curve for antibiotic selection of mammalian cells (kill curve) [Internet]. Cambridge; 2018. p. 1. Available from: https://horizondiscovery.com/-/media/Files/Horizon/resources/Protocols/…
75. Barrett KC, Mendes RD, Smits WK, Wijk YMVH Van, Pieters R, Meijerink JPP. Lentiviral gene transfer into human and murine hematopoietic stem cells : size matters. BMC Res Notes. 2016;1–6.
76. Ikeda Y, Collins MKL, Radcliffe PA, Mitrophanous KA, Takeuchi Y. Gene transduction efficiency in cells of different species by HIV and EIAV vectors. Gene Ther. 2002;9(14):932–8.
77. Nguyen TH, Oberholzer J, Birraux J, Majno P, Morel P, Trono D. Highly efficient lentiviral vector-mediated transduction of nondividing, fully reimplantable primary hepatocytes. Mol Ther. 2002;6(2):199–209.
78. Parker DGA, Kaufmann C, Brereton HM, Anson DS, Jessup CF, Marshall K. Lentivirus-mediated gene transfer to the rat , ovine and human cornea. 2007;(14):760–7.
79. Oellig C, Seliger B. Gene Transfer Into Brain Tumor Cell Lines : Reporter Gene Expression Using Various Cellular and Viral Promoters. 1990;396:390–6.
80. Su R, Baylink DJ, Neises A, Kiroyan JB, Meng X, Payne KJ, et al. Efficient Generation of Integration-Free iPS Cells from Human Adult Peripheral Blood Using BCL-XL Together with Yamanaka Factors. 2013;8(5).
81. Benabdellah K, Cobo M, Muñoz P, Toscano MG, Martin F. Development of an all-in-one lentiviral vector system based on the original TetR for the easy generation of Tet-on cell lines. PLoS One. 2011;6(8).
82. Ellis J. Silencing and variegation of gammaretrovirus and lentivirus vectors. Hum Gene Ther. 2005;16(11):1241–6.
83. Lee CI, Kohn DB, Ekert JE, Tarantal AF. Morphological Analysis and Lentiviral Transduction of Fetal Monkey Bone Marrow-Derived Mesenchymal Stem Cells. Mol Ther. 2004;9(1):112–23.
84. Schott JW, Jaeschke NM, Hoffmann D, Maetzig T, Ballmaier M, Godinho T, et al. Deciphering the Impact of Parameters Influencing Transgene Expression Kinetics after Repeated Cell Transduction with Integration-Deficient Retroviral Vectors. 2015;405–18.
85. Baens M, Noels H, Broeckx V, Hagens S, Fevery S, Billiau AD, et al. The Dark Side of EGFP : Defective Polyubiquitination. 2006;2(1):1–6.
86. Hsueh YJ, Chen HC, Wu SE, Wang TK, Chen JK, Ma DHK. Lysophosphatidic acid induces YAP-promoted proliferation of human corneal endothelial cells via PI3K and ROCK pathways. Mol Ther - Methods Clin Dev. 2015;2(March):15014.
87. ThermoFisher. Puromycin [Internet]. [cited 2020 Apr 13]. Available from: https://www.thermofisher.com/be/en/home/life-science/cell-culture/trans…
88. ThermoFisher. Viral vector [Internet]. [cited 2019 Dec 29]. Available from: https://www.thermofisher.com/be/en/home/references/gibco-cell-culture-b…
89. Leibniz Institute DSMZ. HCEC-B4G12 [Internet]. [cited 2020 Jan 6]. Available from: https://www.dsmz.de/collection/catalogue/details/culture/ACC-647
90. Janson B. Encyclopedia of Ophthalmology. Encycl Ophthalmol. 2016;2006–7.
91. System Biosciences. PiggyBac Transposon Vector System: User Manual. Vol. 14. 2018.









