LINE-1 als humaan pathogeen
Het menselijke DNA is niet statisch, maar kan continu veranderen door de opeenstapeling van stukjes mobiel DNA. Binnen dit mobiel DNA zijn er verschillende klasses, waarvan retrotransposons de belangrijkste groep vormt. Deze zijn in staat om zichzelf te vermenigvuldigen via een kopieer‑en‑plak mechanisme. Long Interspersed Nuclear Element-1 (LINE-1) is het enige retrotransposon dat nog in staat is zichzelf te vermenigvuldigen. Ongecontroleerde LINE-1 retrotranspositie kan worden geassocieerd met heel wat neurologische aandoeningen, zoals Rett syndroom en Ataxia Telangiectasa. Echter, op dit moment is de specifieke rol van LINE-1 nog niet volledig gekend. Er is nood aan verder onderzoek naar de rol van gastheerfactoren en het proces van LINE-1 retrotranspositie. In mijn scriptie heb ik de rol van transportine-1 (TNPO1) en transportine-3 (TNPO3) in LINE-1 retrotranspositie onderzocht. In een tweede deel werd een assay geoptimaliseerd in een neuronale cellijn en in een laatste deel werden hersenen van muizen aangekleurd voor een proteïne van LINE-1.
1. Het proces van LINE-1 retrotranspositie
Ongeveer 17% van ons DNA bestaat uit LINE-1 kopieën, waarvan de meerderheid echter geen retrotranspositie meer kan ondergaan. Er zijn slechts 80 à 100 actieve kopieën aanwezig in ons DNA die kunnen bijdragen tot variatie. Een LINE-1 element, aanwezig in het menselijke DNA in de celkern, kan worden afgeschreven naar boodschapper RNA. Vervolgens wordt dit RNA naar het cytosol overgebracht, waar de info in het RNA wordt vertaald naar proteïnen. Dit proces resulteert in de expressie van 2 proteïnen, namelijk het ORF1 proteïne (ORF1p) en het ORF2 proteïne (ORF2p). Vervolgens wordt een complex gevormd bestaande uit het boodschapper RNA, het ORF1p en het ORF2p. Dit complex wordt terug naar de celkern gebracht en een nieuwe kopie van LINE-1 kan zich integreren in het DNA. Op deze manier is een nieuwe kopie van LINE-1 aanwezig in het menselijke DNA dat ook zichzelf kan kopiëren (Figuur 1).
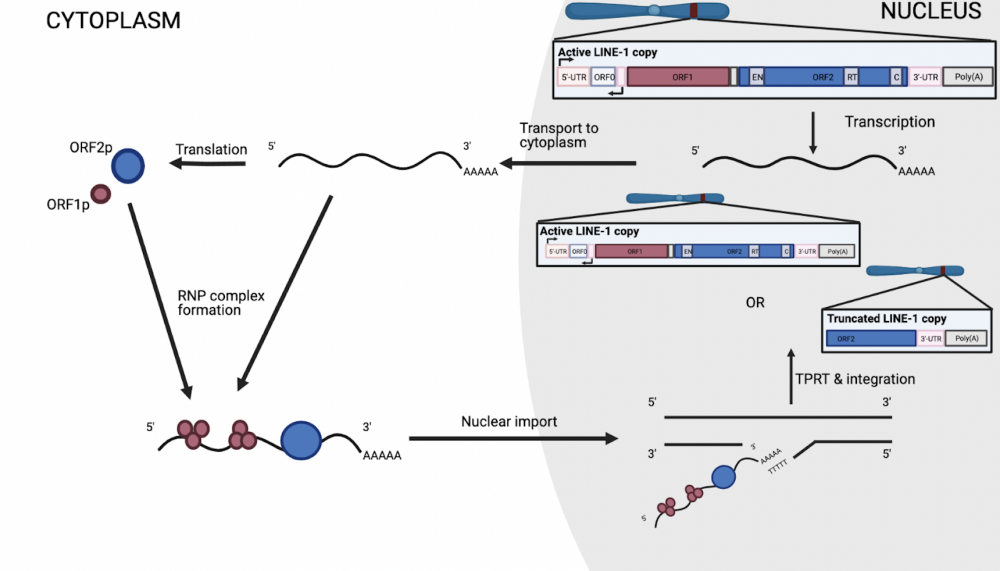
Figuur 1: Het proces van LINE-1 retrotranspositie
2. De LINE-1 retrotranspositie test
De LINE-1 retrotranspositie test laat toe om de rol van gastheerfactoren te onderzoeken in het proces van LINE-1 retrotranspositie. In deze test worden cellen getransfecteerd met een plasmide dat het LINE-1 element bevat (L1RP) gekoppeld aan een reporter, namelijk het EGFP proteïne. Nadat dit plasmide in de cellen is opgenomen, kan LINE-1 retrotranspositie plaatsvinden in de cel. Het EGFP proteïne laat toe om de retrotranspositie te visualiseren. Er zal enkel expressie zijn van het EGFP proteïne als er ook LINE-1 retrotranspositie optreedt in de cel. Als een controle in deze test, worden de cellen getransfecteerd met een LINE-1 plasmide die geen retrotranspositie meer kan ondergaan door mutatie (JM111).
3. Identificatie van TNPO1 en TNPO3 als gastheerfactor van LINE-1
Net zoals het HIV virus, heeft LINE-1 ook gastheerfactoren nodig in de gastcel om zich te kunnen vermenigvuldigen. In een eerste deel van mijn scriptie werd de rol van TNPO1 en TNPO3 onderzocht voor hun mogelijke rol in de import van het LINE-1 complex in de nucleus. Aan de hand van de LINE-1 retrotranspositie test werd nagegaan of beide een rol spelen in LINE-1 retrotranspositie. Er werden cellen gecreeërd waarin de expressie van het TNPO1 of het TNPO3 proteïne werd onderdrukt. Op deze manier werd een TNPO1 knockdown (KD) en TNPO3 KD cellijn gecreeërd. Deze cellijnen werden nadien getest in de LINE-1 retrotranspositie test en de expressie van het EGFP proteïne werd gevolgd voor alle condities. Er was een duidelijke daling in de expressie van het EGFP proteïne voor zowel de cellijn met minder TNPO1 als de cellijn met minder TNPO3 (Figuur 2). Deze resultaten lijken de hypothese te bevestigen, maar verder onderzoek is nodig naar hun specifieke rol in het proces.
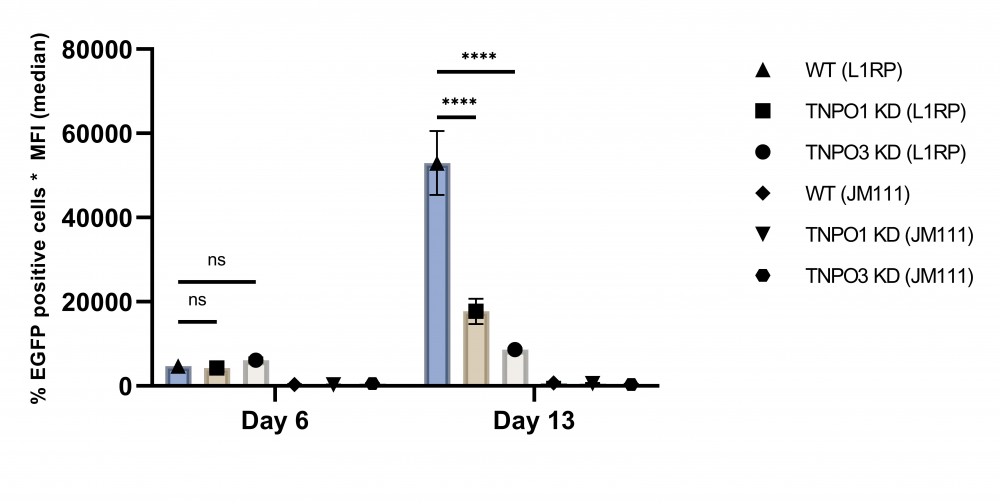
Figuur 2: Daling in EGFP expressie in TNPO1 en TNPO3 KD cellijnen
4. Optimalisatie van LINE-1 retrotranspositie test in een neuronale cellijn
In het verleden werd reeds aangetoond dat LINE-1 retrotranspositie kan optreden in de hersenen en kan bijdragen aan de ontwikkeling van neurologische aandoeningen. Omdat gastheerfactoren specifiek kunnen zijn voor bepaalde cellijnen, is het relevant om ook de LINE-1 retrotranspositie test te kunnen uitvoeren in een neuronale cellijn. In een tweede deel van mijn scriptie heb ik geprobeerd om de test te optimaliseren in SH-SY5Y cellen. Om de test succesvol te kunnen uitvoeren is het belangrijk dat de cellen het plasmide opnemen in hun cytosol/celkern. Er werden verschillende methodes gebruikt om dit te optimaliseren, maar geen enkele geteste methode was in staat om het plasmide binnen te brengen in deze SH-SY5Y cellen. Verder onderzoek dient uit te wijzen of er andere methoden bestaan die wel in staat zijn om het LINE-1 plasmide in de cel te brengen. Eens dit is bereikt, kan de test worden uitgevoerd in deze cellijn. Op deze manier kunnen ook gastheerfactoren specifiek voor een bepaald celtype geïdentificeerd worden.
5. Optimalisatie van kleuring voor het ORF1p
Met het oog op verder onderzoek naar de potentiële rol van LINE-1 retrotranspositie in neurologische aandoeningen, voornamelijk de pathogenese van de ziekte van Parkinson, is detectie van het ORF1p in de hersenen van de muis nodig. In een laatste deel van mijn scriptie heb ik zowel de immunohistochemische als de immunofluorescente kleuring voor het ORF1p geoptimaliseerd op hersencoupes van wild-type muizen. Bovendien laat de optimalisatie van de immunofluorescente kleuring toe om een dubbele kleuring uit te voeren. Wanneer een dubbele kleuring werd uitgevoerd voor tyrosine hydroxylase (TH), kenmerkend voor dopaminerge neuronen, en het ORF1p bleek een hoge expressie van het ORF1p in de TH positieve cellen in de substantia nigra (Figuur 3). De optimalisatie van de 2 verschillende kleuringen in wild-type muizen laat toe om andere muismodellen te screenen voor hun expressie van het ORF1p.
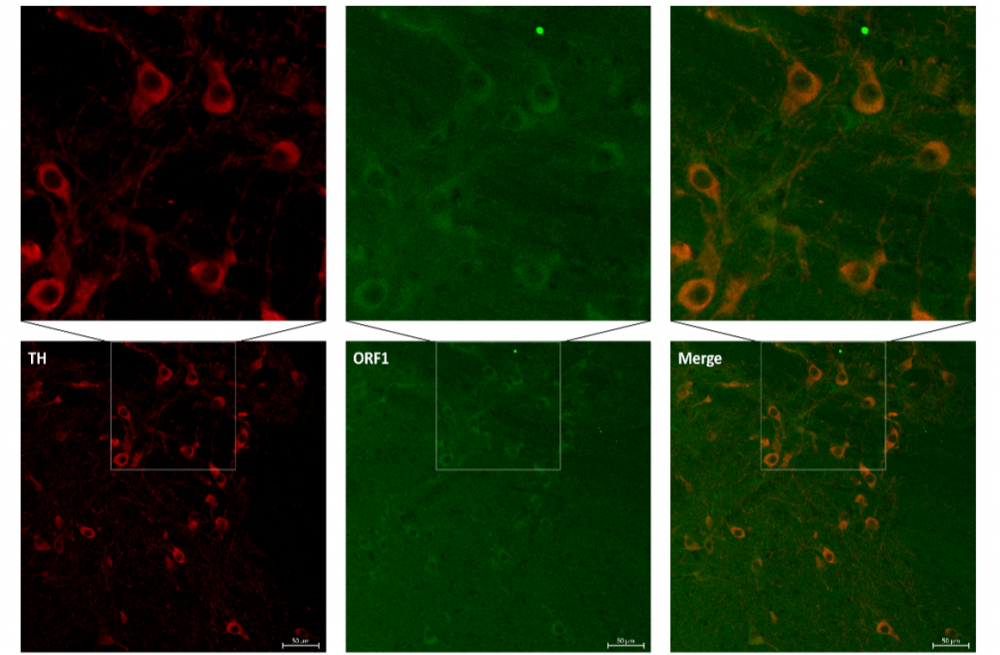
Figuur 3: Expressie van TH en ORF1p in substantia nigra
6. Conclusie
Met mijn scriptie heb ik bijgedragen aan het basisonderzoek naar LINE-1 retrotranspositie. Er is echter een nood aan verder onderzoek naar de betrekking van gastheerfactoren. Ook dient de rol van LINE-1 in de ontwikkeling van neurologische aandoeningen verder onderzocht te worden. Directe onderdrukking van LINE-1 of indirecte onderdrukking via inhibitie van gastheerfactoren, kan mogelijks leiden tot potentiële behandelingsstrategieën van neurologische aandoeningen.
Bibliografie
-
Elbarbary RA, Lucas BA, et al. Retrotransposons as regulators of gene expression. Science. 2016;351(6274).
-
Kazazian HH, Moran JV. Mobile DNA in Health and Disease. N Engl J Med. 2017;377(4):361- 70.
-
Cordaux R, Batzer MA. The impact of retrotransposons on human genome evolution. Nat Rev Genet. 2009;10(10):691-703.
-
Hancks DC, Kazazian H. Roles for retrotransposon insertions in human disease. Mob DNA. 2016;7(1):9.
-
McClintock B. The origin and behavior of mutable loci in maize. Genetics. 1950;36:344-55.
-
Dombroski B, Mathias S, et al. Isolation of an active human transposable element. Science.
1991;254(5039):1805-8.
-
Munoz-Lopez M, Garcia-Perez JL. DNA transposons: nature and applications in genomics.
Curr Genomics. 2010;11:115-28.
-
Saleh A, Macia A, et al. Transposable Elements, Inflammation, and Neurological Disease.
Front Neurol. 2019;10:894.
-
Scott EC, Devine SE. The Role of Somatic L1 Retrotransposition in Human Cancers. Viruses.
2017;9(6).
-
Brouha B, Schustak J, et al. Hot L1s account for the bulk of retrotransposition in the human
population. PNAS. 2003;100(9):5280-5.
-
Krestel H, Meier JC. RNA Editing and Retrotransposons in Neurology. Front Mol Neurosci.
2018;11:163.
-
Rice GI, Meyzer C, et al. Reverse-Transcriptase Inhibitors in the Aicardi–Goutières Syndrome.
N Engl J Med. 2018;379(23):2275-7.
-
Ariumi Y. Guardian of the Human Genome: Host Defense Mechanisms against LINE-1
Retrotransposition. Front Chem. 2016;4:28.
-
Beck CR, Garcia-Perez JL, et al. LINE-1 elements in structural variation and disease. Annu Rev
Genomics Hum Genet. 2011;12:187-215.
-
Denli AM, Narvaiza I, et al. Primate-specific ORF0 contributes to retrotransposon-mediated
diversity. Cell. 2015;163(3):583-93.
-
Lavie L, Maldener E, et al. The human L1 promoter: variable transcription initiation sites and
a major impact of upstream flanking sequence on promoter activity. Genome Res.
2004;14(11):2253-60.
-
Richardson SR, Doucet AJ, et al. The Influence of LINE-1 and SINE Retrotransposons on
Mammalian Genomes. Microbiol Spectr. 2015;3(2).
-
Terry DM, Devine SE. Aberrantly High Levels of Somatic LINE-1 Expression and
Retrotransposition in Human Neurological Disorders. Front Genet. 2019;10:1244.
-
Muotri AR, Chu VT, et al. Somatic mosaicism in neuronal precursor cells mediated by L1
retrotransposition. Nature. 2005;435(7044):903-10.
-
Muotri AR, Zhao C, et al. Environmental influence on L1 retrotransposons in the adult
hippocampus. Hippocampus. 2009;19(10):1002-7.
-
Coufal NG, Garcia-Perez JL, et al. L1 retrotransposition in human neural progenitor cells.
Nature. 2009;460(7259):1127-31.
-
Suarez NA, Macia A, et al. LINE-1 retrotransposons in healthy and diseased human brain. Dev
Neurobiol. 2018;78(5):434-55.
-
Macia A, Widmann TJ, et al. Engineered LINE-1 retrotransposition in nondividing human
neurons. Genome Res. 2017;27(3):335-48.
-
Baillie JK, Barnett MW, et al. Somatic retrotransposition alters the genetic landscape of the
human brain. Nature. 2011;479(7374):534-7.
47
-
Evrony GD, Cai X, et al. Single-neuron sequencing analysis of L1 retrotransposition and somatic mutation in the human brain. Cell. 2012;151(3):483-96.
-
Upton KR, Gerhardt DJ, et al. Ubiquitous L1 mosaicism in hippocampal neurons. Cell. 2015;161(2):228-39.
-
Kopera HC, Larson PA, et al. LINE-1 Cultured Cell Retrotransposition Assay. Methods Mol Biol. 2016;1400:139-56.
-
Rangwala SH, Kazazian HH, Jr. The L1 retrotransposition assay: a retrospective and toolkit. Methods. 2009;49(3):219-26.
-
Moran JV, Holmes SE, et al. High Frequency Retrotransposition in Cultured Mammalian Cells. Cell Press. 1996;87:917-27.
-
Raiz J, Damert A, et al. The non-autonomous retrotransposon SVA is trans-mobilized by the human LINE-1 protein machinery. Nucleic Acids Res. 2012;40(4):1666-83.
-
Ostertag EM, Luning ET, et al. Determination of L1 retrotransposition kinetics in cultured cells. Nucleic Acids Research. 2000;28(6):1418-23.
-
Del Re B, Marcantonio P, et al. Evaluation of LINE-1 mobility in neuroblastoma cells by in vitro retrotransposition reporter assay: FACS analysis can detect only the tip of the iceberg of the inserted L1 elements. Exp Cell Res. 2010;316(20):3358-67.
-
Kempen MHC, Bodea GO, et al. Neuronal Genome Plasticity: Retrotransposons, Environment and Disease. In: Cristofari G, editor. Human Retrotransposons in Health and Disease. Cham: Springer International Publishing; 2017. p. 107-25.
-
Sanchez-Luque FJ, Richardson SR, et al. Retrotransposon Capture Sequencing (RC-Seq): A Targeted, High-Throughput Approach to Resolve Somatic L1 Retrotransposition in Humans. In: Garcia-Pérez JL, editor. Transposons and Retrotransposons: Methods and Protocols. New York, NY: Springer New York; 2016. p. 47-77.
-
Tausig F, Wolf FJ. Streptavidin - a substance with avidin-like properties produced by microorganisms. Biochem Biophys Res Commun. 1964;14(3):205-9.
-
Thomas CA, Paquola AC, et al. LINE-1 retrotransposition in the nervous system. Annu Rev Cell Dev Biol. 2012;28:555-73.
-
Graham V, Khudyakov J, et al. SOX2 Functions to Maintain Neural Progenitor Identity. Neuron. 2003;39(5):749-65.
-
Muotri AR, Marchetto MC, et al. L1 retrotransposition in neurons is modulated by MeCP2. Nature. 2010;468(7322):443-6.
-
Lennartsson A, Arner E, et al. Remodeling of retrotransposon elements during epigenetic induction of adult visual cortical plasticity by HDAC inhibitors. Epigenetics & Chromatin. 2015;8(1).
-
Van Meter M, Kashyap M, et al. SIRT6 represses LINE1 retrotransposons by ribosylating KAP1 but this repression fails with stress and age. Nat Commun. 2014;5:5011.
-
Tedeschi A, Di Giovanni S. The non-apoptotic role of p53 in neuronal biology: enlightening the dark side of the moon. EMBO Rep. 2009;10(6):576-83.
-
Wylie A, Jones AE, et al. p53 genes function to restrain mobile elements. Genes Dev. 2016;30(1):64-77.
-
Kuwabara T, Hsieh J, et al. Wnt-mediated activation of NeuroD1 and retro-elements during adult neurogenesis. Nat Neurosci. 2009;12(9):1097-105.
-
Athanikar JN, Badge RM, et al. A YY1-binding site is required for accurate human LINE-1 transcription initiation. Nucleic Acids Res. 2004;32(13):3846-55.
-
Tchénio T, Casella JF, et al. Members of the SRY family regulate the human LINE retrotransposons. Nucleic Acids Res. 2000;28(2):411-5.
-
Yang N, Zhang L, et al. An important role for RUNX3 in human L1 transcription and retrotransposition. Nucleic Acids Res. 2003;31(16):4929-40.
-
Shi X, Seluanov A, et al. Cell divisions are required for L1 retrotransposition. Mol Cell Biol. 2007;27(4):1264-70.
48
-
Idica A, Sevrioukov EA, et al. MicroRNA miR-128 represses LINE-1 (L1) retrotransposition by down-regulating the nuclear import factor TNPO1. J Biol Chem. 2017;292(50):20494-508.
-
Twyffels L, Gueydan C, et al. Transportin-1 and Transportin-2: protein nuclear import and
beyond. FEBS Lett. 2014;588(10):1857-68.
-
Kataoka N, Bachorik JL, et al. Transportin-SR, a nuclear import receptor for SR proteins. J Cell
Biol. 1999;145(6):1145-52.
-
Lai MC, Lin RI, et al. A human importin-beta family protein, transportin-SR2, interacts with
the phosphorylated RS domain of SR proteins. J Biol Chem. 2000;275(11):7950-7.
-
Fu X. The superfamily of arginine/serine-rich splicing factors. RNA. 1995;1(7):663-80.
-
Christ F, Thys W, et al. Transportin-SR2 imports HIV into the nucleus. Curr Biol.
2008;18(16):1192-202.
-
Fernandez J, Machado AK, et al. Transportin-1 binds to the HIV-1 capsid via a nuclear
localization signal and triggers uncoating. Nat Microbiol. 2019;4(11):1840-50.
-
Zhang X, Zhang R, et al. New Understanding of the Relevant Role of LINE-1 Retrotransposition
in Human Disease and Immune Modulation. Front Cell Dev Biol. 2020;8:657.
-
Savitsky K, Bar-Shira A, et al. A single ataxia telangiectasia gene with a product similar to PI-3
kinase. Science. 1995;268(5218):1749-53.
-
Bar-Shira A, Rashi-Elkeles S, et al. ATM-dependent activation of the gene encoding MAP
kinase phosphatase 5 by radiomimetic DNA damage. Oncogene. 2002;21(5):849-55.
-
Coufal NG, Garcia-Perez JL, et al. Ataxia telangiectasia mutated (ATM) modulates long
interspersed element-1 (L1) retrotransposition in human neural stem cells. PNAS.
2011;108(51):20382-7.
-
Blennow K, de Leon MJ, et al. Alzheimer's disease. The Lancet. 2006;368(9533):387-403.
-
Tam OH, Ostrow LW, et al. Diseases of the nERVous system: retrotransposon activity in
neurodegenerative disease. Mob DNA. 2019;10:32.
-
Bollati V, Galimberti D, et al. DNA methylation in repetitive elements and Alzheimer disease.
Brain Behav Immun. 2011;25(6):1078-83.
-
Hernández HG, Mahecha MF, et al. Global Long Interspersed Nuclear Element 1 DNA
Methylation in a Colombian Sample of Patients With Late-Onset Alzheimer’s Disease. Am J
Alzheimers Dis Other Demen. 2013;29(1):50-3.
-
Searles Nielsen S, Checkoway H, et al. LINE-1 DNA methylation, smoking and risk of
Parkinson's disease. J Parkinsons Dis. 2012;2(4):303-8.
-
Davie CA. A review of Parkinson's disease. Br Med Bull. 2008;86:109-27.
-
Desplats P, Spencer B, et al. Alpha-synuclein sequesters Dnmt1 from the nucleus: a novel
mechanism for epigenetic alterations in Lewy body diseases. J Biol Chem. 2011;286(11):9031-
7.
-
Di Lorenzo Alho AT, Suemoto CK, et al. Three-dimensional and stereological characterization
of the human substantia nigra during aging. Brain Struct Funct. 2016;221(7):3393-403.
-
Blaudin de The FX, Rekaik H, et al. Engrailed homeoprotein blocks degeneration in adult
dopaminergic neurons through LINE-1 repression. EMBO J. 2018;37(15).
-
Rekaik H, Blaudin de Thé F-X, et al. Engrailed Homeoprotein Protects Mesencephalic
Dopaminergic Neurons from Oxidative Stress. Cell Rep. 2015;13(2):242-50.
-
Sonnier L, Le Pen G, et al. Progressive loss of dopaminergic neurons in the ventral midbrain
of adult mice heterozygote for Engrailed1. J Neurosci. 2007;27(5):1063-71.
-
Zhang XM, Yin M, et al. Cell-based assays for Parkinson's disease using differentiated human
LUHMES cells. Acta Pharmacol Sin. 2014;35(7):945-56.
-
Scholz D, Poltl D, et al. Rapid, complete and large-scale generation of post-mitotic neurons
from the human LUHMES cell line. J Neurochem. 2011;119(5):957-71.
-
Baeken MW, Moosmann B, et al. Retrotransposon activation by distressed mitochondria in
neurons. Biochem Biophys Res Commun. 2020;525(3):570-5.
49
-
Pfaff AL, Bubb VJ, et al. An Increased Burden of Highly Active Retrotransposition Competent L1s Is Associated with Parkinson's Disease Risk and Progression in the PPMI Cohort. Int J Mol Sci. 2020;21(18).
-
Dai L, Huang Q, et al. Effect of reverse transcriptase inhibitors on LINE-1 and Ty1 reverse transcriptase activities and on LINE-1 retrotransposition. BMC Biochem. 2011;12:18.
-
Nguyen Van Nhien A, Tomassi C, et al. First Synthesis and Evaluation of the Inhibitory Effects of Aza Analogues of TSAO on HIV-1 Replication. J Med Chem. 2005;48:4276-84.
-
Arts EJ, Wainberg MA. Mechanisms of Nucleoside Analog Antiviral Activity and Resistance during Human Immunodeficiency Virus Reverse Transcription. Antimicrob Agents Chemother. 1996;40(3):527-40.
-
De Clercq E. HIV resistance to reverse transcriptase inhibitors. Biochem Pharmacol. 1994;47(2):155-69.
-
De Clercq E. HIV-1-specific RT inhibitors: Highly selective inhibitors of human immunodeficiency virus type 1 that are specifically targeted at the viral reverse transcriptase. Med Res Rev. 1993;13(3):229-58.
-
Merluzzi V, Hargrave K, et al. Inhibition of HIV-1 replication by a nonnucleoside reverse transcriptase inhibitor. Science. 1990;250(4986):1411-3.
-
Sluis-Cremer N, Tachedjian G. Mechanisms of inhibition of HIV replication by non-nucleoside reverse transcriptase inhibitors. Virus Res. 2008;134(1-2):147-56.
-
Jones RB, Garrison KE, et al. Nucleoside analogue reverse transcriptase inhibitors differentially inhibit human LINE-1 retrotransposition. PLoS One. 2008;3(2):e1547.
-
Banuelos-Sanchez G, Sanchez L, et al. Synthesis and Characterization of Specific Reverse Transcriptase Inhibitors for Mammalian LINE-1 Retrotransposons. Cell Chem Biol. 2019;26(8):1095-109
-
Crow YJ, Manel N. Aicardi-Goutieres syndrome and the type I interferonopathies. Nat Rev Immunol. 2015;15(7):429-40.
-
Crow YJ, Hayward BE, et al. Mutations in the gene encoding the 3'-5' DNA exonuclease TREX1 cause Aicardi-Goutieres syndrome at the AGS1 locus. Nat Genet. 2006;38(8):917-20.
-
Rice GI, Bond J, et al. Mutations involved in Aicardi-Goutieres syndrome implicate SAMHD1 as regulator of the innate immune response. Nat Genet. 2009;41(7):829-32.
-
Reyniers L, Del Giudice MG, et al. Differential protein-protein interactions of LRRK1 and LRRK2 indicate roles in distinct cellular signaling pathways. J Neurochem. 2014;131(2):239- 50.
-
Sariyer IK. Transfection of neuronal cultures. Methods Mol Biol. 2013;1078:133-9.
-
Wiesenhofer B, Kaufmann WA, et al. Improved lipid-mediated gene transfer in C6 glioma
cells and primary glial cells using FuGENETM. J Neurosci Methods. 1999;92:145-52.
-
Kovalevich J, Langford D. Considerations for the use of SH-SY5Y neuroblastoma cells in
neurobiology. Methods Mol Biol. 2013;1078:9-21.
-
Gresch O, Engel FB, et al. New non-viral method for gene transfer into primary cells.
Methods. 2004;33(2):151-63.
-
Hamdorf M, Idica A, et al. miR-128 represses L1 retrotransposition by binding directly to L1
RNA. Nat Struct Mol Biol. 2015;22(10):824-31.
-
Kubo S, Seleme MdC, et al. L1 retrotransposition in nondividing and primary human somatic
cells. Proceedings of the National Academy of Sciences. 2006;103:8036-41.
-
Di Guardo G. Lipofuscin, lipofuscin-like pigments and autofluorescence. Eur J Histochem.
2015;59(1):2485.
-
Terman A, Brunk UT. Lipofuscin: Mechanisms of formation and increase with age. APMIS.
1998;106:265-76.
-
Goodier JL, Ostertag EM, et al. A novel active L1 retrotransposon subfamily in the mouse.
Genome Res. 2001;11(10):1677-85.