Virussen in de strijd tegen antibioticaresistentie
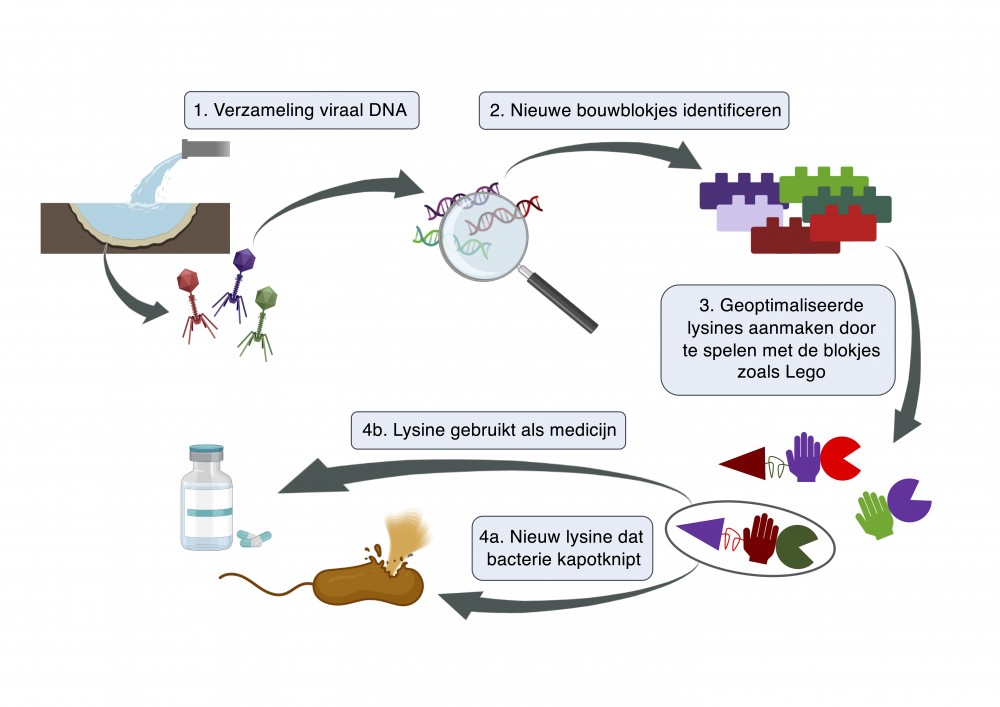
Antibioticaresistentie wordt gevreesd tot tien miljoen doden per jaar te veroorzaken tegen 2050. Het is dus absoluut van mondiaal belang om alternatieve antibiotica te ontwikkelen. Een zeer beloftevol alternatief zijn “lysines”. Deze lysines worden geproduceerd door virussen die bacteriën doden, bacteriofagen, of ook wel kortweg fagen genoemd. Met een techniek die vergeleken kan worden met Legoblokjes zal het mogelijk zijn om geoptimaliseerde lysines te maken die ingezet kunnen worden tegen tal van gevaarlijke bacteriën.
Antibioticaresistentie
Ieder van ons heeft het voorbije jaar ervaren welke gevolgen infectieziekten kunnen hebben op mens en maatschappij. De ziekte COVID-19 wordt dan wel veroorzaakt door een virale infectie, ook bacteriële infecties kunnen grote gevolgen hebben. Terwijl het einde van de COVID-19 pandemie in zicht is, neemt de dreiging van bacteriële infecties steeds toe. Heel lang konden bacteriële infecties onder controle gehouden worden dankzij antibiotica waardoor al miljoenen levens gered werden. Helaas hebben vele bacteriën resistentie ontwikkeld tegen deze antibiotica, waardoor ze minder goed of zelfs helemaal niet meer werken. Hierdoor vormen resistente bacteriën een wereldwijde bedreiging voor de volksgezondheid. Indien er geen actie wordt ondernomen zullen ze in 2050 tot tien miljoen doden per jaar veroorzaken. Dit is zelfs meer dan het aantal overlijdens door het coronavirus of door kanker.
Er zijn twee redenen voor de grote stijging van het sterftecijfer ten gevolge van antibioticaresistente bacteriën. Vroeger werden er veel nieuwe antibioticaklassen ontdekt, voornamelijk in de jaren 50 en 60, maar de laatste decennia vinden wetenschappers geen nieuwe antibiotica. Daarbovenop blijven ook het aantal resistente bacteriën en het aantal verschillende resistentiemechanismen toenemen. Er is dus absoluut nood aan alternatieve klassen van antibiotica met nieuwe werkingsmechanismen om dit probleem aan te pakken.
Lysines als alternatief voor klassieke antibiotica
Bacteriofaag-gecodeerde lysines hebben bijzonder veel potentieel als een nieuwe klasse van antibiotica. Deze lysines worden in de natuur geproduceerd door bacteriofagen. Dit zijn virussen die bacteriën infecteren. De bacteriofagen dringen de bacteriecel binnen waarna de bacteriën zullen functioneren als ware bacteriofaag-productiemachines. Op het einde van de infectiecyclus zullen de nieuwgevormde bacteriofagen de bacteriën van binnenuit doen openbarsten, waardoor ze worden weggeslingerd op zoek naar nieuwe bacteriële gastheren. De schaartjes waarmee de bacteriofagen zorgen voor het openbarsten van de bacterie worden lysines genoemd. Als lysines de ziekteverwekkende bacterie kunnen aanvallen langs de buitenkant, kunnen ze ook gebruikt worden als een echt medicijn. Zoals het oude gezegde luidt: het is slim om vrienden te worden met de vijand (de bacteriofaag) van onze vijand (de bacteriën).
Spelen met Legoblokjes
Lysines bestaan uit verschillende modules. Zo’n module kan vergeleken worden met een Legoblokje. Elk Legoblokje heeft een functie, het zorgt bijvoorbeeld voor specificiteit of efficiëntie. Met de VersaTile techniek kan er vlot met deze Legoblokjes gespeeld worden. Je kiest welke blokjes je zal gebruiken en op welke positie ze moeten gezet worden. Vervolgens worden de blokjes gecombineerd en worden er lysines aangemaakt waarbij de eigenschappen van het lysine geoptimaliseerd worden.
Nieuwe Legoblokjes identificeren
Om vele verschillende bouwblokjes te vinden moeten er nieuwe natuurlijke lysines geïdentificeerd worden. Een mogelijke benadering hiervoor is gebruik te maken van functional metagenomics. Deze term bestaat uit twee delen. Metagenomics omvat het bestuderen van genetisch materiaal rechtstreeks gewonnen uit een omgevingsstaal. Dit biedt het enorme voordeel dat ook ongekende bacteriofagen onder de loep worden genomen. De momenteel gekende bacteriofagen zijn immers maar het topje van de ijsberg. Een grote potentiële visvijver van nieuwe lysines met uitstekende eigenschappen kan dus aangeboord worden. In het verkregen viraal DNA kan dan gezocht worden naar lysines. Met een functionele screening kunnen er volledig nieuwe lysines gevonden worden. De detectie gebeurt op basis van functie: enkel diegene die de gewenste functie vertonen, in dit geval dus het doden van gevaarlijke bacteriën, zullen geselecteerd worden.
Stappenplan om lysines te ontdekken
We hebben in ons onderzoek de basis gelegd voor een ambitieuze aanpak om met hoge efficiëntie uitgebreid metagenomen te kunnen screenen voor lysines. Verschillende stappen voor het bereiden van de metagenomen en het detecteren van lysines werden onder de loep genomen en geoptimaliseerd. De resultaten vormen een veelbelovende toekomst voor de ontwikkeling van nieuwe lysines. En wie weet liggen binnenkort de eerste lysine-medicijnen op de plank in jouw apotheek.
Bibliografie
ADDIN Mendeley Bibliography CSL_BIBLIOGRAPHY AAT Bioquest. (n.d.). Fluorescein dicaprylate WS1. Retrieved May 4, 2021, from https://www.aatbio.com/products/fluorescein-dicaprylate-ws1
Adams, M. H. (1959). Bacteriophages and Their Structural Organisation. Interscience publishers. www.intechopen.com
Agilent. (2015). Instruction Manual: BL21-CodonPlus Competent Cells.
Alav, I., Sutton, J. M., & Rahman, K. M. (2018). Role of bacterial efflux pumps in biofilm formation. Journal of Antimicrobial Chemotherapy, 73(8), 2003–2020. https://doi.org/10.1093/jac/dky042
Bai, J., Lee, S., & Ryu, S. (2020). Identification and in vitro characterization of a novel phage endolysin that targets gram-negative bacteria. Microorganisms, 8(3). https://doi.org/10.3390/microorganisms8030447
Baret, J. C., Miller, O. J., Taly, V., Ryckelynck, M., El-Harrak, A., Frenz, L., Rick, C., Samuels, M. L., Hutchison, J. B., Agresti, J. J., Link, D. R., Weitz, D. A., & Griffiths, A. D. (2009). Fluorescence-activated droplet sorting (FADS): Efficient microfluidic cell sorting based on enzymatic activity. Lab on a Chip, 9(13), 1850–1858. https://doi.org/10.1039/b902504a
Berg, E. G. (2015, April 28). A New Spin On The Old Gram Stain. C&en. https://cen.acs.org/articles/93/web/2015/04/New-Spin-Old-Gram-Stain.html
Bozdogan, B., & Appelbaum, P. C. (2004). Oxazolidinones: Activity, mode of action, and mechanism of resistance. In International Journal of Antimicrobial Agents (Vol. 23, Issue 2, pp. 113–119). Elsevier. https://doi.org/10.1016/j.ijantimicag.2003.11.003
Briers, Y. (2020a). Directed evolution of proteins. In Biochemische en moleculaire analyse. Universiteit Gent.
Briers, Y. (2020b). Phage-inspired antibiotics. In Biochemische en moleculaire analyse. Universiteit Gent.
Briers, Y., Peeters, L. M., Volckaert, G., & Lavigne, R. (2011). The lysis cassette of bacteriophage фKMV encodes a signal-arrest-release endolysin and a pinholin. Bacteriophage, 1(1), 25–30. https://doi.org/10.4161/bact.1.1.14868
Briers, Y., Volckaert, G., Cornelissen, A., Lagaert, S., Michiels, C. W., Hertveldt, K., & Lavigne, R. (2007). Muralytic activity and modular structure of the endolysins of Pseudomonas aeruginosa bacteriophages φKZ and EL. Molecular Microbiology, 65(5), 1334–1344. https://doi.org/10.1111/j.1365-2958.2007.05870.x
Briers, Y., Walmagh, M., Grymonprez, B., Biebl, M., Pirnay, J. P., Defraine, V., Michiels, J., Cenens, W., Aertsen, A., Miller, S., & Lavigne, R. (2014). Art-175 is a highly efficient antibacterial against multidrug-resistant strains and persisters of Pseudomonas aeruginosa. Antimicrobial Agents and Chemotherapy, 58(7), 3774–3784. https://doi.org/10.1128/AAC.02668-14
Briers, Y., Walmagh, M., Van Puyenbroeck, V., Cornelissen, A., Cenens, W., Aertsen, A., Oliveira, H., Azeredo, J., Verween, G., Pirnay, J. P., Miller, S., Volckaert, G., & Lavigne, R. (2014). Engineered endolysin-based “Artilysins” to combat multidrug-resistant gram-negative pathogens. MBio, 5(4). https://doi.org/10.1128/mBio.01379-14
Brochet, M., Couvé, E., Zouine, M., Poyart, C., & Glaser, P. (2008). A naturally occurring gene amplification leading to sulfonamide and trimethoprim resistance in Streptococcus agalactiae. Journal of Bacteriology, 190(2), 672–680. https://doi.org/10.1128/JB.01357-07
Bush, K., & Bradford, P. A. (2016). β-lactams and β-lactamase inhibitors: An overview. Cold Spring Harbor Perspectives in Medicine, 6(8), a025247. https://doi.org/10.1101/cshperspect.a025247
Capasso, C., & Supuran, C. T. (2014). Sulfa and trimethoprim-like drugs-antimetabolites acting as carbonic anhydrase, dihydropteroate synthase and dihydrofolate reductase inhibitors. Journal of Enzyme Inhibition and Medicinal Chemistry, 29(3), 379–387. https://doi.org/10.3109/14756366.2013.787422
Centers for Disease Control and Prevention. (2019). Antibiotic Resistance Threats in the United States, 2019. https://doi.org/10.15620/cdc:82532
Chan, B. K., & Abedon, S. T. (2012). Phage therapy pharmacology: Phage cocktails. In Advances in Applied Microbiology (Vol. 78, pp. 1–23). Academic Press Inc. https://doi.org/10.1016/B978-0-12-394805-2.00001-4
ClinicalTrials.gov, N. (2020, November 16). Direct Lysis of Staph Aureus Resistant Pathogen Trial of Exebacase - Full Text View - ClinicalTrials.gov. https://clinicaltrials.gov/ct2/show/NCT04160468
ContraFect. (2020). Exebacase. https://www.contrafect.com/pipeline/exebacase
Cox, G., & Wright, G. D. (2013). Intrinsic antibiotic resistance: Mechanisms, origins, challenges and solutions. International Journal of Medical Microbiology, 303(6–7), 287–292. https://doi.org/10.1016/j.ijmm.2013.02.009
D’Costa, V. M., & Wright, G. D. (2017). Biochemical Logic of Antibiotic Inactivation and Modification. In Antimicrobial Drug Resistance (pp. 97–113). Springer International Publishing. https://doi.org/10.1007/978-3-319-46718-4_8
Datta, S., Rajnish, K. N., Samuel, M. S., Pugazlendhi, A., & Selvarajan, E. (2020). Metagenomic applications in microbial diversity, bioremediation, pollution monitoring, enzyme and drug discovery. A review. In Environmental Chemistry Letters (Vol. 18, Issue 4, pp. 1229–1241). Springer. https://doi.org/10.1007/s10311-020-01010-z
De Jonge, M. R., Koymans, L. H. M., Guillemont, J. E. G., Koul, A., & Andries, K. (2007). A computational model of the inhibition of Mycobacterium tuberculosis ATPase by a new drug candidate R207910. Proteins: Structure, Function and Genetics, 67(4), 971–980. https://doi.org/10.1002/prot.21376
De Maesschalck, V., Gutiérrez, D., Paeshuyse, J., Lavigne, R., & Briers, Y. (2020). Advanced engineering of third-generation lysins and formulation strategies for clinical applications. Critical Reviews in Microbiology, 1–17. https://doi.org/10.1080/1040841X.2020.1809346
DeCastro, M. E., Rodríguez-Belmonte, E., & González-Siso, M. I. (2016). Metagenomics of thermophiles with a focus on discovery of novel thermozymes. Frontiers in Microbiology, 7(SEP). https://doi.org/10.3389/fmicb.2016.01521
Defraine, V., Schuermans, J., Grymonprez, B., Govers, S. K., Aertsen, A., Fauvart, M., Michiels, J., Lavigne, R., & Briers, Y. (2016). Efficacy of Artilysin Art-175 against Resistant and Persistent Acinetobacter baumannii. 60(6), 3480–3488. https://doi.org/10.1128/AAC.00285-16
Drlica, K., & Zhao, X. (1997). DNA gyrase, topoisomerase IV, and the 4-quinolones. Microbiology and Molecular Biology Reviews, 61(3).
Edwards, R. A., & Rohwer, F. (2005). Viral metagenomics. Nature Reviews, 3(6), 504–510. https://doi.org/10.1038/nrmicro1163
Fenton, M., Ross, P., Mcauliffe, O., O’Mahony, J., & Coffey, A. (2010). Recombinant bacteriophage lysins as antibacterials. Bioengineered Bugs, 1(1), 9–16. https://doi.org/10.4161/bbug.1.1.9818
Ferrer, M., Beloqui, A., Vieites, J. M., Guazzaroni, M. E., Berger, I., & Aharoni, A. (2009). Interplay of metagenomics and in vitro compartmentalization. Microbial Biotechnology, 2(1), 31–39. https://doi.org/10.1111/j.1751-7915.2008.00057.x
Forsberg, K. J., Bhatt, I. V., Schmidtke, D. T., Javanmardi, K., Dillard, K. E., Stoddard, B. L., Finkelstein, I. J., Kaiser, B. K., & Malik, H. S. (2019). Functional metagenomics-guided discovery of potent cas9 inhibitors in the human microbiome. ELife, 8. https://doi.org/10.7554/eLife.46540
García, P., Madera, C., Martínez, B., Rodríguez, A., & Evaristo Suárez, J. (2009). Prevalence of bacteriophages infecting Staphylococcus aureus in dairy samples and their potential as biocontrol agents. Journal of Dairy Science, 92(7), 3019–3026. https://doi.org/10.3168/jds.2008-1744
Gerstmans, H. (2021). PhD dissertation: Development of the VersaTile technique and a microdroplet assay for the ultrahigh-throughput engineering of lysins as antibacterial agents.
Gerstmans, H., Criel, B., & Briers, Y. (2018). Synthetic biology of modular endolysins. Biotechnology Advances, 36(3), 624–640. https://doi.org/10.1016/j.biotechadv.2017.12.009
Gerstmans, H., Grimon, D., Gutiérrez, D., Lood, C., Rodríguez, A., van Noort, V., Lammertyn, J., Lavigne, R., & Briers, Y. (2020). A VersaTile-driven platform for rapid hit-to-lead development of engineered lysins. Science Advances, 6(23), 11. https://doi.org/10.1126/sciadv.aaz1136
Gerstmans, H., Rodríguez-Rubio, L., Lavigne, R., & Briers, Y. (2016). From endolysins to Artilysin®s: Novel enzyme-based approaches to kill drug-resistant bacteria. Biochemical Society Transactions, 44(1), 123–128. https://doi.org/10.1042/BST20150192
Goodridge, L. D. (2010). Designing Phage Therapeutics. In Current Pharmaceutical Biotechnology (1st ed., Vol. 11, Issue 1). Bentham Science Publishers. https://doi.org/10.2174/138920110790725348
Grishin, A. V., Shestak, N. V., Lavrova, N. V., Lyashchuk, A. M., Popova, L. I., Strukova, N. V., Generalova, M. S., Ryazanova, A. V., Polyakov, N. B., Galushkina, Z. M., Soboleva, L. A., Boksha, I. S., Karyagina, A. S., & Lunin, V. G. (2019). Fusion of lysostaphin to an albumin binding domain prolongs its half-life and bactericidal activity in the systemic circulation. Molecules, 24(16). https://doi.org/10.3390/molecules24162892
Gutiérrez, D., & Briers, Y. (2021). Lysins breaking down the walls of Gram-negative bacteria, no longer a no-go. Current Opinion in Biotechnology, 68, 15–22. https://doi.org/10.1016/j.copbio.2020.08.014
Gutiérrez, D., Delgado, S., Vázquez-Sánchez, D., Martínez, B., Cabo, M. L., Rodríguez, A., Herrera, J. J., & García, P. (2012). Incidence of Staphylococcus aureus and Analysis of Associated Bacterial Communities on Food Industry Surfaces. Appl Environ Microbiol. https://doi.org/10.1128/AEM.02045-12
Gutiérrez, D., Fernández, L., Rodríguez, A., & García, P. (2018). Are phage lytic proteins the secret weapon to kill staphylococcus aureus? MBio, 9(1), 1–17. https://doi.org/10.1128/mBio.01923-17
Gutiérrez, D., Garrido, V., Fernández, L., Portilla, S., Rodríguez, A., Grilló, M. J., & García, P. (2020). Phage Lytic Protein LysRODI Prevents Staphylococcal Mastitis in Mice. Frontiers in Microbiology, 11. https://doi.org/10.3389/fmicb.2020.00007
Gutiérrez, D., Vandenheuvel, D., Martínez, B., Rodríguez, A., Lavigne, R., & García, P. (2015). Two phages, phiIPLA-RODI and phiIPLA-C1C, lyse mono-and dual-species staphylococcal biofilms. Applied and Environmental Microbiology, 81(10), 3336–3348. https://doi.org/10.1128/AEM.03560-14
Hancock, R. E. W., & Brinkman, F. S. L. (2002). Function of Pseudomonas porins in uptake and efflux. Annual Review of Microbiology, 56, 17–38. https://doi.org/10.1146/annurev.micro.56.012302.160310
Hancock, R., & Fitz-James, P. C. (1964). Some differences in the action of penicillin, bacitracin, and vancomycin on Bacillus megaterium. Journal of Bacteriology, 87(5), 1044–1050. https://doi.org/10.1128/JB.87.5.1044-1050.1964
Harhala, M., Nelson, D. C., Miernikiewicz, P., Heselpoth, R. D., Brzezicka, B., Majewska, J., Linden, S. B., Shang, X., Szymczak, A., Lecion, D., Marek-Bukowiec, K., Kłak, M., Wojciechowicz, B., Lahutta, K., Konieczny, A., & Dąbrowska, K. (2018). Safety studies of pneumococcal endolysins Cpl-1 and Pal. Viruses, 10(11). https://doi.org/10.3390/v10110638
Ho, M. X., Hudson, B. P., Das, K., Arnold, E., & Ebright, R. H. (2009). Structures of RNA polymerase-antibiotic complexes. Current Opinion in Structural Biology, 19(6), 715–723. https://doi.org/10.1016/j.sbi.2009.10.010
Hoffman, S. B. (2001). Mechanisms of Antibiotic Resistance. Compendium on Continuing Education for the Practicing Veterinarian, 23(5), 464–472. https://doi.org/10.1128/microbiolspec.vmbf-0016-2015
Hugenholtz, P., & Tyson, G. W. (2008). Metagenomics. Nature, 455, 481–483. https://doi.org/10.1038//455481a
Invitrogen. (2013). One Shot TOP10 Competent Cells 280126 Rev A.0.
Jana, S., & Deb, J. K. (2006). Molecular understanding of aminoglycoside action and resistance. Applied Microbiology and Biotechnology, 70(2), 140–150. https://doi.org/10.1007/s00253-005-0279-0
Janus, M. (2020). Development of a combined in vitro transcription and translation system for rapid cell-free screening of metagenomic DNA. Staats-Und Universitätsbibliothek Hamburg Carl von Ossietzky.
Johnson, J., Jain, K., & Madamwar, D. (2017). Functional Metagenomics: Exploring Nature’s Gold Mine. In Current Developments in Biotechnology and Bioengineering: Functional Genomics and Metabolic Engineering (pp. 27–43). Elsevier Inc. https://doi.org/10.1016/B978-0-444-63667-6.00002-X
Keen, E. C. (2015). A century of phage research: Bacteriophages and the shaping of modern biology HHS Public Access. Bioessays, 37(1), 6–9. https://doi.org/10.1002/bies.201400152
Lam, K. N., Cheng, J., Engel, K., Neufeld, J. D., & Charles, T. C. (2015). Current and future resources for functional metagenomics. Frontiers in Microbiology, 6(OCT), 1196. https://doi.org/10.3389/fmicb.2015.01196
Lambert, P. A. (2005). Bacterial resistance to antibiotics: Modified target sites. Advanced Drug Delivery Reviews, 57(10), 1471–1485. https://doi.org/10.1016/j.addr.2005.04.003
Ledsgaard, L., Kilstrup, M., Karatt-Vellatt, A., McCafferty, J., & Laustsen, A. (2018). Basics of Antibody Phage Display Technology. Toxins, 10(6), 236. https://doi.org/10.3390/toxins10060236
Lewis, K. (2013). Platforms for antibiotic discovery. Nature Reviews Drug Discovery, 12(5), 371–387. https://doi.org/10.1038/nrd3975
Magiorakos, A. P., Srinivasan, A., Carey, R. B., Carmeli, Y., Falagas, M. E., Giske, C. G., Harbarth, S., Hindler, J. F., Kahlmeter, G., Olsson-Liljequist, B., Paterson, D. L., Rice, L. B., Stelling, J., Struelens, M. J., Vatopoulos, A., Weber, J. T., & Monnet, D. L. (2012). Multidrug-resistant, extensively drug-resistant and pandrug-resistant bacteria: An international expert proposal for interim standard definitions for acquired resistance. Clinical Microbiology and Infection, 18(3), 268–281. https://doi.org/10.1111/j.1469-0691.2011.03570.x
Mechanisms of Antibacterial Drugs. (n.d.). Lumen: Microbiology. Retrieved November 4, 2020, from https://courses.lumenlearning.com/microbiology/chapter/mechanisms-of-an…
Moubareck, C. A. (2020). Polymyxins and bacterial membranes: A review of antibacterial activity and mechanisms of resistance. Membranes, 10(8), 1–30. https://doi.org/10.3390/membranes10080181
Mulani, M. S., Kamble, E. E., Kumkar, S. N., Tawre, M. S., & Pardesi, K. R. (2019). Emerging strategies to combat ESKAPE pathogens in the era of antimicrobial resistance: A review. Frontiers in Microbiology, 10(APR), 539. https://doi.org/10.3389/fmicb.2019.00539
Nanninga, N. (2014). Cell Structure, Organization, Bacteria and Archaea. In Reference Module in Biomedical Sciences (pp. 1–21). Elsevier. https://doi.org/10.1016/b978-0-12-801238-3.02309-6
NCBI. (n.d.). Staphylococcus phage phiIPLA-RODI, complete genome. Retrieved May 3, 2021, from https://www.ncbi.nlm.nih.gov/nuccore/KP027446
NEB. (n.d.). What type of ends does dsDNA Fragmentase create? Retrieved May 11, 2021, from https://international.neb.com/faqs/2019/05/28/what-type-of-ends-does-ds…
O’Flaherty, S., Ross, R. P., & Coffey, A. (2009). Bacteriophage and their lysins for elimination of infectious bacteria: Review article. FEMS Microbiology Reviews, 33(4), 801–819. https://doi.org/10.1111/j.1574-6976.2009.00176.x
O’Neill, J. (2014). Antimicrobial Resistance : Tackling a crisis for the health and wealth of nations. In Review on Antimicrobial Resistance (Issue December).
Packer, M. S., & Liu, D. R. (2015). Methods for the directed evolution of proteins. Nature Reviews Genetics, 16(7), 379–394. https://doi.org/10.1038/nrg3927
Pagès, J. M., James, C. E., & Winterhalter, M. (2008). The porin and the permeating antibiotic: A selective diffusion barrier in Gram-negative bacteria. In Nature Reviews Microbiology (Vol. 6, Issue 12, pp. 893–903). Nature Publishing Group. https://doi.org/10.1038/nrmicro1994
Promega Connections. (n.d.). Three Factors That Can Hurt Your Assay Results. Retrieved May 12, 2021, from https://www.promegaconnections.com/dont-let-these-three-common-issues-h…
Que, Y. A., & Moreillon, P. (2015). Staphylococcus aureus (Including Staphylococcal Toxic Shock Syndrome). In Mandell, Douglas, and Bennett’s Principles and Practice of Infectious Diseases (Vol. 2, pp. 2237-2271.e5). Elsevier Inc. https://doi.org/10.1016/B978-1-4557-4801-3.00196-X
Queenan, A. M., & Bush, K. (2007). Carbapenemases: The versatile β-lactamases. Clinical Microbiology Reviews, 20(3), 440–458. https://doi.org/10.1128/CMR.00001-07
Raghunath, D. (2010). New metallo β-lactamase NDM-1. Indian Journal of Medical Research, 132(11), 478–481. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3028958/
Rodríguez-Rubio, L., Chang, W. L., Gutiérrez, D., Lavigne, R., Martínez, B., Rodríguez, A., Govers, S. K., Aertsen, A., Hirl, C., Biebl, M., Briers, Y., & García, P. (2016). “Artilysation” of endolysin λSa2lys strongly improves its enzymatic and antibacterial activity against streptococci. Scientific Reports, 6(1), 1–11. https://doi.org/10.1038/srep35382
Sage Science. (n.d.). Our Technology. Retrieved May 20, 2021, from https://sagescience.com/our-technology/
Santajit, S., & Indrawattana, N. (2016). Mechanisms of Antimicrobial Resistance in ESKAPE Pathogens. In BioMed Research International (Vol. 2016). Hindawi Limited. https://doi.org/10.1155/2016/2475067
Schmelcher, M., Donovan, D. M., & Loessner, M. J. (2012). Bacteriophage endolysins as novel antimicrobials. Future Microbiology, 7(10), 1147–1171. https://doi.org/10.2217/fmb.12.97
Schmitz, F. J., & Fluit, A. C. (2010). Mechanisms of antibacterial resistance. In Infectious Diseases: Third Edition (Vol. 2, pp. 1308–1322). Elsevier Inc. https://doi.org/10.1016/B978-0-323-04579-7.00131-3
Schroeder, E., de Souza, O., Santos, D., Blanchard, J., & Basso, L. (2005). Drugs that Inhibit Mycolic Acid Biosynthesis in Mycobacterium tuberculosis. Current Pharmaceutical Biotechnology, 3(3), 197–225. https://doi.org/10.2174/1389201023378328
Sharma, S., Chatterjee, S., Datta, S., Prasad, R., Dubey, D., Prasad, R. K., & Vairale, M. G. (2017). Bacteriophages and its applications: an overview. Folia Microbiologica, 62, 17–55. https://doi.org/10.1007/s12223-016-0471-x
Smillie, C., Garcillán-Barcia, M. P., Francia, M. V., Rocha, E. P. C., & de la Cruz, F. (2010). Mobility of Plasmids. Microbiology and Molecular Biology Reviews, 74(3), 434–452. https://doi.org/10.1128/mmbr.00020-10
Steward, G. F., & Culley, A. I. (2010). Extraction and purification of nucleic acids from viruses. In Manual of Aquatic Viral Ecology (pp. 154–165). American Society of Limnology and Oceanography. https://doi.org/10.4319/mave.2010.978-0-9845591-0-7.154
Straus, S. K., & Hancock, R. E. W. (2006). Mode of action of the new antibiotic for Gram-positive pathogens daptomycin: Comparison with cationic antimicrobial peptides and lipopeptides. Biochimica et Biophysica (BBA) Acta - Biomembranes, 1758(9), 1215–1223. https://doi.org/10.1016/j.bbamem.2006.02.009
Sulakvelidze, A., Alavidze, Z., & Morris, J. (2001). Bacteriophage therapy. Antimicrobial Agents and Chemotherapy, 45(3), 649–659. https://doi.org/10.1128/AAC.45.3.649-659.2001
Thermo Fisher Scientific. (n.d.). The Technology Behind TOPO Cloning. Retrieved May 13, 2021, from https://www.thermofisher.com/be/en/home/life-science/cloning/topo/topo-…
Uchiyama, T., & Miyazaki, K. (2009). Functional metagenomics for enzyme discovery: challenges to efficient screening. Current Opinion in Biotechnology, 20(6), 616–622. https://doi.org/10.1016/j.copbio.2009.09.010
UGent magazine. (2020, September). UGent magazine. UGent Magazine: Blad Voor Alumni En Andere Durfdenkers, 1, 40.
Valiquette, L., & Laupland, K. B. (2015). Digging for new solutions. Canadian Journal of Infectious Diseases and Medical Microbiology, 26(6), 289–290. http://www.hindawi.com
Van Bambeke, F., Balzi, E., & Tulkens, P. M. (2000). Antibiotic efflux pumps. Biochemical Pharmacology, 60(4), 457–470. https://doi.org/10.1016/S0006-2952(00)00291-4
Vander Elst, N., & Meyer, E. (2018). Potential therapeutic application of bacteriophages and phage-derived endolysins as alternative treatment of bovine mastitis. Vlaams Diergeneeskundig Tijdschrift, 87(4), 181–187. https://doi.org/10.21825/vdt.v87i4.16065
Vollmer, W., & Born, P. (2010). Bacterial Cell Envelope Peptidoglycan. In Microbial Glycobiology (pp. 15–28). Elsevier Inc. https://doi.org/10.1016/B978-0-12-374546-0.00002-X
Walmagh, M., Briers, Y., Santos, S. dos B., Azeredo, J., & Lavigne, R. (2012). Characterization of Modular Bacteriophage Endolysins from Myoviridae Phages OBP, 201φ2-1 and PVP-SE1. PLoS ONE, 7(5). https://doi.org/10.1371/journal.pone.0036991
Whitman, W. B., Coleman, D. C., & Wiebe, W. J. (1998). Prokaryotes: The unseen majority. Proceedings of the National Academy of Sciences of the United States of America, 95(12), 6578–6583. https://doi.org/10.1073/pnas.95.12.6578
Wikipedia Contributors. (2021). Poisson distribution. Wikipedia, The Free Encyclopedia.
Wirmer, J., & Westhof, E. (2006). Molecular Contacts Between Antibiotics and the 30S Ribosomal Particle. Methods in Enzymology, 415, 180–202. https://doi.org/10.1016/S0076-6879(06)15012-0
Young, R. (2014). Phage lysis: Three steps, three choices, one outcome. Journal of Microbiology, 52(3), 243–258. https://doi.org/10.1007/s12275-014-4087-z
Young, R., Wang, I.-N., & Roof, W. D. (2000). Phages will out: Strategies of host cell lysis. Trends in Microbiology, 8(3), 120–128. https://doi.org/10.1016/S0966-842X(00)01705-4









