Van CT naar 3D: Hoe AI de zorg kan versnellen
Artificiële intelligentie of AI heeft de wereld in zijn greep. Deze snelle opkomst zorgt in alle sectoren voor grote opwinding, maar ook voor extra bezorgdheid over mogelijke gevolgen. De medische sector vormt hier geen uitzondering op. Wat kan AI nu echt betekenen voor onze gezondheidszorg? Zullen artsen binnenkort hun plaats moeten afgeven aan hyperintelligente robots? Hoewel we dit nog niet hebben bereikt en misschien ook nooit zullen bereiken, biedt AI een breed scala aan mogelijkheden om de gezondheidszorg op een slimme manier te verbeteren. Deze scriptie stelt het gebruik van AI voor om snelle en accurate 3D-modellen te bekomen, die orthopedische zorg kunnen helpen versnellen.
3D-modellen via AI
AI kan een grote rol spelen in het verwerken van medische beelden op een snelle en effectieve manier. Hoe gaat zoiets in zijn werk? Stel je voor dat je een geavanceerd model een CT-scan geeft van bijna het volledige lichaam, zonder enige aanvullende informatie. Binnen enkele seconden kan dit model gedetailleerde informatie geven over de anatomie van de botten in de onderste ledematen van de patiënt. Op deze manier krijg je niet alleen vlotte toegang tot de scan zelf, maar ook tot 3D-modellen van de interne structuren van de patiënt.
Dit vormt een belangrijke eerste stap in de richting van verbeterde orthopedische zorg, met snellere diagnosemogelijkheden en diepgaander onderzoek. Wanneer bijvoorbeeld een wielrenner zoals Evenepoel een breuk in de heup oploopt, beschikt de chirurg die een mogelijkse operatie voorbereidt over aanzienlijk waardevollere informatie wanneer een 3D-model van het bot met de breuk beschikbaar zou zijn. Hierdoor kan de chirurg grondiger beslissen of een ingreep nodig is en zich beter voorbereiden.
Veel arbeid: zowel voor een specialist als een computer
Om de specifieke anatomie van de botten bij elke individuele patiënt te verkrijgen, dienen de medische beelden gelabeld te worden, vergelijkbaar met het markeren van de botten op een grote afbeelding met behulp van een fluostift. Een kernprobleem voor orthopedische en biomechanische onderzoekers is dat dit een arbeidsintensief proces is. Het verwerken van één CT-scan door een expert vergt al vlug enkele uren. Ondanks voortdurend onderzoek naar geautomatiseerde methoden om dit proces te versnellen, bestaat er momenteel nog geen universele standaard die zowel snelle als nauwkeurige resultaten levert.
Om snelle en nauwkeurige resultaten te verkrijgen wordt steeds vaker gekeken naar AI vanwege de potentie om beide eigenschappen te combineren. Het ontwikkelen van zo'n model brengt echter nieuwe uitdagingen met zich mee. AI-modellen leren patronen herkennen door een groot aantal voorbeelden te gebruiken en hun voorspellingen geleidelijk te verbeteren door feedback. Wanneer het leerproces is voltooid, kan het model voorspellingen doen voor ongeziene gevallen. Dit proces valt te vergelijken met hoe ons menselijk brein werkt, met talloze aanpasbare connecties. Door deze connecties op een slimme wijze te wijzigen, kan de herkenning van patronen voortdurend verbeterd worden.
Hier vloeit de eerste moeilijkheid uit. Door de hoge resolutie en grootte van de CT-scans, vergt het heel veel rekenwerk en geheugen van de computer om deze connecties telkens opnieuw te verfijnen op basis van de volledige afbeelding. Wanneer de afbeelding te groot wordt, is het zelfs onmogelijk om deze te verwerken in zijn geheel. Hierdoor wordt een grote CT-scan vaak in stukken geknipt die apart worden verwerkt. Hierdoor is er niet alleen een verlies aan context, maar duurt het ook lang voor bestaande technieken om een voorspelling te voltooien. Om deze moeilijkheden te verhelpen wordt een manier voorgesteld zodat de scans grotendeels in zijn geheel worden verwerkt. Eerst worden de interessante gebieden uit de CT-scan gehaald, met behoud van de nodige context, om hierop een fijnere voorspelling te doen. Op deze manier is het mogelijk om de anatomie van de botten in de onderste ledematen in een paar seconden te voorspellen met hoge precisie. Een paar seconden versus een aantal uur, het gebruik van AI kan de experts dus heel wat werk besparen!
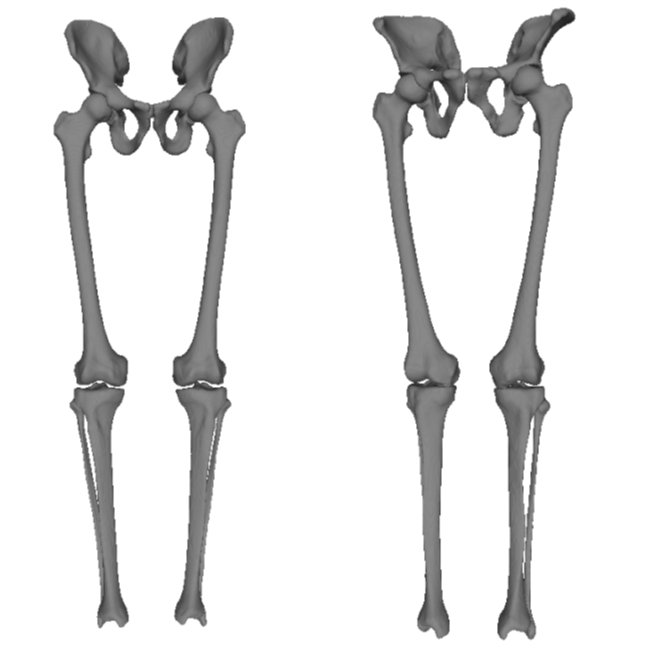
Voorspellingen gemaakt door het AI model.
Onbestaande data?
Een bijkomende uitdaging bij de ontwikkeling van een model is de beschikbaarheid van voldoende leerzame voorbeelden. Het leerproces draait om het presenteren van correcte voorbeelden aan de computer, en het is duidelijk dat hoe meer voorbeelden beschikbaar zijn, des te beter het model kan leren. Dit is vaak een grote uitdaging in de gezondheidszorg. Het verkrijgen van één correct voorbeeld, verkregen via het werk van een specialist, kan uren in beslag nemen, waardoor het moeilijk is om een uitgebreide dataset met voorbeelden samen te stellen.
Om dit probleem aan te pakken is niet alleen gekeken naar het bouwen van een model, maar wordt ook een manier voorgesteld om duizenden nieuwe voorbeelden te genereren uit een beperktere dataset. Zo’n voorbeeld bestaat uit een CT-scan, niet genomen met behulp van de scanner, maar artificieel ontwikkeld. Deze is dan vergezeld met de aanduiding van de interessante anatomische structuren op de juiste locaties. Deze benadering maakt het mogelijk om voorbeelden te creëren die vergelijkbaar kunnen zijn met die van echte patiënten. Dit opent de deur naar de ontwikkeling van toekomstige modellen die met meer precisie voorspellingen kunnen doen, zelfs wanneer ze beschikken over een beperkte dataset.
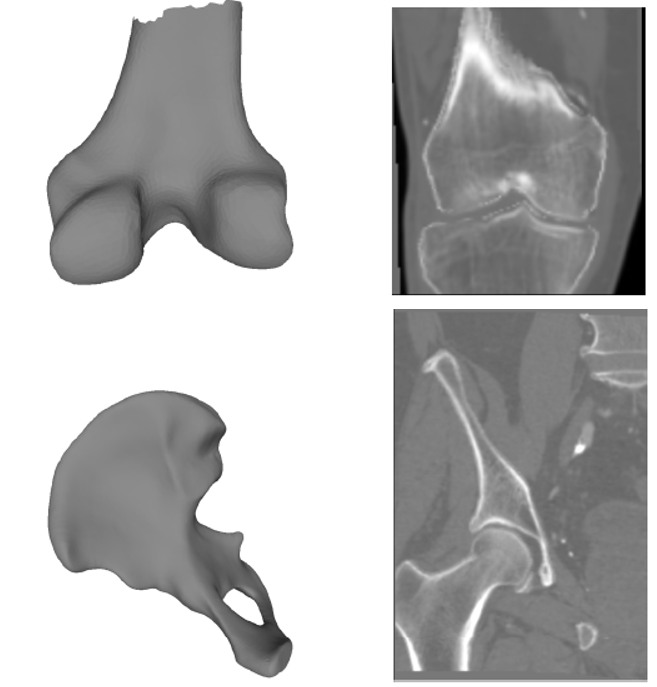
Gegenereerde 3D-modellen en CT scans.
Artsen en AI: hand in hand
Een toekomst waarin AI de rol van artsen volledig overneemt, lijkt nog ver weg. Desalniettemin wordt het steeds belangrijker voor artsen om de opkomende AI-ontwikkelingen te omarmen. Het gebruik van de voorgestelde methode, waarbij een uitgebreide CT-scan in enkele seconden kan worden verwerkt in plaats van uren handmatig werk, heeft het potentieel om specialisten die momenteel veel tijd besteden aan het annoteren van CT-scans, te bevrijden voor andere cruciale taken. De voorgestelde methode om extra gegevens te genereren biedt hiernaast een effectieve manier om sneller hoogwaardige datasets te verkrijgen, waardoor de nood voor manueel werk vermindert en er meer mogelijkheden ontstaan voor het efficiënt trainen van AI-modellen. Op deze manier komt er door de samenwerking van AI en het personeel in de gezondheidszorg meer ruimte vrij om extra levens te redden!
Bibliografie
[1] E. A. Audenaert, J. Van Houcke, D. F. Almeida, L. Paelinck, M. Peiffer, G. Steenackers,
and D. Vandermeulen, “Cascaded statistical shape model based segmentation of the
full lower limb in CT,” Computer Methods in Biomechanics and Biomedical Engineering,
vol. 22, no. 6, pp. 644–657, 2019.
[2] C. R. Henak, A. E. Anderson, and J. A. Weiss, “Subject-specific analysis of joint contact
mechanics: Application to the study of osteoarthritis and surgical planning,” Journal of
Biomechanical Engineering, vol. 135, no. 2, pp. 1–26, 2013.
[3] M. Hans-Peter and T. Heimann, “Statistical shape models for 3d medical image segmentation:
{A} review,” Medical Image Analysis, vol. 13, pp. 543–563, 2009.
[4] C. Chu, C. Chen, L. Liu, and G. Zheng, “FACTS: Fully Automatic CT Segmentation of a
Hip Joint,” Annals of Biomedical Engineering, vol. 43, no. 5, pp. 1247–1259, 2015.
[5] G. Litjens, T. Kooi, B. E. Bejnordi, A. A. A. Setio, F. Ciompi, M. Ghafoorian, J. A.
van der Laak, B. van Ginneken, and C. I. S´anchez, “A survey on deep learning in medical
image analysis,” Medical Image Analysis, vol. 42, no. December 2012, pp. 60–88, 2017.
[6] C. D. Naylor, “On the prospects for a (Deep) learning health care system,” JAMA -
Journal of the American Medical Association, vol. 320, no. 11, pp. 1099–1100, 2018.
[7] O. Ronneberger, P. Fischer, and T. Brox, “U-net: Convolutional networks for biomedical
image segmentation,” Lecture Notes in Computer Science (including subseries Lecture
Notes in Artificial Intelligence and Lecture Notes in Bioinformatics), vol. 9351, pp. 234–
241, 2015.
[8] F. Ambellan, A. Tack, M. Ehlke, and S. Zachow, “Automated segmentation of knee bone
and cartilage combining statistical shape knowledge and convolutional neural networks:
Data from the osteoarthritis initiative,” Medical Image Analysis, vol. 52, pp. 109–118,
2019.
[9] P. Liu, H. Han, Y. Du, H. Zhu, Y. Li, F. Gu, H. Xiao, J. Li, C. Zhao, L. Xiao, X. Wu, and
S. K. Zhou, “Deep learning to segment pelvic bones: large-scale CT datasets and baseline
models,” International Journal of Computer Assisted Radiology and Surgery, vol. 16, no. 5,
pp. 749–756, 2021.[10] Y. Deng, L. Wang, C. Zhao, S. Tang, X. Cheng, H. W. Deng, and W. Zhou, “A deep
learning-based approach to automatic proximal femur segmentation in quantitative CT
images,” Medical and Biological Engineering and Computing, vol. 60, no. 5, pp. 1417–
1429, 2022.
[11] A. Klein, J. Warszawski, J. Hillengaß, and K. H. Maier-Hein, “Automatic bone segmentation
in whole-body CT images,” International Journal of Computer Assisted Radiology
and Surgery, vol. 14, no. 1, pp. 21–29, 2019.
[12] R. J. Kuiper, R. J. Sakkers, M. van Stralen, V. Arbabi, M. A. Viergever, H. Weinans, and
P. R. Seevinck, “Efficient cascaded v-net optimization for lower extremity ct segmentation
validated using bone morphology assessment,” Journal of Orthopaedic Research, vol. 40,
pp. 2894–2907, 12 Dec. 2022.
[13] E. Marieb and K. Hoehn, Human Anatomy & Physiology, 11th ed. Pearson Eduction, 2019,
ch. The Skeleton, pp. 231–282.
[14] J. M. DeSilva and K. R. Rosenberg, “Anatomy, development, and function of the human
pelvis,” The Anatomical Record, vol. 300, no. 4, pp. 628–632, 2017.
[15] F. Netter, Netter Atlas of Human Anatomy: Classic Regional Approach, 8th ed. Elsevier -
Health Sciences Division, 2022.
[16] T. Fukuda, T. Yonenaga, T. Miyasaka, T. Kimura, M. Jinzaki, and H. Ojiri, “CT in
osteoarthritis: its clinical role and recent advances,” Skeletal Radiology, no. 0123456789,
2022.
[17] A. Virz`ı, C. O. Muller, J. B. Marret, E. Mille, L. Berteloot, D. Gr´event, N. Boddaert, P.
Gori, S. Sarnacki, and I. Bloch, “Comprehensive review of 3d segmentation software tools
for mri usable for pelvic surgery planning,” Journal of Digital Imaging, vol. 33, pp. 99–110,
1 Feb. 2020.
[18] N. J. Mardis, “Emerging technology and applications of 3D printing in the medical field,”
en, Missouri medicine, vol. 115, no. 4, pp. 368–373, Jul. 2018.
[19] L. Pugliese, S. Marconi, E. Negrello, V. Mauri, A. Peri, V. Gallo, F. Auricchio, and A.
Pietrabissa, “The clinical use of 3D printing in surgery,” Updates in Surgery, vol. 70, no. 3,
pp. 381–388, 2018.
[20] P. De Backer, S. Vermijs, C. Van Praet, P. De Visschere, S. Vandenbulcke, A. Mottaran,
C. A. Bravi, C. Berquin, E. Lambert, S. Dautricourt, W. Goedertier, A. Mottrie, C.
Debbaut, and K. Decaestecker, “A Novel Three-dimensional Planning Tool for Selective
Clamping During Partial Nephrectomy: Validation of a Perfusion Zone Algorithm,” European
Urology, vol. 83, pp. 413–421, 2023.
[21] H. Lamecker, “Variational and Statistical Shape Modeling for 3D Geometry Reconstruction,”
Naturwissenschaften, p. 123, 2008.[22] F. Bernardini, J. Mittleman, H. Rushmeier, C. Silva, and G. Taubin, “The ball-pivoting
algorithm for surface reconstruction,” IEEE Transactions on Visualization and Computer
Graphics, vol. 5, no. 4, pp. 349–359, 1999.
[23] M. Gao, N. Ruan, J. Shi, and W. Zhou, “Deep Neural Network for 3D Shape Classification
Based on Mesh Feature,” Sensors, vol. 22, no. 18, pp. 1–11, 2022.
[24] O. van Kaick, H. Zhang, G. Hamarneh, and D. Cohen-Or, “A survey on shape correspondence,”
Eurographics Symposium on Geometry Processing, vol. 30, no. 6, pp. 1681–1707,
2011.
[25] Y. Sahillio˘glu, “Recent advances in shape correspondence,” Visual Computer, vol. 36, no. 8,
pp. 1705–1721, 2020.
[26] T. Heimann and H. P. Meinzer, “Statistical shape models for 3D medical image segmentation:
A review,” Medical Image Analysis, vol. 13, no. 4, pp. 543–563, 2009.
[27] I. Goodfellow, Y. Bengio, and A. Courville, Deep Learning. MIT Press, 2016, http://
www.deeplearningbook.org.
[28] T. Panch, P. Szolovits, and R. Atun, “Artificial intelligence, machine learning and health
systems,” Journal of Global Health, vol. 8, no. 2, pp. 1–8, 2018.
[29] P. Ongsulee, “Artificial intelligence, machine learning and deep learning,” International
Conference on ICT and Knowledge Engineering, pp. 1–6, 2018.
[30] H. D. Block, “The perceptron: A model for brain functioning,” Reviews of Modern Physics,
vol. 34, pp. 123–135, 1962.
[31] D. Plaut, S. Nowlan, and G. Hinton, “Experiments on learning by back propagation,”
Technical Report CMU-CS-86-126, no. June, 1986.
[32] S. Ruder, “An overview of gradient descent optimization algorithms,” CoRR, vol. abs/1609.04747,
2016.
[33] J. L. Ba and D. P. Kingma, “Adam: A method for stochastic optimization,” 3rd International
Conference on Learning Representations, ICLR 2015 - Conference Track Proceedings,
pp. 1–15, 2015.
[34] R. M. Schmidt, F. Schneider, and P. Hennig, “Descending through a crowded valley -
benchmarking deep learning optimizers,” in Proceedings of the 38th International Conference
on Machine Learning, M. Meila and T. Zhang, Eds., ser. Proceedings of Machine
Learning Research, vol. 139, PMLR, 18–24 Jul 2021, pp. 9367–9376.
[35] K. O’Shea and R. Nash, “An introduction to convolutional neural networks,” CoRR,
vol. abs/1511.08458, 2015.
[36] M. A. Islam, M. Kowal, S. Jia, K. G. Derpanis, and N. D. B. Bruce, “Position, padding and
predictions: A deeper look at position information in cnns,” CoRR, vol. abs/2101.12322,
2021.[37] H. Gholamalinezhad and H. Khosravi, “Pooling methods in deep neural networks, a review,”
CoRR, vol. abs/2009.07485, 2020.
[38] D. L. Pham´e, C. Xu´e, and J. L. Prince´e, “A survey of current methods in medical image
segmentation,” 1998.
[39] L. Lenchik, L. Heacock, A. A. Weaver, R. D. Boutin, T. S. Cook, J. Itri, C. G. Filippi,
R. P. Gullapalli, J. Lee, M. Zagurovskaya, T. Retson, K. Godwin, J. Nicholson, and P. A.
Narayana, “Automated segmentation of tissues using ct and mri: A systematic review,”
Academic Radiology, vol. 26, pp. 1695–1706, 12 Dec. 2019.
[40] D. Marzorati, M. Sarti, L. Mainardi, A. Manzotti, and P. Cerveri, “Deep 3D convolutional
networks to segment bones affected by severe osteoarthritis in CT scans for PSI-based
knee surgical planning,” IEEE Access, vol. 8, pp. 196 394–196 407, 2020.
[41] M. Krˇcah, G. Sz´ekely, and R. Blanc, “Fully automatic and fast segmentation of the femur
bone from 3D-CT images with no shape prior,” Proceedings - International Symposium on
Biomedical Imaging, pp. 2087–2090, 2011.
[42] D. L. Pham, C. Xu, and J. L. Prince, “Current methods in medical image segmentation,”
Annual Review of Biomedical Engineering, vol. 2, no. 2000, pp. 315–337, 2000.
[43] N. Otsu, P. L. Smith, D. B. Reid, C. Environment, L. Palo, P. Alto, and P. L. Smith, “A
Threshold Selection Method from Gray-Level Histograms,” IEEE Transactions on Systems,
Man, and Cybernetics, vol. C, no. 1, pp. 62–66, 1979.
[44] J. Zhang, C. H. Yan, C. K. Chui, and S. H. Ong, “Fast segmentation of bone in CT
images using 3D adaptive thresholding,” Computers in Biology and Medicine, vol. 40,
no. 2, pp. 231–236, 2010.
[45] C. Goodall, “Procrustes Methods in the Statistical Analysis of Shape,” Journal of the
Royal Statistical Society, vol. 53, no. 2, pp. 285–339, 1991.
[46] N. Siddique, S. Paheding, C. P. Elkin, and V. Devabhaktuni, “U-net and its variants for
medical image segmentation: A review of theory and applications,” IEEE Access, 2021.
[47] J. Cates, S. Elhabian, and R. Whitaker, “Shapeworks: Particle-based shape correspondence
and visualization software,” in Statistical Shape and Deformation Analysis, Elsevier, 2017,
pp. 257–298.
[48] R. Bhalodia, S. Elhabian, J. Adams, W. Tao, L. Kavan, and R. Whitaker, “DeepSSM: A
Blueprint for Image-to-Shape Deep Learning Models,” 2021.
[49] A. Adam, Mesh voxelisation, https://nl.mathworks.com/matlabcentral/fileexchange/
27390-mesh-voxelisation, 2023.[50] A. Paszke, S. Gross, F. Massa, A. Lerer, J. Bradbury, G. Chanan, T. Killeen, Z. Lin, N.
Gimelshein, L. Antiga, A. Desmaison, A. Kopf, E. Yang, Z. DeVito, M. Raison, A. Tejani, S.
Chilamkurthy, B. Steiner, L. Fang, J. Bai, and S. Chintala, “Pytorch: An imperative style,
high-performance deep learning library,” in Advances in Neural Information Processing
Systems 32, Curran Associates, Inc., 2019, pp. 8024–8035.
[51] BRATS challenge, https://www.med.upenn.edu/sbia/brats2018.html, 2018.
[52] ISLES challenge, http://www.isles-challenge.org/ISLES2018/, 2019.
[53] A. P. Zijdenbos, B. M. Dawant, R. A. Margolin, and A. C. Palmer, “Morphometric Analysis
of White Matter Lesions in MR Images: Method and Validation,” IEEE Transactions on
Medical Imaging, vol. 13, no. 4, pp. 716–724, 1994.
[54] D. P. Huttenlocher, W. J. Rucklidge, and G. A. Klanderman, “Comparing images using
the Hausdorff distance under translation,” Proceedings of the IEEE Computer Society
Conference on Computer Vision and Pattern Recognition, vol. 1992-June, no. 9, pp. 654–
656, 1992.
[55] W. E. Lorensen and H. E. Cline, “Marching Cubes: a High Resolution 3D Surface Construction
Algorithm.,” Computer Graphics (ACM), vol. 21, no. 4, pp. 163–169, 1987.
[56] S. Van derWalt, J. L. Sch¨onberger, J. Nunez-Iglesias, F. Boulogne, J. D.Warner, N. Yager,
E. Gouillart, and T. Yu, “Scikit-image: Image processing in python,” PeerJ, vol. 2, e453,
2014.
[57] P. Cignoni, M. Callieri, M. Corsini, M. Dellepiane, F. Ganovelli, and G. Ranzuglia, “Mesh-
Lab: an Open-Source Mesh Processing Tool,” in Eurographics Italian Chapter Conference,
V. Scarano, R. D. Chiara, and U. Erra, Eds., The Eurographics Association, 2008.
[58] G. Taubin, “Signal processing approach to fair surface design,” Proceedings of the ACM
SIGGRAPH Conference on Computer Graphics, pp. 351–358, 1995.
[59] Z. Chen and H. Zhang, “Learning implicit fields for generative shape modeling,” Proceedings
of the IEEE Computer Society Conference on Computer Vision and Pattern Recognition,
vol. 2019-June, pp. 5932–5941, 2019.
[60] F. Chen, Y. Xie, P. Xu, Z. Zhao, D. Zhang, and H. Liao, “Efficient lower-limb segmentation
for large-scale volumetric ct by using projection view and voxel group attention,” Medical
and Biological Engineering and Computing, vol. 60, pp. 2201–2216, 8 Aug. 2022.
[61] D. L¨udke, T. Amiranashvili, F. Ambellan, I. Ezhov, B. H. Menze, and S. Zachow, “Landmarkfree
statistical shape modeling via neural flow deformations,” in Medical Image Computing
and Computer Assisted Intervention – MICCAI 2022, Springer Nature Switzerland, 2022,
pp. 453–463.
[62] F. L. Bookstein, “Principal Warps: Thin-Plate Splines and the Decomposition of Deformations,”
IEEE Transactions on Pattern Analysis and Machine Intelligence, vol. 11, no. 6,
pp. 567–585, 1989.[63] H. Huy Khahn, Thin Plate Splines Warping, https://khanhha.github.io/posts/Thin-
Plate-Splines-Warping, 2019.
[64] D. C. L¨udke, “Neural flow-based deformations for statistical shape modelling,” 2022, Thesis
FLOW SSM.
[65] K. Gupta and M. Chandraker, “Neural mesh flow: 3D manifold mesh generation via diffeomorphic
flows,” Advances in Neural Information Processing Systems, vol. 2020-December,
no. NeurIPS, pp. 1–12, 2020.
[66] R. Davies, “Learning shape: optimal models for analysing natural variability,” Learning,
2002.
[67] L. V. D. Maaten and G. Hinton, “Visualizing Data using t-SNE,” vol. 9, pp. 2579–2605,
2008.
[68] A. Brando, Mixture density networks (mdn) for distribution and uncertainty estimation,
Report of the Master’s Thesis: Mixture Density Networks for distribution and uncertainty
estimation., 2017.
[69] V. Jorgetti, L. M. Dos Reis, and S. M. Ott, “Ethnic differences in bone and mineral
metabolism in healthy people and patients with CKD,” Kidney International, vol. 85,
no. 6, pp. 1283–1289, 2014.









