Hoe een simpele knip in het DNA het de geheimen van begin van het leven kan onthullen
- GwennyCosemans
Hoe een simpele knip in het DNA het de geheimen van begin van het leven kan onthullen
Wereldwijd is het voor 15% van alle koppels moeilijk tot onmogelijk om op een natuurlijke wijze een genetisch eigen kind te krijgen. Wat een mogelijke oplossing kan bieden is het toepassen van technieken zoals in vitro fertilisatie (IVF) of inter-cytoplasmatische sperma injectie (ICSI). Alhoewel er grote vooruitgang gemaakt is in dit veld, blijft het probleem bestaan dat na het terugplaatsen van de embryo’s, er geen innesteling in de baarmoeder plaatsvindt, en er dus geen zwangerschap optreedt. In deze masterscriptie probeerden we meer te weten te komen over de genen die betrokken zijn bij de implantatie van het vroege embryo, omdat begrip van het probleem essentieel is voor het vinden van oplossingen. Dit doen wij door (figuurlijk) de schaar te zetten in bepaalde genen, waarvan we vermoeden dat ze betrokken zijn bij deze processen. Als we zien wat er misgaat wanneer deze genen uitgeschakeld zijn, weten we natuurlijk wat er wel had moeten gebeuren wanneer deze genen naar behoren zouden functioneren. Maar kan je zomaar een knip in het DNA geven? Volg even mee om te zien hoe wij dit gerealiseerd hebben met een gen bewerkingssysteem die ‘CRISPR/Cas9’ genoemd wordt.
Het begin des levens: de vroege embryonale ontwikkeling (Figuur 1)
Het leven begint wanneer een eicel bevrucht wordt door een spermacel, en er een zygote gevormd wordt. Daarna zullen er verschillende celdelingen plaatsvinden, en zal het embryo groeien tot het op dag 3.5 een klomp van cellen zal vormen, die we een morula noemen. Vervolgens ontwikkelt de morula verder tot een blastocyst, die naast een met vloeistof gevulde holte ook bestaat uit 2 celtypes: het trophectoderm en de inner cell mass. Het trophectoderm is verantwoordelijk voor de vorming van de placenta en de innesteling in de baarmoeder, terwijl de inner cell mass de foetus (en dus de baby) zal vormen. Aangezien het trophectoderm betrokken is bij de innesteling en implantatie van het embryo, is dit het celtype waarin we precies geïnteresseerd zijn. De correcte vorming van het trophectoderm is echter essentieel om een correcte implantatie van het embryo (en dus een correcte zwangerschap) te verkrijgen. Kortom zullen wij de processen bestuderen die cruciaal zijn voor de vorming van de trophectoderm.
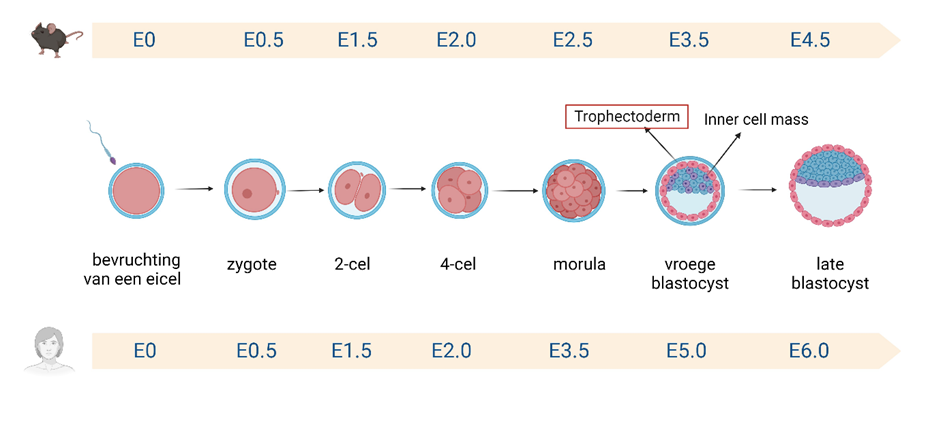
Figuur 1: De vroege embryonale ontwikkeling van muis en mens embryo’s.
De muis als modelsysteem voor de mens
In België is het door de zogenaamde ‘embryowet’ toegelaten om menselijke embryo’s te creëren voor onderzoek. Maar... er is een grote ‘maar’. Het is niet makkelijk om kwalitatief, en genoeg materiaal te verkrijgen om menselijke embryo’s te creëren die wij kunnen gebruiken voor ons onderzoek. Daarom kiezen wij ervoor om de muis als modelsysteem te gebruiken voor de mens in deze studie. Om embryo’s te verkrijgen van muizen, worden vrouwelijke muizen hiervoor behandeld met 2 hormonen om een zogenaamde superovulatie uit te lokken. Dit wil zeggen dat ze tot wel 30 eicellen (!) kunnen verkrijgen per cyclus. Dit laat ons toe om wel 30 embryo’s te gebruiken voor onderzoek per behandelde muis na paring met een mannetje.
Wat een (CRIS)Prachtige manier om gen functie te onderzoeken
Om de functie van bepaalde genen te onderzoeken, zullen we gebruik maken van het CRISPR/Cas9 gen bewerkingssysteem. CRISPR/Cas9 kan als het ware de schaar zetten in een bepaald stukje DNA, om op deze manier de functie van een gen te kunnen bestuderen (Figuur 2). Het CRISPR/Cas9 systeem kan je op maat ontwerpen, zodat het knipt in het stukje DNA van jouw keuze! Met deze knip genereren we een zogenaamde knock-out van een gen, zodat het zijn functie verliest.
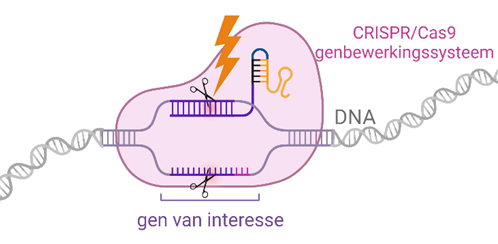
Figuur 2: Het CRISPR/Cas9 gen bewerkingssysteem knipt in het DNA om op deze manier een gen van interesse uit te schakelen.
Van enkele genen (Gata2, Gata3, Tfap2c en Tfap2a) hebben we echter een vermoeden dat ze cruciaal zijn voor de vorming van het trophectoderm, maar hun functie is nog niet uitgeklaard. Door CRISPR/Cas9 op maat te produceren om de Gata3, Gata2, Tfap2c en Tfap2a genen aan te vallen (om een knock-out te verkrijgen), kunnen we op deze manier hun functie te weten komen. Enkele vragen die we ons hierbij stellen zijn: ‘Zal het mogelijk zijn om het trophectoderm (en dus een blastocyst) te vormen bij knock-out van een van deze genen?’, ‘Zijn er verschillen tussen de ontwikkeling van knock-out embryo’s en normale embryo’s?’, ‘Zal er een vertraagde ontwikkeling optreden in knock-out embryo’s?’.
Maar... hoe?
Om de gen functie van Gata3, Gata2, Tfap2c en Tfap2a te onderzoeken, startten we na behandeling van de vrouwelijke muizen met het collecteren van embryo’s op het zygotestadium, en werd het CRISPR/Cas9 complex toegediend met een techniek die ook wel elektroporatie genoemd wordt. De naam zegt het zelf, door elektrische pulsen toe te dienen aan de zygotes, worden er tijdelijk poriën gemaakt in de celwand van de zygote, waardoor het CRISPR/Cas9 systeem in het embryo kon opgenomen worden, om zo het DNA te kunnen bereiken. Vervolgens maakt CRISPR/Cas9 een knip in het DNA van onze genen van interesse, zodat hun functie uitgeschakeld wordt. Op deze manier kunnen we door de embryo’s op te volgen zien of de normale ontwikkeling verstoord is, en kunnen we dus ook zien of deze genen belangrijk zijn in de vorming van het trophectoderm.
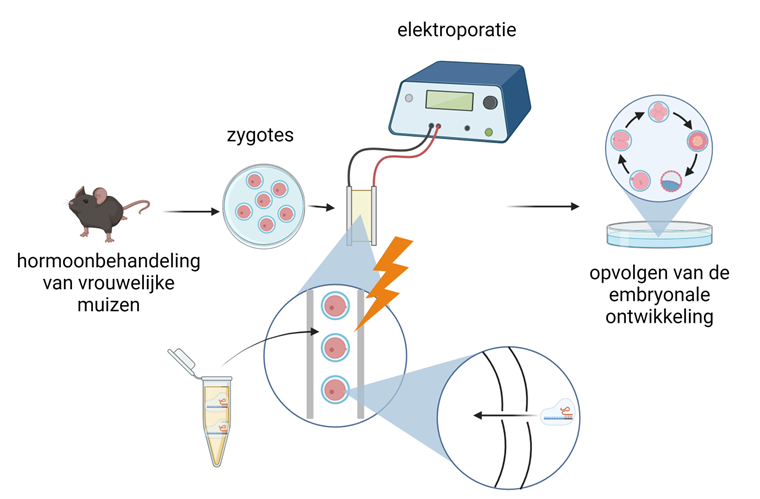
Figuur 3: Gevolgde methode voor het verkrijgen van knock-out muizenembryo’s.
Bevindingen
Uit onze resultaten bleek dat Tfap2c en Tfap2a wel degelijk een functie hebben in het vormen van het trophectoderm (en dus ook de blastocyst), aangezien een gestoorde ontwikkeling ontdekt werd bij Tfap2c en Tfap2a knock-out embryo’s. Echter, dit is niet het geval voor Gata2 en Gata3, aangezien een normale ontwikkeling geobserveerd werd in knock-out embryo’s. Dit onderzoek brengt ons dichter bij het ontrafelen van de geheimen die het begin van het leven (of de vroege embryonale ontwikkeling) voor ons schuilhoudt. Elke gen functie die ontrafeld wordt, is een stukje van de puzzel die gelegd wordt die ons dichter zal brengen bij de oplossing voor koppels die vandaag geen opties meer hebben om een genetisch eigen kind te verwekken.
Bibliografie
1 Adamson, G. D. et al. International Committee for Monitoring Assisted Reproductive Technology: world report on assisted reproductive technology, 2011. Fertility and sterility 110, 1067-1080 (2018).
2 Petraglia, F., Serour, G. I. & Chapron, C. The changing prevalence of infertility. International Journal of Gynecology & Obstetrics 123, S4-S8 (2013).
3 Fasouliotis, S. J. & Schenker, J. G. Failures in assisted reproductive technology: an overview. European Journal of Obstetrics and Gynecology and Reproductive Biology 107, 4-18 (2003).
4 Molè, M. A., Weberling, A. & Zernicka-Goetz, M. Comparative analysis of human and mouse development: From zygote to pre-gastrulation. Current topics in developmental biology 136, 113-138 (2020).
5 Chazaud, C. & Yamanaka, Y. Lineage specification in the mouse preimplantation embryo. Development 143, 1063-1074 (2016).
6 Niakan, K. K., Han, J., Pedersen, R. A., Simon, C. & Pera, R. A. R. Human pre-implantation embryo development. Development 139, 829-841 (2012).
7 White, M., Bissiere, S., Alvarez, Y. D. & Plachta, N. Mouse embryo compaction. Current topics in developmental biology 120, 235-258 (2016).
8 Zhu, M. et al. Developmental clock and mechanism of de novo polarization of the mouse embryo. Science 370, eabd2703 (2020).
9 Zhu, M. et al. Human embryo polarization requires PLC signaling to mediate trophectoderm specification. Elife 10, e65068 (2021).
10 Malassiné, A. & Cronier, L. Hormones and human trophoblast differentiation. Endocrine 19, 3-11 (2002).
11 Strumpf, D. et al. Cdx2 is required for correct cell fate specification and differentiation of trophectoderm in the mouse blastocyst. (2005).
12 Tanaka, S., Kunath, T., Hadjantonakis, A.-K., Nagy, A. & Rossant, J. Promotion of trophoblast stem cell proliferation by FGF4. Science 282, 2072-2075 (1998).
13 Chang, C.-W., Wakeland, A. K. & Parast, M. M. Trophoblast lineage specification, differentiation and their regulation by oxygen tension. Journal of Endocrinology 236, R43-R56 (2018).
14 James, J., Carter, A. M. & Chamley, L. Human placentation from nidation to 5 weeks of gestation. Part I: What do we know about formative placental development following implantation? Placenta 33, 327-334 (2012).
15 Louvet, S., Aghion, J., Santa-Maria, A., Mangeat, P. & Maro, B. Ezrin becomes restricted to outer cells following asymmetrical division in the preimplantation mouse embryo. Developmental biology 177, 568-579 (1996).
16 Plusa, B. et al. Downregulation of Par3 and aPKC function directs cells towards the ICM in the preimplantation mouse embryo. Journal of cell science 118, 505-515 (2005).
17 Hamatani, T., Carter, M. G., Sharov, A. A. & Ko, M. S. Dynamics of global gene expression changes during mouse preimplantation development. Developmental cell 6, 117-131 (2004).
18 Hirate, Y. et al. Polarity-dependent distribution of angiomotin localizes Hippo signaling in preimplantation embryos. Current biology 23, 1181-1194 (2013).
19 Sasaki, H. in Seminars in cell & developmental biology. 80-87 (Elsevier).
20 Wicklow, E. et al. HIPPO pathway members restrict SOX2 to the inner cell mass where it promotes ICM fates in the mouse blastocyst. PLoS genetics 10, e1004618 (2014).
21 Keramari, M. et al. Sox2 is essential for formation of trophectoderm in the preimplantation embryo. PloS one 5, e13952 (2010).
22 Mitsui, K. et al. The homeoprotein Nanog is required for maintenance of pluripotency in mouse epiblast and ES cells. Cell 113, 631-642 (2003).
23 Palmieri, S. L., Peter, W., Hess, H. & Schöler, H. R. Oct-4 transcription factor is differentially expressed in the mouse embryo during establishment of the first two extraembryonic cell lineages involved in implantation. Developmental biology 166, 259-267 (1994).
24 Karasek, C., Ashry, M., Driscoll, C. S. & Knott, J. G. A tale of two cell-fates: role of the Hippo signaling pathway and transcription factors in early lineage formation in mouse preimplantation embryos. Molecular human reproduction 26, 653-664 (2020).
25 Niwa, H., Miyazaki, J.-i. & Smith, A. G. Quantitative expression of Oct-3/4 defines differentiation, dedifferentiation or self-renewal of ES cells. Nature genetics 24, 372-376 (2000).
26 Nishioka, N. et al. The Hippo signaling pathway components Lats and Yap pattern Tead4 activity to distinguish mouse trophectoderm from inner cell mass. Developmental cell 16, 398-410 (2009).
27 Mihajlović, A. I. & Bruce, A. W. Rho-associated protein kinase regulates subcellular localisation of Angiomotin and Hippo-signalling during preimplantation mouse embryo development. Reproductive BioMedicine Online 33, 381-390 (2016).
28 Shi, X. et al. Rho differentially regulates the Hippo pathway by modulating the interaction between Amot and Nf2 in the blastocyst. Development 144, 3957-3967 (2017).
29 Graham, S. J. et al. BMP signalling regulates the pre-implantation development of extra-embryonic cell lineages in the mouse embryo. Nature communications 5, 1-11 (2014).
30 Home, P. et al. Altered subcellular localization of transcription factor TEAD4 regulates first mammalian cell lineage commitment. Proceedings of the National Academy of Sciences 109, 7362-7367 (2012).
31 De Paepe, C. et al. BMP4 plays a role in apoptosis during human preimplantation development. Molecular reproduction and development 86, 53-62 (2019).
32 Oron, E. & Ivanova, N. Cell fate regulation in early mammalian development. Physical biology 9, 045002 (2012).
33 Petropoulos, S. et al. Single-cell RNA-seq reveals lineage and X chromosome dynamics in human preimplantation embryos. Cell 165, 1012-1026 (2016).
34 Niakan, K. K. & Eggan, K. Analysis of human embryos from zygote to blastocyst reveals distinct gene expression patterns relative to the mouse. Developmental biology 375, 54-64 (2013).
35 Kuijk, E. W. et al. The roles of FGF and MAP kinase signaling in the segregation of the epiblast and hypoblast cell lineages in bovine and human embryos. Development 139, 871-882 (2012).
36 Christodoulou, N. et al. Morphogenesis of extra-embryonic tissues directs the remodelling of the mouse embryo at implantation. Nature communications 10, 1-12 (2019).
37 Mobley, R. J. et al. MAP3K4 controls the chromatin modifier HDAC6 during trophoblast stem cell epithelial-to-mesenchymal transition. Cell reports 18, 2387-2400 (2017).
38 Wang, J., Mayernik, L. & Armant, D. R. Trophoblast adhesion of the peri-implantation mouse blastocyst is regulated by integrin signaling that targets phospholipase C. Developmental biology 302, 143-153 (2007).
39 Hertig, A. T. & Rock, J. On the development of the early human ovum, with special reference to the trophoblast of the previllous stage: A desoription of 7 normal and 5 pathologic human ova. American Journal of Obstetrics and Gynecology 47, 149-184 (1944).
40 Lindenberg, S., Hyttel, P., Sjøgren, A. & Greve, T. A comparative study of attachment of human, bovine and mouse blastocysts to uterine epithelial monolayer. Human Reproduction 4, 446-456 (1989).
41 Aplin, J. D. & Ruane, P. T. Embryo–epithelium interactions during implantation at a glance. Journal of cell science 130, 15-22 (2017).
42 Weimar, C. H., Uiterweer, E. D. P., Teklenburg, G., Heijnen, C. J. & Macklon, N. S. In-vitro model systems for the study of human embryo–endometrium interactions. Reproductive biomedicine online 27, 461-476 (2013).
43 Lv, B. et al. Single-cell RNA sequencing reveals regulatory mechanism for trophoblast cell-fate divergence in human peri-implantation conceptuses. PLoS biology 17, e3000187 (2019).
44 Johnson, M. & Maro, B. The distribution of cytoplasmic actin in mouse 8-cell blastomeres. (1984).
45 Nikas, G., Ao, A., Winston, R. M. & Handyside, A. H. Compaction and surface polarity in the human embryo in vitro. Biology of reproduction 55, 32-37 (1996).
46 Stamatiadis, P. et al. TEAD4 regulates trophectoderm differentiation upstream of CDX2 in a GATA3-independent manner in the human preimplantation embryo. Human Reproduction 37, 1760-1773 (2022).
47 Fogarty, N. M. et al. Genome editing reveals a role for OCT4 in human embryogenesis. Nature 550, 67-73 (2017).
48 Stamatiadis, P. et al. Comparative analysis of mouse and human preimplantation development following POU5F1 CRISPR/Cas9 targeting reveals interspecies differences. Human Reproduction 36, 1242-1252 (2021).
49 Yamanaka, Y., Lanner, F. & Rossant, J. FGF signal-dependent segregation of primitive endoderm and epiblast in the mouse blastocyst. Development 137, 715-724 (2010).
50 Blakeley, P. et al. Defining the three cell lineages of the human blastocyst by single-cell RNA-seq. Development 142, 3151-3165 (2015).
51 Hertig, A. T., Rock, J. & Adams, E. C. A description of 34 human ova within the first 17 days of development. American Journal of Anatomy 98, 435-493 (1956).
52 Christodoulou, N. et al. Sequential formation and resolution of multiple rosettes drive embryo remodelling after implantation. Nature cell biology 20, 1278-1289 (2018).
53 Eckert, D., Buhl, S., Weber, S., Jäger, R. & Schorle, H. The AP-2 family of transcription factors. Genome biology 6, 1-8 (2005).
54 Li, H., Sheridan, R. & Williams, T. Analysis of TFAP2A mutations in branchio-oculo-facial syndrome indicates functional complexity within the AP-2α DNA-binding domain. Human molecular genetics 22, 3195-3206 (2013).
55 Meier, P., Koedood, M., Philipp, J., Fontana, A. & Mitchell, P. J. Alternative mRNAs encode multiple isoforms of transcription factor AP-2 during murine embryogenesis. Developmental biology 169, 1-14 (1995).
56 Ohtaka-Maruyama, C., Hanaoka, F. & Chepelinsky, A. B. A novel alternative spliced variant of the transcription factor AP2α is expressed in the murine ocular lens. Developmental biology 202, 125-135 (1998).
57 Bauer, R. et al. The genomic structure of the human AP-2 transcription factor. Nucleic acids research 22, 1413-1420 (1994).
58 Hilger-Eversheim, K., Moser, M., Schorle, H. & Buettner, R. Regulatory roles of AP-2 transcription factors in vertebrate development, apoptosis and cell-cycle control. Gene 260, 1-12 (2000).
59 Bai, Q. et al. Dissecting the first transcriptional divergence during human embryonic development. Stem Cell Reviews and Reports 8, 150-162 (2012).
60 Kuckenberg, P., Kubaczka, C. & Schorle, H. The role of transcription factor Tcfap2c/TFAP2C in trophectoderm development. Reproductive biomedicine online 25, 12-20 (2012).
61 Winger, Q., Huang, J., Auman, H. J., Lewandoski, M. & Williams, T. Analysis of transcription factor AP-2 expression and function during mouse preimplantation development. Biology of reproduction 75, 324-333 (2006).
62 Richardson, B. D., Cheng, Y.-H., Langland, R. A. & Handwerger, S. Differential expression of AP-2γ and AP-2α during human trophoblast differentiation. Life sciences 69, 2157-2165 (2001).
63 Viger, R. S., Guittot, S. M., Anttonen, M., Wilson, D. B. & Heikinheimo, M. Role of the GATA family of transcription factors in endocrine development, function, and disease. Molecular endocrinology 22, 781-798 (2008).
64 Bates, D. L., Chen, Y., Kim, G., Guo, L. & Chen, L. Crystal structures of multiple GATA zinc fingers bound to DNA reveal new insights into DNA recognition and self-association by GATA. Journal of molecular biology 381, 1292-1306 (2008).
65 Zhou, Y. et al. Dynamic regulation of GATA2 in fate determination in hematopoiesis: possible approach to hPSC-derived hematopoietic stem/progenitor cells. Blood Science 2, 1-6 (2020).
66 Adomas, A. B. et al. Breast tumor specific mutation in GATA3 affects physiological mechanisms regulating transcription factor turnover. BMC cancer 14, 1-14 (2014).
67 Cosemans, G. et al. CRISPR/Cas9 mediated knock-out (KO) reveals a divergent role for trophectoderm markers GATA2/3 in the mouse and human preimplantation embryo. HUMAN REPRODUCTION 37 (2022).
68 Home, P. et al. GATA3 is selectively expressed in the trophectoderm of peri-implantation embryo and directly regulates Cdx2 gene expression. Journal of Biological Chemistry 284, 28729-28737 (2009).
69 Home, P. et al. Genetic redundancy of GATA factors in the extraembryonic trophoblast lineage ensures the progression of preimplantation and postimplantation mammalian development. Development 144, 876-888 (2017).
70 Bai, H., Sakurai, T., Godkin, J. D. & Imakawa, K. Expression and potential role of GATA factors in trophoblast development. Journal of Reproduction and Development 59, 1-6 (2013).
71 Krendl, C. et al. GATA2/3-TFAP2A/C transcription factor network couples human pluripotent stem cell differentiation to trophectoderm with repression of pluripotency. Proceedings of the National Academy of Sciences 114, E9579-E9588 (2017).
72 Ma, Y., Zhang, L. & Huang, X. Genome modification by CRISPR/Cas9. The FEBS journal 281, 5186-5193 (2014).
73 Redman, M., King, A., Watson, C. & King, D. What is CRISPR/Cas9? Archives of Disease in Childhood-Education and Practice 101, 213-215 (2016).
74 Bekaert, B. et al. in Seminars in Cell & Developmental Biology. (Elsevier).
75 Yang, H. et al. Methods favoring homology-directed repair choice in response to CRISPR/Cas9 induced-double strand breaks. International journal of molecular sciences 21, 6461 (2020).
76 Jang, H.-K., Song, B., Hwang, G.-H. & Bae, S. Current trends in gene recovery mediated by the CRISPR-Cas system. Experimental & Molecular Medicine 52, 1016-1027 (2020).
77 Tuladhar, R. et al. CRISPR-Cas9-based mutagenesis frequently provokes on-target mRNA misregulation. Nature communications 10, 4056 (2019).
78 Dumitrache, L. C. et al. Trex2 enables spontaneous sister chromatid exchanges without facilitating DNA double-strand break repair. Genetics 188, 787-797 (2011).
79 Liu, M. et al. Methodologies for improving HDR efficiency. Frontiers in genetics 9, 691 (2019).
80 Ata, H. et al. Robust activation of microhomology-mediated end joining for precision gene editing applications. PLoS genetics 14, e1007652 (2018).
81 Manghwar, H. et al. CRISPR/Cas systems in genome editing: methodologies and tools for sgRNA design, off‐target evaluation, and strategies to mitigate off‐target effects. Advanced science 7, 1902312 (2020).
82 Kim, S., Kim, D., Cho, S. W., Kim, J. & Kim, J.-S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome research 24, 1012-1019 (2014).
83 Lamas-Toranzo, I. et al. Strategies to reduce genetic mosaicism following CRISPR-mediated genome edition in bovine embryos. Scientific reports 9, 1-8 (2019).
84 Adikusuma, F. et al. Large deletions induced by Cas9 cleavage. Nature 560, E8-E9 (2018).
85 Egli, D. et al. Inter-homologue repair in fertilized human eggs? Nature 560, E5-E7 (2018).
86 Lee, H. & Kim, J.-S. Unexpected CRISPR on-target effects. Nature biotechnology 36, 703-704 (2018).
87 Alanis-Lobato, G. et al. Frequent loss of heterozygosity in CRISPR-Cas9–edited early human embryos. Proceedings of the National Academy of Sciences 118, e2004832117 (2021).
88 Togi, S., Ura, H. & Niida, Y. Optimization and Validation of Multimodular, Long-Range PCR–Based Next-Generation Sequencing Assays for Comprehensive Detection of Mutation in Tuberous Sclerosis Complex. The Journal of Molecular Diagnostics 23, 424-446 (2021).
89 Deleye, L. et al. Shallow whole genome sequencing is well suited for the detection of chromosomal aberrations in human blastocysts. Fertility and sterility 104, 1276-1285. e1271 (2015).
90 Scholefield, J. & Harrison, P. T. Prime editing–an update on the field. Gene Therapy 28, 396-401 (2021).
91 Anzalone, A. V. et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature 576, 149-157 (2019).
92 Porto, E. M., Komor, A. C., Slaymaker, I. M. & Yeo, G. W. Base editing: advances and therapeutic opportunities. Nature Reviews Drug Discovery 19, 839-859 (2020).
93 Gaudelli, N. M. et al. Programmable base editing of A• T to G• C in genomic DNA without DNA cleavage. Nature 551, 464-471 (2017).
94 Habib, O., Habib, G., Hwang, G.-H. & Bae, S. Comprehensive analysis of prime editing outcomes in human embryonic stem cells. Nucleic acids research 50, 1187-1197 (2022).
95 Liang, P. et al. Correction of β-thalassemia mutant by base editor in human embryos. Protein & cell 8, 811-822 (2017).
96 Liang, P. et al. Effective gene editing by high-fidelity base editor 2 in mouse zygotes. Protein & cell 8, 601-611 (2017).
97 Schene, I. F. et al. Prime editing for functional repair in patient-derived disease models. Nature communications 11, 5352 (2020).
98 Mou, H. et al. CRISPR/Cas9-mediated genome editing induces exon skipping by alternative splicing or exon deletion. Genome biology 18, 1-8 (2017).
99 Boel, A. et al. BATCH-GE: Batch analysis of Next-Generation Sequencing data for genome editing assessment. Scientific reports 6, 1-10 (2016).
100 Robinson, J. T. et al. Integrative genomics viewer. Nature biotechnology 29, 24-26 (2011).
101 Bai, Q. et al. Temporal analysis of genome alterations induced by single-cell passaging in human embryonic stem cells. Stem cells and development 24, 653-662 (2015).
102 Hall, B. et al. Genome editing in mice using CRISPR/Cas9 technology. Current Protocols in Cell Biology 81, e57 (2018).
103 Ren, F. et al. Efficiency optimization of CRISPR/Cas9-mediated targeted mutagenesis in grape. Frontiers in plant science 10, 612 (2019).
104 Ren, X. et al. Enhanced specificity and efficiency of the CRISPR/Cas9 system with optimized sgRNA parameters in Drosophila. Cell reports 9, 1151-1162 (2014).
105 Pandolfi, P. P. et al. Targeted disruption of the GATA3 gene causes severe abnormalities in the nervous system and in fetal liver haematopoiesis. Nature genetics 11, 40-44 (1995).
106 Ma, G. T. et al. GATA-2 and GATA-3 regulate trophoblast-specific gene expression in vivo. Development 124, 907-914 (1997).
107 Gaztelumendi, N. & Nogués, C. Chromosome instability in mouse embryonic stem cells. Scientific Reports 4, 1-8 (2014).
108 Li, J. et al. Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9. Journal of molecular cell biology 7, 284-298 (2015).
109 Tfap2a. (2023, February). Retrieved from Ensembl: https://www.ensembl.org/Mus_musculus/Gene/Summary?db=core;g=ENSMUSG0000…
110 TFAP2A. (2023, February). Retrieved from Ensembl: https://www.ensembl.org/Homo_sapiens/Gene/Summary?db=core;g=ENSG0000013…
111 Tfap2c. (2023, February). Retrieved from Ensembl: https://www.ensembl.org/Mus_musculus/Gene/Summary?db=core;g=ENSMUSG0000…
112 TFAP2C. (2023, February). Retrieved from Ensembl: https://www.ensembl.org/Homo_sapiens/Gene/Summary?db=core;g=ENSG0000008…
113 Gata2. (2023, February). Retrieved from Ensembl: https://www.ensembl.org/Mus_musculus/Gene/Summary?db=core;g=ENSMUSG0000…
114 GATA2. (2023, February). Retrieved from Ensembl: https://www.ensembl.org/Homo_sapiens/Gene/Summary?db=core;g=ENSG0000017…
115 Gata3. (2023, February). Retrieved from Ensembl: https://www.ensembl.org/Mus_musculus/Gene/Summary?db=core;g=ENSMUSG0000…
116 GATA3. (2023, February). Retrieved from Ensembl: https://www.ensembl.org/Homo_sapiens/Gene/Summary?db=core;g=ENSG0000010…
117 What is fragment analysis? (n.d.). Retrieved from Thermo Fisher Scientific: https://www.thermofisher.com/be/en/home/life-science/sequencing/sequenc….
119 Integrative Genomics Viewer. Retrieved from https://software.broadinstitute.org/software/igv/
All figures were created using www.biorender.com.









