Van afval naar biobrandstof
De klimaatverandering, uitstoot van enorme hoeveelheden broeikasgassen, uitputting van fossiele brandstoffen, … Het zijn grote problemen die ons allemaal aangaan maar waarvoor we vandaag nog steeds geen concrete oplossing hebben. Oplossingen voor complexe problemen liggen vaak verborgen in de genialiteit van de natuur. Wat als ik u vertel dat we onze CO2 uitstoot drastisch kunnen verlagen door gebruik te maken van gespecialiseerde multi-enzymcomplexen, gebouwd met Legoblokjes…
‘I want Europe to become the first climate-neutral continent in the world by 2050’ kondigt Ursula von der Leyen vastberaden aan op de persconferentie van de European Green Deal. Een klimaatneutraal Europa betekent dat ons continent niet langer zal bijdragen aan de opwarming van de aarde door de uitstoot van broeikasgassen, zoals CO2.
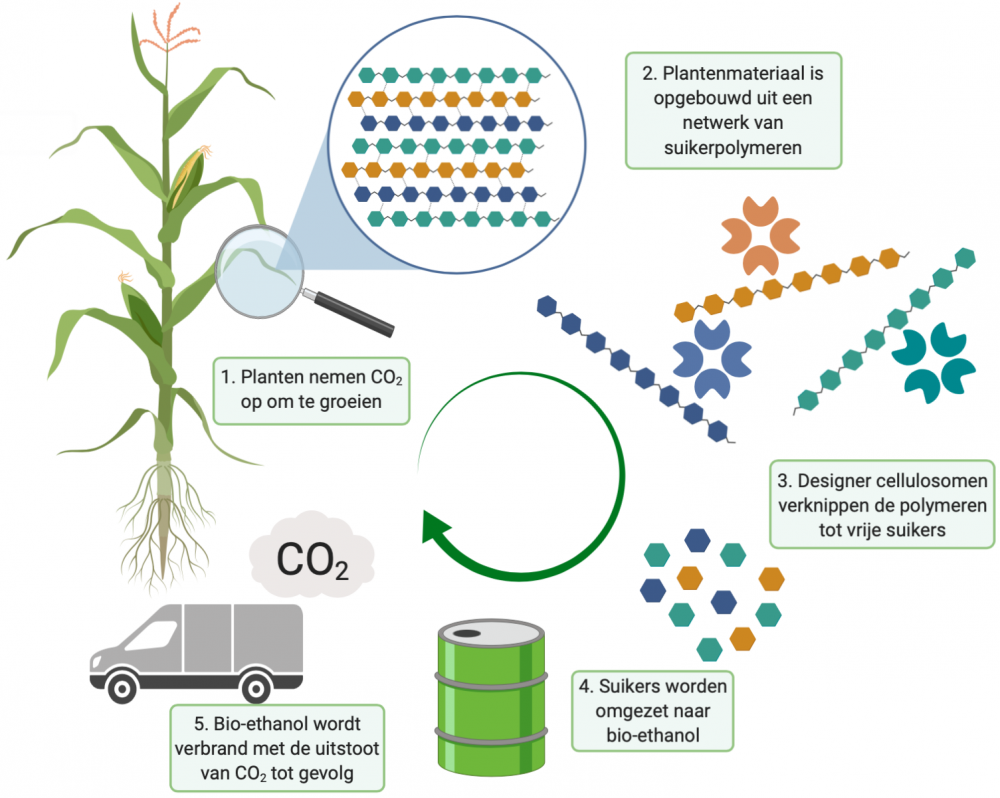
Eén van de belangrijkste strategieën waar vandaag op ingezet wordt om onze CO2 uitstoot te reduceren is bio-ethanol. Wanneer bio-ethanol verbrand wordt in de motor van een auto komt, net zoals bij de verbranding van benzine, CO2 vrij. Maar omdat bio-ethanol geproduceerd wordt uit de suikers aanwezig in plantaardig materiaal, wordt een gesloten cyclus gevormd: CO2 wordt uitgestoten en komt in de atmosfeer terecht. Deze wordt opgenomen door planten die CO2 nodig hebben om te groeien en van die planten kan dan weer nieuwe bio-ethanol gemaakt worden.
Jammer genoeg is plantenmateriaal een zeer complexe grondstof, wat de ontwikkeling en commercialisering van bio-ethanol sterk vertraagt. Daarom besloot ik om met mijn thesisonderzoek mijn steentje bij te dragen binnen deze problematiek.
Food vs. fuel
Waarschijnlijk hebben de benzinerijders onder u ook al eens bio-ethanol getankt. De E5 en E10 brandstoffen bestaan namelijk voor 5 en 10% uit eerste generatie bio-ethanol. De suikers die gebruikt worden als startmateriaal zijn afkomstig van de eetbare delen van de plant, bijvoorbeeld zetmeel uit tarwe of mais. Deze suikers kunnen door gist gefermenteerd worden tot ethanol. De publieke opinie over het gebruik van voedingsgewassen voor de productie van biobrandstoffen is echter negatief, zo bleek uit de ‘food vs fuel’ discussie. Het voelt inderdaad niet juist dat men in de Verenigde Staten duizenden hectares mais aanplant voor bio-ethanol productie, terwijl meer dan 10% van de Amerikanen honger lijdt.
Stel je nu eens voor dat men al deze maiskolven kan consumeren, terwijl er van het landbouwafval, zoals de maisstengels, bio-ethanol gemaakt wordt. Met andere woorden, de suikers die we fermenteren tot bio-ethanol zijn afkomstig van de niet-eetbare delen van de plant en gaan dus niet in competitie met de voedingsketen. Hier spreekt men van tweede generatie bio-ethanol, en dit is waar mijn onderzoek begint.
Plantencelwanden bestaan uit parelsnoeren
Het plantenmateriaal dat omgezet wordt naar tweede generatie bio-ethanol bevat lignocellulose. Lignocellulose is aanwezig in elke plantencelwand en opgebouwd uit een complex netwerk van twee suikerpolymeren, cellulose en hemicellulose, en een aromatisch polymeer, lignine. Een polymeer is een lange keten van enkelvoudige moleculen, zoals een parelsnoer bestaand uit een aaneenschakeling van meerdere parels. Bij (hemi)cellulose zijn deze ‘parels’ suikermonomeren.
De drie polymeren in lignocellulose zijn sterk met elkaar verbonden en vormen een kluwen van verschillende ketens, zoals meerdere parelsnoeren die sterk in de knoop zitten. Dit maakt het erg moeilijk om hieruit suikers vrij te stellen. Bovendien is de verhouding en samenstelling van de drie polymeren sterk afhankelijk van de plantensoort, de regio en het seizoen. Tot op vandaag bestaat er nog geen robuuste en kostenefficiënte methode om lignocellulose beschikbaar te maken.
Gespecialiseerd multi-enzymcomplex
Gelukkig heeft de natuur een oplossing gevonden voor de afbraak van deze complexe structuur. Bacteriën gebruiken lignocellulose als voedingsbron. Aerobe bacteriën produceren hiervoor grote hoeveelheden vrije enzymen. De enzymen functioneren als schaartjes, ze kunnen op verschillende plaatsen de ketens knippen. Zo vallen de polymeren uiteen in enkelvoudige suikers, dewelke de bacterie kan opnemen en gebruiken om te groeien.
Anaerobe bacteriën hebben gespecialiseerde multi-enzymcomplexen ontwikkeld om deze taak uit te voeren. Zo’n complex noemen we een cellulosoom. Het bestaat uit een ruggengraat en verschillende enzymen die zich op deze ruggengraat kunnen vastklikken, via een binding die doet denken aan een stekker -het dockerine- en een stopcontact -het cohesine. Bovendien kan het volledige complex zich vasthechten aan één van de polymeren. Dit zorgt er, in combinatie met de fysieke nabijheid van de enzymen, voor dat een cellulosoom het polymeernetwerk op een veel efficiëntere manier kan afbreken dan vrije enzymen.
Eén cellulosoom bestaat uit verschillende modules: cohesines, dockerines, enzymen, … Deze modules kunnen onafhankelijk van elkaar functioneren. Zo kwam het idee om modules afkomstig van verschillende natuurlijke cellulosomen te combineren tot een designer cellulosoom. In een designer cellulosoom kunnen wetenschappers zelf bepalen welke enzymen opgenomen worden in het complex en op welke positie ze komen te staan.
Tijdens mijn thesisonderzoek heb ik mijn eigen designer cellulosoom gemaakt. Dit cellulosoom is gespecialiseerd in de afbraak van xyloglucaan, één van de suikerpolymeren aanwezig in lignocellulose. Hiervoor werden vier enzymen (endoxyloglucanase, β-galactosidase, α-xylosidase en β-glucosidase) bijeengebracht die samenwerken om xyloglucaan te verknippen tot vrije suikers.
Spelen met Lego
Een designer cellulosoom is opgebouwd uit meerdere modules, daarom vraagt de constructie op DNA-niveau veel tijd. Om deze bottleneck aan te pakken heeft onze onderzoeksgroep de VersaTile techniek ontwikkeld. De VersaTile techniek uitvoeren is net zoals spelen met Legoblokjes. Eerst kies je de blokjes die je wil gebruiken en bepaal je op welke positie deze moeten komen. Vervolgens worden de blokjes gecombineerd volgens een welbepaald design. Zo wordt elke module van het cellulosoom snel aangemaakt. Daarna kunnen deze modules gecombineerd worden tot een multi-enzymcomplex.
Meer designer cellulosomen, minder CO2!
Met mijn thesisonderzoek heb ik bewezen dat het mogelijk is om een designer cellulosoom te creëren dat xyloglucaan kan afbreken tot vrije suikers. ‘Maar hoe gaan we daar onze CO2 uitstoot mee verlagen?’, hoor ik u nu denken. Wel, er kunnen oneindig veel verschillende designer cellulosomen aangemaakt worden. Door voor ieder polymeer aanwezig in lignocellulose een gespecialiseerd cellulosoom te ontwerpen, kan het polymeernetwerk volledig afgebroken worden tot fermenteerbare suikers. Zo kan plantenmateriaal zoals landbouwafval omgezet worden tot bio-ethanol, en wordt een gesloten CO2 cyclus gevormd. Een oplossing die niets te vroeg komt, want over zeven jaar is het koolstofbudget van onze planeet uitgeput.
Bibliografie
Arfi, Y., Shamshoum, M., Rogachev, I., Peleg, Y. & Bayer, E. A. (2014). Integration of bacterial lytic polysaccharide monooxygenases into designer cellulosomes promotes enhanced cellulose degradation. Proceedings of the National Academy of Sciences of the United States of America, 111(25), 9109–9114.
Artzi, L., Bayer, E. A. & Moraïs, S. (2017). Cellulosomes: Bacterial nanomachines for dismantling plant polysaccharides. Nature Reviews Microbiology, 15(2), 83–95.
Attia, M. A., Nelson, C. E., Offen, W. A., Jain, N., Davies, G. J., Gardner, J. G. & Brumer, H. (2018). In vitro and in vivo characterization of three Cellvibrio japonicus glycoside hydrolase family 5 members reveals potent xyloglucan backbone-cleaving functions. Biotechnology for Biofuels, 11(1), 1–16.
Baruah, J., Nath, B. K., Sharma, R., Kumar, S., Deka, R. C., Baruah, D. C. & Kalita, E. (2018). Recent trends in the pretreatment of lignocellulosic biomass for value-added products. Frontiers in Energy Research, 6(DEC), 1–19.
Bayer, E. A. (2017). Cellulosomes and designer cellulosomes: why toy with Nature? Environmental Microbiology Reports, 9(1), 14–15.
Bayer, E. A., Belaich, J.-P., Shoham, Y. & Lamed, R. (2004). The Cellulosomes: Multienzyme Machines for Degradation of Plant Cell Wall Polysaccharides. Annual Review of Microbiology, 58(1), 521–554.
Beylot, M. H., McKie, V. A., Voragen, A. G. J., Doeswijk-Voragen, C. H. L. & Gilbert, H. J. (2001). The Pseudomonas cellulosa glycoside hydrolase family 51 arabinofuranosidase exhibits wide substrate specificity. Biochemical Journal, 358(3), 607–614.
Bloomberg New Energy Finance. (2010). Next-generation ethanol and biochemicals : what ’ s in it for Europe ?
Cappellaro, C., Hauser, K., Mrśa, V., Watzele, M., Watzele, G., Gruber, C. & Tanner, W. (1991). Saccharomyces cerevisiae a- and alpha-agglutinin: characterization of their molecular interaction. The EMBO Journal, 10(13), 4081–4088.
Carlu, L. (2019). De ontwikkeling van een trivalent designer cellulosoom voor een efficiënte degradatie van xyloglucaan. (Masterproef), Gent, Universitiet Gent.
Carvalho, A. L., Dias, F. M. V., Prates, J. A. M., Nagy, T., Gilbert, H. J., Davies, G. J., Ferreira,L. M. A., Romão, M. J. & Fontes, C. M.G.A. (2003).Cellulosome assembly revealed by the crystal structure of the cohesin-dockerin complex. Proceedings of the National Academy of Sciences of the United States of America, 100(24), 13809–13814.
Caspi, J., Barak, Y., Haimovitz, R., Irwin, D., Lamed, R., Wilson, D. B. & Bayer, E. A. (2009). Effect of linker length and dockerin position on conversion of a Thermobifida fusca endoglucanase to the cellulosomal mode. Applied and Environmental Microbiology, 75(23), 7335–7342.
Chauvaux, S., Beguin, P., Aubert, J. P., Bhat, K. M., Gow, L. A., Wood, T. M. & Bairoch, A. (1990). Calcium-binding affinity and calcium-enhanced activity of Clostridium thermocellum endoglucanase D. Biochemical Journal, 265(1), 261–265.
Chen, X., Zaro, J. L. & Shen, W. C. (2013). Fusion protein linkers: Property, design and functionality. Advanced Drug Delivery Reviews, 65(10), 1357–1369.
Cherf G.M. & Cochran J.R. (2015) Applications of Yeast Surface Display for Protein Engineering. In Liu B. (ed) Yeast Surface Display. Methods in Molecular Biology, vol 1319 (p. 155-175). New York: Humana Press.
CINBIOS. (2015). Industriële Biotechnologie : een roadmap voor Vlaanderen.
Cutillas-Iturralde, A., Zarra, I., Fry, S. C. & Lorences, E. P. (1994). Implication of persimmon fruit hemicellulose metabolism in the softening process. Importance of xyloglucan endotransglycosylase. Physiologia Plantarum, 91(2), 169–176.
Dassa, B., Borovok, I., Ruimy-Israeli, V., Lamed, R., Flint, H. J., Duncan, S. H. Henrissat, B.,
Coutinho, P., Morrison, M., Mosoni, P., Yeoman, C. J., White, B. A. & Bayer, E. A. (2014). Rumen cellulosomics: Divergent fiber-degrading strategies revealed by comparative genome-wide analysis of six ruminococcal strains. PLoS ONE, 9(7).
Davidi, L., Moraïs, S., Artzi, L., Knop, D., Hadar, Y., Arfi, Y. & Bayer, E. A. (2016). Toward combined delignification and saccharification of wheat straw by a laccase-containing designer cellulosome. Proceedings of the National Academy of Sciences of the United States of America, 113(39), 10854–10859.
DeBoy, R. T., Mongodin, E. F., Fouts, D. E., Tailford, L. E., Khouri, H., Emerson, J. B.,
Mohamoud, Y., Watkins, K., Henrissat, B., Gilbert, H. J. & Nelson, K. E. (2008). Insights into plant cell wall degradation from the genome sequence of the soil bacterium Cellvibrio japonicus. Journal of Bacteriology, 190(15), 5455–5463.
Dueber, J. E., Wu, G. C., Malmirchegini, G. R., Moon, T. S., Petzold, C. J., Ullal, A. V., Prather, K. L. J. & Keasling, J. D. (2009). Synthetic protein scaffolds provide modular control over metabolic flux. Nature Biotechnology, 27(8), 753–759.
Elicityl. (2012). Oligotech: the library of complex sugars. Poduct catalogue 4th ed.
Engler, C., Gruetzner, R., Kandzia, R. & Marillonnet, S. (2009). Golden gate shuffling: A one-pot DNA shuffling method based on type ils restriction enzymes. PLoS ONE, 4(5).
Fan, L. H., Zhang, Z. J., Yu, X. Y., Xue, Y. X. & Tan, T. W. (2012). Self-surface assembly of cellulosomes with two miniscaffoldins on Saccharomyces cerevisiae for cellulosic ethanol production. Proceedings of the National Academy of Sciences of the United States of America, 109(33), 13260–13265.
Fierobe, H. P., Mingardon, F., Mechaly, A., Bélaïch, A., Rincon, M. T., Pagès, S., Lamed, R.,
Tardif, C., Bélaïch, J. P. & Bayer, E. A. (2005). Action of designer cellulosomes on homogeneous Versus complex substrates: Controlled incorporation of three distinct enzymes into a defined trifunctional scaffoldin. Journal of Biological Chemistry, 280(16), 16325–16334.
Fry, S. C. (1989). The Structure and Functions of Xyloglucan. Journal of Experimental Botany, 40(210), 1–11.
Gardner, J. G. (2016). Polysaccharide degradation systems of the saprophytic bacterium Cellvibrio japonicus. World Journal of Microbiology and Biotechnology, 32(7), 1–12.
Gunnoo, M., Cazade, P. A., Galera-Prat, A., Nash, M. A., Czjzek, M., Cieplak, M., Alvarez B.,Aguilar M., Karpol A., Gaub H., Carrión-Vázquez M., Bayer E.A. & Thompson, D. (2016). Nanoscale Engineering of Designer Cellulosomes. Advanced Materials, 28(27), 5619–5647.
Harmsen, P., Huijgen, W., Bermudez, L. & Bakker, R. (2010). Literature review of physical and chemical pretreatment processes for lignocellulosic biomass. (Rapport), Wageningen, Wageningen UR Food & Biobased Research.
Hayashi, T. (1989). Xyloglucans in the primary cell wall. Annu. Rev. Plant Physiol. Plant Mol. Biol., 40, 139–168.
Hayashi, T. & Kaida, R. (2011). Functions of xyloglucan in plant cells. Molecular Plant, 4(1), 17–24.
Heinze, T., El Seoud, O. & Koschella, A. (2018). Cellulose Derivatives: Synthesis, Structure, and Properties. Cham: Springer International Publishing.
Hernandez-Gomez, M. C., Rydahl, M. G., Rogowski, A., Morland, C., Cartmell, A., Crouch,
L., Labourel, A., Fontes, C. M. G. A., Willats, W. G. T., Gilbert, H. J. & Knox, J. P. (2015). Recognition of xyloglucan by the crystalline cellulose-binding site of a family 3a carbohydrate-binding module. FEBS Letters, 589(18), 2297–2303.
Horn, S. J., Vaaje-kolstad, G., Westereng, B. & Eijsink, V. G. (2012). Novel enzymes for the degradation of cellulose. Biotechnology for Biofuels 5, 45.
Humphry, D. R., Black, G. W. & Cummings, S. P. (2003). Reclassification of 'Pseudomonas fluorescens subsp. cellulosa' NCIMB 10462 (Ueda et al. 1952) as Cellvibrio japonicus sp. nov. and revival of Cellvibrio vulgaris sp. nov., nom. rev. and Cellvibrio fulvus sp. nov., nom. rev. International Journal of Systematic and Evolutionary Microbiology, 53(2), 393–400.
Kim, S., Baek, S., Lee, K. & Hahn, J. (2013). Cellulosic ethanol production using a yeast consortium displaying a minicellulosome and β-glucosidase. Microbial Cell Factories, 12(14).
Kondo, A. & Ueda, M. (2004). Yeast cell-surface display - Applications of molecular display. Applied Microbiology and Biotechnology, 64(1), 28–40.
Larsbrink, J., Thompson, A. J., Lundqvist, M., Gardner, J. G., Davies, G. J. & Brumer, H. (2014). A complex gene locus enables xyloglucan utilization in the model saprophyte Cellvibrio japonicus. Molecular Microbiology, 94(2), 418–433.
Lipke, P. N., & Kurjan, J. (1992). Sexual agglutination in budding yeasts: Structure, function, and regulation of adhesion glycoproteins. Microbiological Reviews, 56(1), 180–194.
Liu, Q., Luo, L. & Zheng, L. (2018). Lignins: Biosynthesis and biological functions in plants. International Journal of Molecular Sciences, 19(2).
Mechaly, A., Fierobe, H. P., Belaich, A., Belaich, J. P., Lamed, R., Shoham, Y. & Bayer, E. A. (2001). Cohesin-dockerin interaction in cellulosome assembly: A single hydroxyl group of a dockerin domain distinguishes between nonrecognition and high affinity recognition. Journal of Biological Chemistry, 276(13), 9883–9888.
Miedes, E. & Lorences, E. P. (2004). Apple (Malus domestica) and tomato (Lycopersicum esculentum) fruits cell-wall hemicelluloses and xyloglucan degradation during Penicillium expansum infection. Journal of Agricultural and Food Chemistry, 52(26), 7957–7963.
Moon, T. S., Yoon, S. H., Lanza, A. M., Roy-Mayhew, J. D. & Jones Prather, K. L. (2009). Production of glucaric acid from a synthetic pathway in recombinant Escherichia coli. Applied and Environmental Microbiology, 75(3), 589–595.
Moraïs, S., Barak, Y., Caspi, J., Hadar, Y., Lamed, R., Shoham, Y., Wilson, D. B. & Bayer, E. A. (2010). Cellulase-xylanase synergy in designer cellulosomes for enhanced degradation of a complex cellulosic substrate. mBio, 1(5).
Moraïs, S., Morag, E., Barak, Y., Goldman, D., Hadar, Y., Lamed, R., Shoham, Y., Wilson, D. B., & Bayer E. A. (2012). Deconstruction of lignocellulose into soluble sugars by native and designer cellulosomes. mBio, 3(6).
Moraïs, S., Stern, J., Kahn, A., Galanopoulou, A. P., Yoav, S., Shamshoum, M., Smith, M. A., Hatzinikolaou, D. G., Arnold, F. H. & Bayer, E. A. (2016). Enhancement of cellulosome-mediated deconstruction of cellulose by improving enzyme thermostability. Biotechnology for biofuels, 9, 164.
Nash, M. A., Smith, S. P., Fontes, C. M. & Bayer, E. A. (2016). Single versus dual-binding conformations in cellulosomal cohesin–dockerin complexes. Current Opinion in Structural Biology, 40, 89–96.
Packer, M. S. & Liu, D. R. (2015). Methods for the directed evolution of proteins. Nature Reviews Genetics, 16(7), 379–394.
Pagès, S., Bélaïch, A., Bélaïch, J. P., Morag, E., Lamed, R., Shoham, Y. & Bayer, E. A. (1997). Species-specificity of the cohesin-dockerin interaction between Clostridium thermocellum and Clostridium cellulolyticum: Prediction of specificity determinants of the dockerin domain. Proteins: Structure, Function and Genetics, 29(4), 517–527.
Park, Y. B. & Cosgrove, D. J. (2015). Xyloglucan and its interactions with other components of the growing cell wall. Plant and Cell Physiology, 56(2), 180–194.
Rana, V. & Rana, D. (2017). Renewable biofuels : bioconversion of lignocellulosic biomass by microbial community. Cham: Springer International Publishing.
Salamitou, S., Tokatlidis, K., Béguin, P. & Aubert, J. P. (1992). Involvement of separate domains of the cellulosomal protein S1 of Clostridium thermocellum in binding to cellulose and in anchoring of catalytic subunits to the cellulosome. FEBS Letters, 304(1), 89–92.
Seidl, P. R. & Goulart, A. K. (2016). Pretreatment processes for lignocellulosic biomass conversion to biofuels and bioproducts. Current Opinion in Green and Sustainable Chemistry, 2, 48–53.
Stern, J., Kahn, A., Vazana, Y., Shamshoum, M., Moraïs, S., Lamed, R. & Bayer, E. A. (2015). Significance of relative position of cellulases in designer cellulosomes for optimized cellulolysis. PLoS ONE, 10(5), 1–17.
Stern, J., Moraïs, S., Lamed, R. & Bayer, E. A. (2016). Adaptor scaffoldins: An original strategy for extended designer cellulosomes, inspired from nature. mBio, 7(2), 1–10.
Sun, S., Sun, S., Cao, X. & Sun, R. (2016). The role of pretreatment in improving the enzymatic hydrolysis of lignocellulosic materials. Bioresource Technology, 199, 49–58.
Takeda, T., Furuta, Y., Awano, T., Mizuno, K., Mitsuishi, Y. & Hayashi, T. (2002). Suppression and acceleration of cell elongation by integration of xyloglucans in pea stem segments. Proceedings of the National Academy of Sciences of the United States of America, 99(13), 9055–9060.
Tang, H., Wang, J., Wang, S., Shen, Y., Petranovic, D., Hou, J. & Bao, X. (2018). Efficient yeast surface-display of novel complex synthetic cellulosomes. Microbial Cell Factories, 17(1), 1–13.
TCI. Geraadpleegd op 1 december 2019 via https://www.tcichemicals.com/BE/nl/support-download/tcimail/application…;
Tsai, S. L., Goyal, G. & Chen, W. (2010). Surface display of a functional minicellulosome by intracellular complementation using a synthetic yeast consortium and its application to cellulose hydrolysis and ethanol production. Applied and Environmental Microbiology, 76(22), 7514–7520.
Tuazon, D. & Gnansounou, E. (2017). Towards an Integrated Sustainability Assessment of Biorefineries. In Gnansounou, E. & Pandey, A. (Eds.) Life-Cycle Assessment of Biorefineries (pp. 259–301). Elsevier B.V.
Vanderstraeten, J. (2016). Combining VersaTile Shuffeling with DSA-FACE for the high-troughput design of multicatalytic hemicellulases for xylanosomes. (Masterproef), Gent, Universitiet Gent.
Vazana, Y., Barak, Y., Unger, T., Peleg, Y., Shamshoum, M., Ben-Yehezkel, T., Mazor, Y.,
Shapiro, E., Lamed, R. & Bayer, E. A. (2013). A synthetic biology approach for evaluating the functional contribution of designer cellulosome components to deconstruction of cellulosic substrates. Biotechnology for Biofuels, 6(1), 1–18.
Wen, F., Sun, J. & Zhao, H. (2010). Yeast surface display of trifunctional minicellulosomes for simultaneous saccharification and fermentation of cellulose to ethanol. Applied and Environmental Microbiology, 76(4), 1251–1260.
Whilster, R. L. (1993). Hemicelluloses. In Whilster, R. L. & BeMiller, J. N. (Eds.) Industrial gums: polysaccharides and their derivatives third edition (pp. 295-309). California: Academic Press, Inc.
Xu, Q., Bayer, E. A., Goldman, M., Kenig, R., Shoham, Y. & Lamed, R. (2004). Architecture of the Bacteroides cellulosolvens Cellulosome: Description of a Cell Surface-Anchoring Scaffoldin and a Family 48 Cellulase. Journal of Bacteriology, 186(4), 968–977.
Xu, Q., Gao, W., Ding, S. Y., Kenig, R., Shoham, Y., Bayer, E. A. & Lamed, R. (2003). The cellulosome system of Acetivibrio cellulolyticus includes a novel type of adaptor protein and a cell surface anchoring protein. Journal of Bacteriology, 185(15), 4548–4557.
Yaniv, O., Morag, E., Borovok, I., Bayer, E. A., Lamed, R., Frolow, F. & Shimon, L. J. (2013). Structure of a family 3a carbohydrate-binding module from the cellulosomal scaffoldin CipA of Clostridium thermocellum with flanking linkers: implications for cellulosome structure. Acta crystallographica. Section F, Structural biology and crystallization communications, 69(Pt 7), 733–737.
You, C. & Zhang, Y. H. P. (2014). Annexation of a high-activity enzyme in a synthetic three-enzyme complex greatly decreases the degree of substrate channeling. ACS Synthetic Biology, 3(6), 380–386.