Hartverwanten in de medische wereld: wat computers ons kunnen bijbrengen over de fouten in ons hart.
Het koppig hart
Het hart is zowaar de koppigste spier van ons lichaam; ze weigert stelselmatig stil te vallen. Zelfs als alle andere spieren uitklokken voor een nachtrust, klopt het hart door. Ingenieurs noemen het hart wel eens “de beste pomp die er bestaat”. Ongeveer elke seconde trekken de atria en ventrikels samen en pompen ze bloed door de rest van het lijf. Dit doen ze door middel van een onwaarschijnlijk complexe chemisch-elektrische interactie van verscheidene ionen. Deze interacties geven aanleiding tot een golfverschijnsel die zich over heel het hart manoeuvreert. Eerst beweegt de golf over de atria, dan wacht die even tussenin, om vervolgens naar de ventrikels te propageren. Waar de golf passeert, daar krijgen je hartspiercellen het signaal om samen te trekken. Die ionen zijn tot op de milliseconde op elkaar ingespeeld. Zelfs al gaat er iets fout, dan heeft je hart tal van failsafe mechanismen om te voorkomen dat het niet verder zou misgaan. Zo kunnen hartspiercellen bijvoorbeeld niet samentrekken vlak nadat er een golf gepasseerd is, en hebben ze even tijd nodig om tot rust te komen. Dit voorkomt dat een golf kan blijven dwalen doorheen de hartspiercellen, wat zou leiden tot een en onophoudelijke samentrekking, of een ongewenst ritme.
Ondanks de inherente regelmaat van het hart, loopt het toch soms mis. Het go-signaal, die de samentrekkingsgolf ontketent, kan vertrekken uit een foute locatie. Dan is het niet meer de regelmatige sinusnode die het ritme dirigeert, maar een onvoorspelbaar klompje spiercellen, die compleet naast de maat slaan. Deze heten focale slagen.
Als de hartspiercellen een te korte rustpauze inlassen, kan de samentrekkingsgolf zich een weg blijven banen doorheen de spiermuren. Dit kan roteren rond zichzelf als een tornado, of rond littekenweefsel of anatomische objecten zoals de longaderen die naar je atria leiden. Deze stoornis heet rotationele activiteit.
Dergelijke problemen kunnen onschuldig zijn, maar kunnen zich ook ontwikkelen tot levensbedreigende aandoeningen. Een samentrekkingsgolf die zich een weg blijft banen doorheen de spiercellen kan evolueren tot pure chaos: een patroon dat niet eens meer op een golf lijkt. De bijhorende samentrekking is even chaotisch: het hart trilt er op los, of valt volledig stil.
Meten doet weten
Een accurate manier vinden om te achterhalen waar en wanneer dergelijke defecten zich voordoen redt tal van mensenlevens. Elektrocardiogrammen (ECGs) zijn veruit de meest populaire manier om de elektrische golf over het hart te meten. Ze vergen geen intrusieve operatie en kunnen gemeten worden tijdens een kort doktersbezoek.
Wat je wint in gebruiksgemak, verlies je helaas in informatie. Vragen zoals “Waar roteert de elektrische golf?” en “Waar bevat het hart littekenweefsel?” zijn allesbehalve evident om te beantwoorden met behulp van enkel ECGs. Dat heeft onderzoekers niet tegengehouden om het alsnog te proberen, uiteraard.
Om het elektrisch hart in kaart te brengen bestaat er nog een methode, genaamd mapping. Hierbij wordt een katheder tegen de hartwand gehouden. Deze onthoudt niet alleen de locatie van de hartwand, maar meet ook het passerende elektrisch signaal. De katheder wordt dan over de wand van één of meer kamers verplaatst tot die volledig in kaart gebracht zijn. Deze methode is echter veel meer intrusief, tijdrovend en kostelijk. Ze wordt daarom pas toegepast geval van nood, of gecombineerd met een al geplande hartoperatie. Het voordeel? De data, die deze methode produceert, is veel informatiever over hoe de elektrische golf over het hart beweegt. Plaatsen van golfrotatie vallen al snel op, net zoals hartcellen die ongewenste signalen uitsturen.
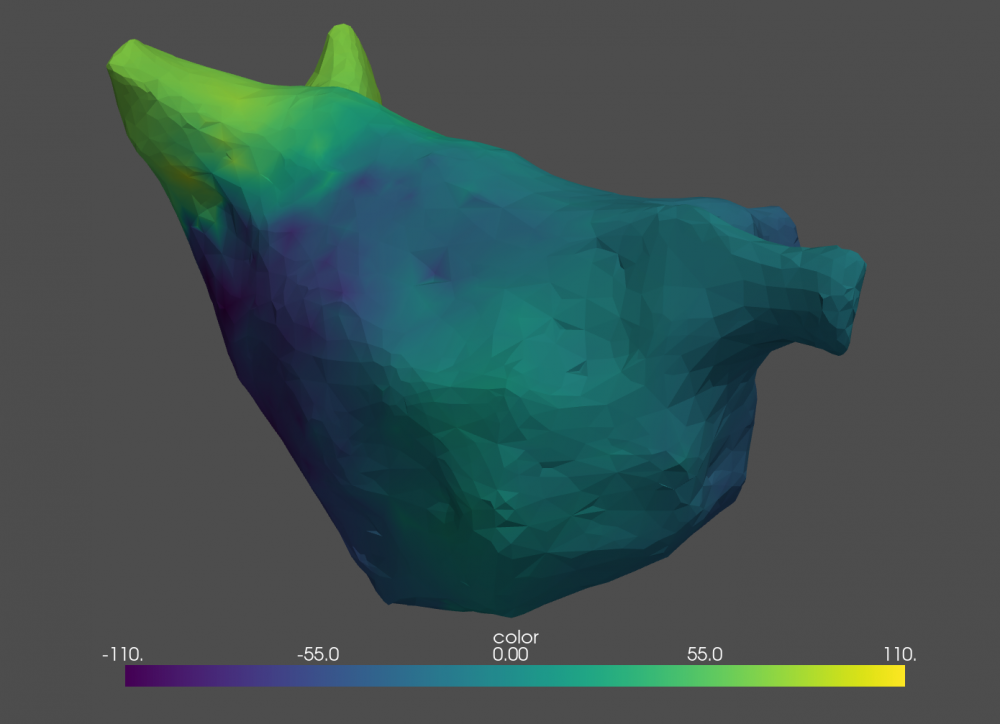
Beide methodes hebben hun voor- en nadelen. Een gemeenschappelijk nadeel is dat de data niet altijd een sluitend antwoord kan bieden. ECGs en mapping data interpreteren vergt expertise. Niet elke interpretatie zal stuiten op consensus: complexe stoornissen vallen nu eenmaal niet gemakkelijk te identificeren, laat staan verhelpen.
Het computerbrein
De complexiteit van het probleem heeft al tal van methodes aangetrokken. Eén methode die al veel succes geboekt heeft, is het gebruik van machine learning en artificiële intelligentie om ECGs te interpreteren. Machine learning is een ongeëvenaarde kampioen als het gaat om linken vinden in complexe systemen en multidimensionele data. Een goed getraind computerbrein kan dingen zien waar de mens zich blind op staart. Impressionante modellen kunnen met ECGs al tal van hartritmestoornissen identificeren, lokaliseren, en zelfs voorspellen alvorens ze plaatsvinden. Ondanks deze successen, is het gebruik van machine learning nog niet uitgebreid naar toepassingen op mapping data.
Met dit werk werden de eerste stappen gezet om de successen van machine learning op klinische data uit te breiden. De eerste stap bestaat erin om exuberante hoeveelheden data te produceren. Hoe meer, hoe beter. Bij voorkeur geven we onszelf of onze naasten geen levensbedreigende hartaandoeningen for sience. Het beste plan is dan om ze te simuleren. Op basis van bestaande mapping data werden 25 modellen van het linkeratrium gereconstrueerd. Deze werden gebruikt in een totaal van 4383 simulaties van twee types hartritmestoornissen: focale slagen en rotationele activiteit.
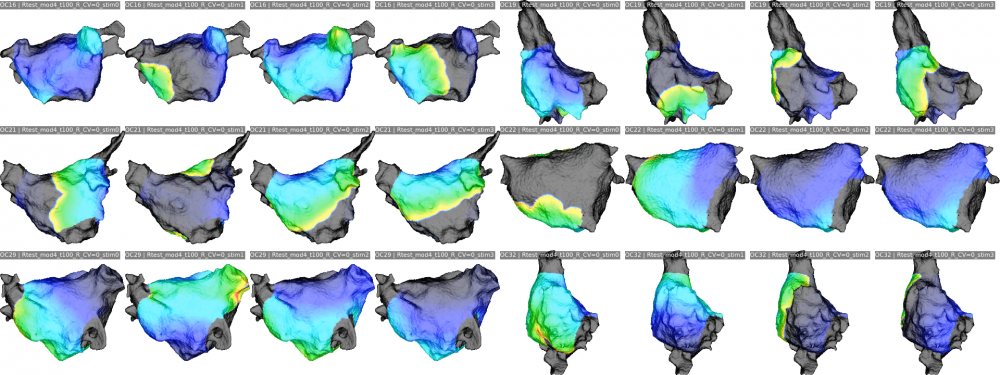
Deze simulaties werden verwerkt en gebruikt om twee dingen te proberen met machine learning. Ten eerste: kan machine learning een onderscheid maken tussen de twee stoornissen? Het antwoord blijkt van wel, en zelfs met zeer hoge accuraatheid. Dit probleem is echter niet één van de complexe situaties die niet door mensen kan geïnterpreteerd worden. Het menselijk oog zou het in dit geval zelfs $0.0007\%$ beter doen dan de machine learning modellen.
Ten tweede: kan machine learning ontdekken waar er zich rotationele activiteit bevindt? Dit probleem is al stukken interessanter. De accuraatheid van de machine learning modellen impliceert dat het mogelijk kan zijn, maar er nog veel werk aan de winkel is alvorens machine learning een waardevolle bijdrage kan leveren bij de analyse van mapping data.
Verder onderzoek zou een waardevolle bijdrage kunnen leveren aan een accurate classificatie van hartritmestoornissen en diens onderliggende oorzaken. Zo draagt machine learning bij tot kostbare inzichten om het koppig hart nog jaren verder te helpen kloppen.
Bibliografie
[1] John E. Hall. Guyton and Hall textbook of medical physiology. Elsevier, 13
edition, 2016.
[2] Elaine Nicpon Marieb and Katja Hoen. Human anatomy & physiology. Pear-
son Education, 2013.
[3] M. Courtemanche, R. J. Ramirez, and S. Nattel. Ionic mechanisms underlying
human atrial action potential properties: insights from a mathematical model.
Am J Physiol, 275(1):H301–321, 07 1998.
[4] S. Dokos, B. Celler, and N. Lovell. Ion currents underlying sinoatrial node
pacemaker activity: a new single cell mathematical model. J Theor Biol,
181(3):245–272, Aug 1996.
[5] Y. Kurata, I. Hisatome, S. Imanishi, and T. Shibamoto. Dynamical descrip-
tion of sinoatrial node pacemaking: improved mathematical model for pri-
mary pacemaker cell. Am J Physiol Heart Circ Physiol, 283(5):H2074–2101,
Nov 2002.
[6] G. D. Veenhuyzen, S. Knecht, M. D. O’Neill, D. Phil, M. Wright, I. Nault,
R. Weerasooriya, W. Rukshen, S. Miyazaki, F. Sacher, M. Hocini, P. Jaïs, and
M. Haïssaguerre. Atrial tachycardias encountered during and after catheter
ablation for atrial fibrillation: part I: classification, incidence, management.
Pacing Clin Electrophysiol, 32(3):393–398, Mar 2009.
[7] U. R. Anoop and K. Verma. The ECG made easy for the dental practitioner.
Indian J Dent Res, 25(3):386–389, 2014.
[8] A. K. Sangaiah, M. Arumugam, and G. B. Bian. An intelligent learning
approach for improving ECG signal classification and arrhythmia analysis.
Artif Intell Med, 103:101788, 03 2020.
[9] Gerald A. Serwer and Ira Shetty. 18 - pediatric pacing and defibrillator use. In
Kenneth A. Ellenbogen, G. Neal Kay, Chu-Pak Lau, and Bruce L. Wilkoff, ed-
itors, Clinical Cardiac Pacing, Defibrillation and Resynchronization Therapy
(Fourth Edition), pages 393–427. W.B. Saunders, Philadelpia, fourth edition
edition, 2011.
[10] Sok-Sithikun Bun, Tahar Delassi, Decebal Gabriel Latcu, Mohammed El
Jamili, Anis Ayari, Abdelkarim Errahmouni, Benjamin Berte, and Nadir
Saoudi. A comparison between multipolar mapping and conventional mapping
of atrial tachycardias in the context of atrial fibrillation ablation. Archives of
Cardiovascular Diseases, 111(1):33–40, 2018.
[11] A. L. HODGKIN and A. F. HUXLEY. A quantitative description of mem-
brane current and its application to conduction and excitation in nerve. J
Physiol, 117(4):500–544, Aug 1952.
[12] D. NOBLE. A modification of the Hodgkin–Huxley equations applicable to
Purkinje fibre action and pace-maker potentials. J Physiol, 160:317–352, Feb
1962.
[13] G. Plank, A. Loewe, A. Neic, C. Augustin, Y. L. Huang, M. A. F. Gsell,
E. Karabelas, M. Nothstein, A. J. Prassl, J. Sánchez, G. Seemann, and E. J.
Vigmond. The openCARP simulation environment for cardiac electrophysi-
ology. Comput Methods Programs Biomed, 208:106223, Jun 2021.
[14] Nele Vandersickel, Enid Van Nieuwenhuyse, Nico Van Cleemput, Jan Goedge-
beur, Milad El Haddad, Jan De Neve, Anthony Demolder, Teresa Strisciuglio,
Mattias Duytschaever, and Alexander V. Panfilov. Directed networks as a
novel way to describe and analyze cardiac excitation: Directed graph map-
ping. Frontiers in Physiology, 10:1138, 2019.
[15] Alison Stewart. Slco1b1 polymorphisms and statin-induced myopathy. PLoS
currents, 5, December 2013.
[16] J. E. Poole and D. Wilber. Outcomes That Matter: Assessing Benefits of
Atrial Fibrillation Catheter Ablation. JACC Clin Electrophysiol, 5(3):340–
342, 03 2019.
[17] J. E. Poole and D. Wilber. Outcomes That Matter: Assessing Benefits of
Atrial Fibrillation Catheter Ablation. JACC Clin Electrophysiol, 5(3):340–
342, 03 2019.
[18] A. J. Camm, G. Y. Lip, R. De Caterina, I. Savelieva, D. Atar, S. H. Hohn-
loser, G. Hindricks, P. Kirchhof, J. J. Bax, H. Baumgartner, C. Ceconi,
V. Dean, C. Deaton, R. Fagard, C. Funck-Bretano, D. Hasdai, A. Hoes,
P. Kirchhof, J. Knuuti, P. Kolh, T. McDonagh, C. Moulin, B. A. Popescu,
Ž. Reiner, U. Sechtem, P. A. Sirnes, M. Tendera, A. Torbicki, A. Vahanian,
S. Windecker, P. Vardas, N. Al-Attar, O. Alfierei, A. Angelini, C. Blömstrom-
Lundqvist, P. Colonna, J. De Sutter, S. Ernst, A. Goette, B. Gorenek,
R. Hatala, H. Heidbüchel, M. Heldal, S. Dalby Kristensen, P. Kolh, J. Y.
Le Heuzey, H. Mavrakis, L. Mont, P. Perrone Filardi, P. Ponikowski, B. Pren-
dergast, F. H. Rutten, U. Schotten, I. C. Van Gelder, and F. W. Verheugt.
2012 focused update of the ESC Guidelines for the management of atrial fib-
rillation: an update of the 2010 ESC Guidelines for the management of atrial
fibrillation. Developed with the special contribution of the European Heart
Rhythm Association. Eur Heart J, 33(21):2719–2747, Nov 2012.
[19] Igor Matias, Nuno Garcia, Sandeep Pirbhulal, Virginie Felizardo, Nuno
Pombo, Henriques Zacarias, Miguel Sousa, and Eftim Zdravevski. Predic-
tion of atrial fibrillation using artificial intelligence on electrocardiograms: A
systematic review. Computer Science Review, 39:100334, 2021.
[20] G Luongo, S Schuler, M W Rivolta, O Doessel, R Sassi, and A Loewe. 236Au-
tomatic classification of 20 different types of atrial tachycardia using 12-lead
ECG signals. EP Europace, 22(Supplement 1), 06 2020. euaa162.048.
[21] Julie K. Shade, Ashish N. Doshi, Eric Sung, Dan M. Popescu, Anum S.
Minhas, Nisha A. Gilotra, Konstantinos N. Aronis, Allison G. Hays, and
Natalia A. Trayanova. Covid-heart: Development and validation of a multi-
variable model for real-time prediction of cardiovascular complications in hos-
pitalized patients with covid-19. medRxiv, 2021.
[22] Giorgio Luongo, Luca Azzolin, Massimo W Rivolta, Tiago P Almeida,
Juan Pablo Martínez, Diogo C Soriano, Olaf Dössel, Roberto Sassi, Pablo
Laguna, and Axel Loewe. Machine learning to find areas of rotors sustaining
atrial fibrillation from the ecg. In 2020 Computing in Cardiology, pages 1–4,
2020.
[23] Buntheng Ly, Sonny Finsterbach, Marta Nuñez-Garcia, Hubert Cochet, and
Maxime Sermesant. Scar-related ventricular arrhythmia prediction from
imaging using explainable deep learning. In Daniel B. Ennis, Luigi E. Perotti,
and Vicky Y. Wang, editors, Functional Imaging and Modeling of the Heart,
pages 461–470, Cham, 2021. Springer International Publishing.
[24] M. Clerx, J. Heijman, P. Collins, and P. G. A. Volders. Predicting changes
to INa from missense mutations in human SCN5A. Sci Rep, 8(1):12797, 08
2018.
[25] B. Li and W. J. Gallin. Computational identification of residues that modulate
voltage sensitivity of voltage-gated potassium channels. BMC Struct Biol,
5:16, Aug 2005.
[26] S. Ramasubramanian and Y. Rudy. The Structural Basis of IKs Ion-Channel
Activation: Mechanistic Insights from Molecular Simulations. Biophys J,
114(11):2584–2594, 06 2018.
[27] B. A. Lawson, K. Burrage, P. Burrage, C. C. Drovandi, and A. Bueno-Orovio.
Slow Recovery of Excitability Increases Ventricular Fibrillation Risk as Iden-
tified by Emulation. Front Physiol, 9:1114, 2018.
[28] R. Grosu, E. Bartocci, F. Corradini, E. Entcheva, S. A. Smolka, and
A. Wasilewska. Learning and detecting emergent behavior in networks of
cardiac myocytes. In Magnus Egerstedt and Bud Mishra, editors, Hybrid
Systems: Computation and Control, pages 229–243, Berlin, Heidelberg, 2008.
Springer Berlin Heidelberg.
[29] Mahesh Kumar Mulimani, Jaya Kumar Alageshan, and Rahul Pandit. Deep-
learning-assisted detection and termination of spiral and broken-spiral waves
in mathematical models for cardiac tissue. Physical Review Research, 2(2),
May 2020.
[30] Cesare Corrado, Steven Williams, Caroline Roney, Gernot Plank, Mark
O’Neill, and Steven Niederer. Using machine learning to identify local cellu-
lar properties that support re-entrant activation in patient-specific models of
atrial fibrillation. EP Europace, 23(Supplement_1):i12–i20, 01 2021.
[31] N. A. Trayanova, D. M. Popescu, and J. K. Shade. Machine Learning in
Arrhythmia and Electrophysiology. Circ Res, 128(4):544–566, Feb 2021.
[32] Ana Maria Sánchez de la Nava, Felipe Atienza, Javier Bermejo, and Fran-
cisco Fernández-Avilés. Artificial intelligence for a personalized diagnosis and
treatment of atrial fibrillation. American Journal of Physiology-Heart and
Circulatory Physiology, 2021.
[33] Cesare Corrado, Steven Williams, Caroline Roney, Gernot Plank, Mark
O’Neill, and Steven Niederer. Using machine learning to identify local cellu-
lar properties that support re-entrant activation in patient-specific models of
atrial fibrillation. EP Europace, 2021.
[34] Hang Si. Tetgen, a delaunay-based quality tetrahedral mesh generator. ACM
Trans. Math. Softw., 41(2), February 2015.
[35] K. K. Aras, N. R. Faye, B. Cathey, and I. R. Efimov. Critical Volume of
Human Myocardium Necessary to Maintain Ventricular Fibrillation. Circ
Arrhythm Electrophysiol, 11(11):e006692, 11 2018.
[36] S. Verheule, J. Eckstein, D. Linz, B. Maesen, E. Bidar, A. Gharaviri, and
U. Schotten. Role of endo-epicardial dissociation of electrical activity and
transmural conduction in the development of persistent atrial fibrillation. Prog
Biophys Mol Biol, 115(2-3):173–185, Aug 2014.
[37] Charles D. Hansen and Chris R. Johnson.
Elsevier-Butterworth Heinemann, 2005.
The visualization handbook.
[38] Paolo Cignoni, Marco Callieri, Massimiliano Corsini, Matteo Dellepiane,
Fabio Ganovelli, and Guido Ranzuglia. MeshLab: an Open-Source Mesh
Processing Tool. In Vittorio Scarano, Rosario De Chiara, and Ugo Erra, edi-
tors, Eurographics Italian Chapter Conference. The Eurographics Association,
2008.
[39] Martin W. Krueger, Viktor Schmidt, Catalina Tobón, Frank M. Weber, Cris-
tian Lorenz, David U. J. Keller, Hans Barschdorf, Michael Burdumy, Pe-
ter Neher, Gernot Plank, Kawal Rhode, Gunnar Seemann, Damien Sanchez-
Quintana, Javier Saiz, Reza Razavi, and Olaf Dössel. Modeling atrial fiber
orientation in patient-specific geometries: A semi-automatic rule-based ap-
proach. In Dimitris N. Metaxas and Leon Axel, editors, Functional Imaging
and Modeling of the Heart, pages 223–232, Berlin, Heidelberg, 2011. Springer
Berlin Heidelberg.
[40] T. E. Fastl, C. Tobon-Gomez, A. Crozier, J. Whitaker, R. Rajani, K. P.
McCarthy, D. Sanchez-Quintana, S. Y. Ho, M. D. O’Neill, G. Plank, M. J.
Bishop, and S. A. Niederer. Personalized computational modeling of left atrial
geometry and transmural myofiber architecture. Med Image Anal, 47:180–190,
07 2018.
[41] Byounghyun Lim, Minki Hwang, Jun-Seop Song, Ah-Jin Ryu, Boyoung
Joung, Eun Bo Shim, Hyungon Ryu, and Hui-Nam Pak. Effectiveness of
atrial fibrillation rotor ablation is dependent on conduction velocity: An in-
silico 3-dimensional modeling study. PLOS ONE, 12(12):1–16, 12 2017.
[42] T. E. Fastl, C. Tobon-Gomez, A. Crozier, J. Whitaker, R. Rajani, K. P.
McCarthy, D. Sanchez-Quintana, S. Y. Ho, M. D. O’Neill, G. Plank, M. J.
Bishop, and S. A. Niederer. Personalized computational modeling of left atrial
geometry and transmural myofiber architecture. Med Image Anal, 47:180–190,
07 2018.
[43] D. Cozma, B. A. Popescu, D. Lighezan, P. Lucian, C. Mornos, C. Ginghina,
and S. I. Dragulescu. Left atrial remodeling: assessment of size and shape to
detect vulnerability to atrial fibrillation. Pacing Clin Electrophysiol, 30 Suppl
1:S147–150, Jan 2007.
[44] S. Y. Ho, J. A. Cabrera, and D. Sanchez-Quintana. Left atrial anatomy
revisited. Circ Arrhythm Electrophysiol, 5(1):220–228, Feb 2012.
[45] C. Bane Sullivan and Alexander A. Kaszynski. Pyvista: 3d plotting and
mesh analysis through a streamlined interface for the visualization toolkit
(vtk). Journal of Open Source Software, 4(37):1450, 2019.
[46] Marco Attene. A lightweight approach to repairing digitized polygon meshes.
The Visual Computer, 26:1393–1406, 2010.
[47] Paolo Cignoni, Marco Callieri, Massimiliano Corsini, Matteo Dellepiane,
Fabio Ganovelli, and Guido Ranzuglia. MeshLab: an Open-Source Mesh
Processing Tool. In Vittorio Scarano, Rosario De Chiara, and Ugo Erra, edi-
tors, Eurographics Italian Chapter Conference. The Eurographics Association,
2008.