Wat hebben de ziekte van Alzheimer en depressie gemeen?
In mijn masterproef heb ik gezocht naar overeenkomsten en verschillen tussen de ziekte van Alzheimer en depressie. Vraagt u zich af waarom deze schijnbaar totaal verschillende ziektes samen werden onderzocht? Lees dan snel verder!
De ziekte van Alzheimer is de meest gekende vorm van dementie. Verschillende factoren hebben een invloed op de onderliggende biologie en de kans op Alzheimer. Hierbij is ouderdom op zich één van de belangrijkste risicofactoren. Depressie, langs de andere kant, is een veel voorkomende en waarschijnlijk de meest gekende psychiatrische ziekte. Depressie kan zich op veel verschillende manieren uiten. Over de jaren heen zijn verschillende hypotheses naar buiten gebracht over de biologische oorzaak van depressie, maar ook hier is er consensus over een multifactoriële aanleg. Bovendien lijdt een groot deel van de patiënten met Alzheimer ook aan depressie. De laatste jaren is er toenemende aandacht voor neuro-inflammatie in verschillende psychiatrische en neurodegeneratieve ziektes. Bij neuro-inflammatie is er een lage graad van ontsteking in de hersenen, wat een langdurig en nefast effect kan hebben op het functioneren van het brein.
De ziekte van Alzheimer en depressie hebben beide een enorme impact op veel mensen, zowel op de patiënten als hun omgeving. Bovendien is er voor beide aandoeningen momenteel geen medicatie beschikbaar die voor iedereen werkt. Verder stijgt de prevalentie door de veeleisende maatschappij van vandaag en de toenemende vergrijzing. Een belangrijke reden voor het tekort aan medicatie is dat de onderliggende biologie van zowel Alzheimer als depressie nog steeds onvoldoende gekend is. Verder onderzoek blijft dus essentieel. Het wordt echter steeds duidelijker dat er een overlap is in de biologie van verschillende psychiatrische en neurodegeneratieve ziektes zoals depressie en Alzheimer, wat impliceert dat deze aandoeningen beter samen bestudeerd worden en niet elk apart. Dit zou de zoektocht naar gemeenschappelijke behandelingen kunnen bevorderen.
Hoe heb ik dit probleem aangepakt?
In mijn masterproef zocht ik naar gemeenschappelijke en uiteenlopende signaalwegen en hun regulatoren bij Alzheimer en depressie. Een signaalweg is een kettingreactie in de cel van verschillende biomoleculen, waardoor een bepaald proces in gang wordt gezet. Hiervoor werden computationele methoden gebruikt die op basis van beschikbare data voorspellingen maken over biologische signalisatie en interacties. Met deze data werden netwerken gebouwd van verschillende genen die een rol spelen in de ziekte. Deze genen konden zowel regulatoren als hun doelgenen zijn. Regulatoren zijn belangrijke moleculen in onze cellen die mee bepalen welke genen actief worden en welke niet. Deze worden dus als regulatoren gezien van het cellulair netwerk. De doelgenen zijn de genen waarop deze regulatoren werken. Om deze netwerken te construeren werden drie verschillende algoritmen gebruikt. Verschillende methoden kunnen gebruikt worden om netweken te bouwen uit data. Echter, elk van deze methoden heeft zijn voor- en nadelen, waardoor het gunstig is om meerdere methoden te gebruiken. De resultaten van deze algoritmen werden dan samengevoegd tot één collectief netwerk om robuustere resultaten te verkrijgen. Dit werd gedaan voor beide ziektes. Daarnaast werden ook meer precieze data gebruikt, afkomstig van individuele cellen (single-cell) van de hersenen. Hiermee werden ook netwerken geconstrueerd. Zo was het mogelijk om de celtypes die belangrijk zijn voor de ziektes beter te bestuderen. De meest gekende cellen van de hersenen zijn neuronen. Daarnaast zijn er nog andere celtypes, zoals microglia, de immuuncellen van het brein.
De netwerken van de twee ziektes werden dan met elkaar vergeleken om gemeenschappelijke en verschillende signaalwegen en hun regulatoren te vinden. Hiervoor werden clusters van genen, die onder dezelfde omstandigheden actief worden, onderzocht. De functies van de genen binnen eenzelfde cluster werden verder geanalyseerd. Verschillende clusters werden gevonden waar de genen immunologische functies uitvoeren, in zowel Alzheimer als depressie. Het immuunsysteem beschermt normaliter ons lichaam tegen vreemde stoffen. Echter, als het immuunsysteem verkeerdelijk geactiveerd wordt, zorgt dit voor negatieve consequenties en schade in het lichaam. Hoewel in beide ziektes immunologische signaalwegen werden teruggevonden, waren deze niet allemaal identiek aan elkaar. Dit wijst erop dat er slechts een gedeeltelijke overlap is. De regulatoren van deze clusters werden dan verder bestudeerd. Van de meeste regulatoren was reeds geweten dat ze een rol spelen in het immuunsysteem en van enkele was geweten dat ze van belang zijn in microglia. Daarnaast werden er gemeenschappelijke regulatoren gevonden die immunologische clusters in zowel Alzheimer als depressie sturen. Deze waren IKZF1, IRF8, NFATC2, RUNX1 en TAL1.
Deze vijf regulatoren werden verder onderzocht in de netwerken geconstrueerd van de single-cell data. Hier werd gedetecteerd dat deze regulatoren en hun doelgenen allemaal tot expressie komen in de microglia, zowel in Alzheimer als depressie. Deze vijf regulatoren sturen tevens elkaar (zie figuur 1). Eerder onderzoek toonde aan dat alle vijf regulatoren een rol spelen in de biologie van Alzheimer, terwijl enkel TAL1 tot nu toe met depressie geassocieerd is geweest. NFATC2 en IRF8 zorgen voor de activatie van microglia, wat tot neuro-inflammatie kan leiden.
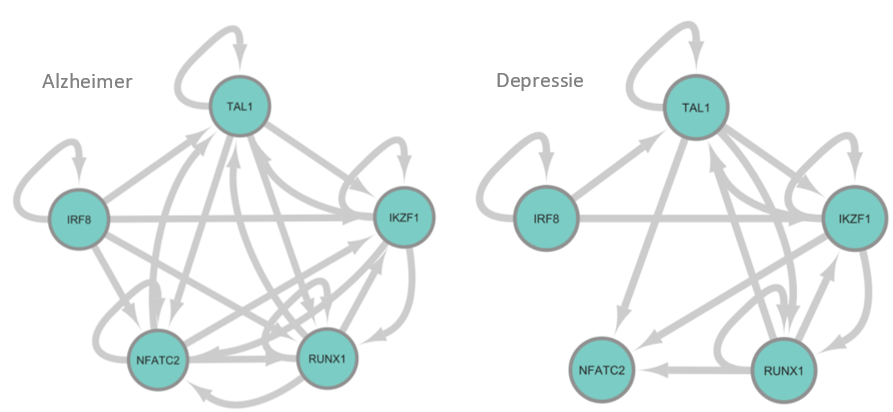 Figuur 1. Netwerken van gemeenschappelijke regulatoren in microglia, betrokken bij immunologische functies. In de blauwe bollen zijn de regulatoren afgebeeld, de pijlen stellen regulatie voor. Links: Alzheimer netwerk, rechts: depressie netwerk, .
Figuur 1. Netwerken van gemeenschappelijke regulatoren in microglia, betrokken bij immunologische functies. In de blauwe bollen zijn de regulatoren afgebeeld, de pijlen stellen regulatie voor. Links: Alzheimer netwerk, rechts: depressie netwerk, .
Naast de immunologische functies werden ook andere signaalwegen ontdekt die gemeenschappelijk zijn tussen de twee ziektes. Ten eerste werd een gestoorde werking van de mitochondria gevonden, wat reeds gekend was in beide ziektes. Dit zorgt ervoor dat de energieproductie van de cellen niet optimaal verloopt. Daarnaast werden genen gevonden die deel uitmaken van het proteasoom in beide netwerken. Het proteasoom is een complex van eiwitten in de cel dat betrokken is bij de afbraak en recyclage van andere eiwitten. Dit was ook reeds geassocieerd met Alzheimer en behandelings-resistente depressie.
Conclusie
Verschillende signaalwegen werden gevonden die ontregeld zijn in Alzheimer en depressie. Er is activatie van microglia, mitochondriale dysfunctie en betrokkenheid van het proteasoom. Verschillende regulatoren werden gevonden die betrokken zijn in de activatie van microglia en disregulatie van het immuunsysteem in beide ziektes. Toekomstig onderzoek is essentieel om hun functie in de biologie van deze ziektes verder te ontrafelen. Daarnaast kunnen deze regulatoren ook gebruikt worden in de zoektocht naar nieuwe medicatie. Biologische netwerken zijn dus een ideale tool om nieuwe invalspunten te vinden voor verschillende ziekten tegelijk.
Bibliografie
1. Pape, K., Tamouza, R., Leboyer, M. & Zipp, F. Immunoneuropsychiatry — novel perspectives on
brain disorders. Nature Reviews Neurology 15, 317–328 (2019).
2. Chew, G. & Petretto, E. Transcriptional Networks of Microglia in Alzheimer’s Disease and Insights
into Pathogenesis. Genes (Basel) 10, (2019).
3. Schwartz, M. & Deczkowska, A. Neurological Disease as a Failure of Brain-Immune Crosstalk: The
Multiple Faces of Neuroinflammation. Trends Immunol 37, 668–679 (2016).
4. Wang, Q., Jie, W., Liu, J. H., Yang, J. M. & Gao, T. M. An astroglial basis of major depressive
disorder? An overview. Glia 65, 1227–1250 (2017).
5. Caruso Bavisotto, C. et al. Extracellular Vesicle-Mediated Cell−Cell Communication in the Nervous
System: Focus on Neurological Diseases. Int J Mol Sci 20, (2019).
6. Hansen, D. V., Hanson, J. E. & Sheng, M. Microglia in Alzheimer’s disease. J Cell Biol 217, 459–472
(2018).
7. De Virgilio, A. et al. Parkinson’s disease: Autoimmunity and neuroinflammation. Autoimmunity
Reviews 15, 1005–11 (2016).
8. Pitsillou, E. et al. The cellular and molecular basis of major depressive disorder: towards a unified
model for understanding clinical depression. Mol Biol Rep 47, 753–770 (2020).
9. Dietz, A. G., Goldman, S. A. & Nedergaard, M. Glial cells in schizophrenia: a unified hypothesis.
Lancet Psychiatry 7, 272–281 (2020).
10. Benedetti, F., Aggio, V., Pratesi, M. L., Greco, G. & Furlan, R. Neuroinflammation in Bipolar
Depression. Front Psychiatry 11, 71 (2020).
11. Huang, Y. et al. Integrated multifactor analysis explores core dysfunctional modules in autism
spectrum disorder. Int J Biol Sci 14, 811–818 (2018).
12. Masi, A., DeMayo, M. M., Glozier, N. & Guastella, A. J. An Overview of Autism Spectrum
Disorder, Heterogeneity and Treatment Options. Neurosci Bull 33, 183–193 (2017).
13. Dobson, R. & Giovannoni, G. Multiple sclerosis - a review. Eur J Neurol 26, 27–40 (2019).
14. Soria Lopez, J. A., González, H. M. & Léger, G. C. Alzheimer’s disease. Handb Clin Neurol 167,
231–255 (2019).
15. Lane, C. A., Hardy, J. & Schott, J. M. Alzheimer’s disease. Eur J Neurol 25, 59–70 (2018).
16. Del Tredici, K. & Braak, H. Idiopathic Parkinson’s Disease: Staging an α-Synucleinopathy with
a Predictable Pathoanatomy. Molecular Mechanisms in Parkinson’s Disease (2000).
17. Troutman, T. D., Kofman, E. & Glass, C. K. Exploiting dynamic enhancer landscapes to decode
macrophage and microglia phenotypes in health and disease. Mol Cell 81, 3888–3903 (2021).
18. Liddelow, S. A. et al. Neurotoxic reactive astrocytes are induced by activated microglia. Nature
541, 481–487 (2017).
19. Liu, C. S., Adibfar, A., Herrmann, N., Gallagher, D. & Lanctôt, K. L. Evidence for Inflammation-
Associated Depression. in Inflammation-Associated Depression: Evidence, Mechanisms and
Implications (eds. Dantzer, R. & Capuron, L.) 3–30 (Springer International Publishing, 2017).
doi:10.1007/7854_2016_2.
20. Boku, S., Nakagawa, S., Toda, H. & Hishimoto, A. Neural basis of major depressive disorder:
Beyond monoamine hypothesis. Psychiatry Clin Neurosci 72, 3–12 (2018).
21. Zhang, Q. et al. Kynurenine regulates NLRP2 inflammasome in astrocytes and its implications
in depression. Brain Behav Immun 88, 471–481 (2020).
22. Drevets, W. C., Wittenberg, G. M., Bullmore, E. T. & Manji, H. K. Immune targets for therapeutic
development in depression: towards precision medicine. Nat Rev Drug Discov 1–21 (2022)
doi:10.1038/s41573-021-00368-1.
23. Saint-Antoine, M. M. & Singh, A. Network inference in systems biology: recent developments,
challenges, and applications. Current Opinion in Biotechnology 63, 89–98 (2020).
24. Banf, M. & Rhee, S. Y. Computational inference of gene regulatory networks: Approaches,
limitations and opportunities. Biochimica et Biophysica Acta - Gene Regulatory Mechanisms 1860,
41–52 (2017).
25. Yan, J., Risacher, S. L., Shen, L. & Saykin, A. J. Network approaches to systems biology
analysis of complex disease: integrative methods for multi-omics data. Briefings in bioinformatics 19,
1370–1381 (2018).
26. Roy, S. et al. Integrated Module and Gene-Specific Regulatory Inference Implicates Upstream
Signaling Networks. PLoS Computational Biology 9, (2013).
27. Blondel, V. D., Guillaume, J.-L., Lambiotte, R. & Lefebvre, E. Fast unfolding of communities in
large networks. J. Stat. Mech. 2008, P10008 (2008).
28. Pearl, J. R. et al. Genome-Scale Transcriptional Regulatory Network Models of Psychiatric and
Neurodegenerative Disorders. Cell Syst 8, 122-135.e7 (2019).
29. Marbach, D. et al. Wisdom of crowds for robust gene network inference. Nat Methods 9, 796–
804 (2012).
30. Faith, J. J. et al. Large-Scale Mapping and Validation of Escherichia coli Transcriptional
Regulation from a Compendium of Expression Profiles. PLOS Biology 5, e8 (2007).
31. Nguyen, H., Tran, D., Tran, B., Pehlivan, B. & Nguyen, T. A comprehensive survey of regulatory
network inference methods using single-cell RNA sequencing data. Briefings in bioinformatics (2020)
doi:10.1093/bib/bbaa190.
32. Huynh-Thu, V. A., Irrthum, A., Wehenkel, L. & Geurts, P. Inferring Regulatory Networks from
Expression Data Using Tree-Based Methods. PLoS One 5, e12776 (2010).
33. Ghosh Roy, G., Geard, N., Verspoor, K. & He, S. PoLoBag: Polynomial Lasso Bagging for
signed gene regulatory network inference from expression data. Bioinformatics 36, 5187–5193
(2021).
34. Bonnet, E., Calzone, L. & Michoel, T. Integrative Multi-omics Module Network Inference with
Lemon-Tree. PLOS Computational Biology 11, e1003983 (2015).
35. Siahpirani, A. F. & Roy, S. A prior-based integrative framework for functional transcriptional
regulatory network inference. Nucleic Acids Res 45, e21 (2017).
36. Erola, P., Bonnet, E. & Michoel, T. Learning Differential Module Networks Across Multiple
Experimental Conditions. in Gene Regulatory Networks: Methods and Protocols (eds. Sanguinetti,
G. & Huynh-Thu, V. A.) 303–321 (Springer, 2019). doi:10.1007/978-1-4939-8882-2_13.
37. Iglesias-Martinez, L. F., De Kegel, B. & Kolch, W. KBoost: a new method to infer gene regulatory
networks from gene expression data. Sci Rep 11, 15461 (2021).
38. Pratapa, A., Jalihal, A. P., Law, J. N., Bharadwaj, A. & Murali, T. M. Benchmarking algorithms
for gene regulatory network inference from single-cell transcriptomic data. Nat Methods 17, 147–154
(2020).
39. Aibar, S. et al. SCENIC: single-cell regulatory network inference and clustering. Nat Methods
14, 1083–1086 (2017).
40. Friedman, J. H. Stochastic gradient boosting. Computational Statistics & Data Analysis 38, 367–
378 (2002).
41. Tantardini, M., Ieva, F., Tajoli, L. & Piccardi, C. Comparing methods for comparing networks.
Scientific reports 9, 17557 (2019).
42. Koutrouli, M., Karatzas, E., Paez-Espino, D. & Pavlopoulos, G. A. A Guide to Conquer the
Biological Network Era Using Graph Theory. Frontiers in Bioengineering and Biotechnology 8, 34
(2020).
43. Yaveroğlu, Ö. N. et al. Revealing the Hidden Language of Complex Networks. Scientific reports
4, 4547 (2014).
44. Gandal, M. J. et al. Shared molecular neuropathology across major psychiatric disorders
parallels polygenic overlap. Science 359, 693–697 (2018).
45. Sadeghi, I. et al. Brain transcriptomic profiling reveals common alterations across
neurodegenerative and psychiatric disorders. bioRxiv 2021.08.16.456345 (2021)
doi:10.1101/2021.08.16.456345.
46. Godini, R., Fallahi, H. & Ebrahimie, E. A comparative system-level analysis of the
neurodegenerative diseases. Journal of Cellular Physiology 234, 5215–5229 (2019).
47. Vermeirssen, V., De Clercq, I., Van Parys, T., Van Breusegem, F. & Van de Peer, Y. Arabidopsis
Ensemble Reverse-Engineered Gene Regulatory Network Discloses Interconnected Transcription
Factors in Oxidative Stress. The Plant Cell 26, 4656 (2014).
48. Defoort, J., Van de Peer, Y. & Vermeirssen, V. Function, dynamics and evolution of network
motif modules in integrated gene regulatory networks of worm and plant. Nucleic Acids Res 46, 6480–
6503 (2018).
49. Robinson, M. D. & Oshlack, A. A scaling normalization method for differential expression
analysis of RNA-seq data. Genome Biology 11, R25 (2010).
50. Lovering, R. C. et al. A GO catalogue of human DNA-binding transcription factors. Biochimica
et Biophysica Acta (BBA) - Gene Regulatory Mechanisms 194765 (2021)
doi:10.1016/j.bbagrm.2021.194765.
51. Meyer, P. E., Lafitte, F. & Bontempi, G. minet: A R/Bioconductor Package for Inferring Large
Transcriptional Networks Using Mutual Information. BMC Bioinformatics 9, 461 (2008).
52. Marbach, D. et al. Tissue-specific regulatory circuits reveal variable modular perturbations
across complex diseases. Nat Methods 13, 366–370 (2016).
53. Alvarez, M. J. et al. Functional characterization of somatic mutations in cancer using network-
based inference of protein activity. Nat Genet 48, 838–847 (2016).
54. Shannon, P. et al. Cytoscape: a software environment for integrated models of biomolecular
interaction networks. Genome Res 13, 2498–504 (2003).
55. Stuart, T. et al. Comprehensive Integration of Single-Cell Data. Cell 177, 1888-1902.e21 (2019).
56. Morabito, S. et al. Single-nucleus chromatin accessibility and transcriptomic characterization of
Alzheimer’s disease. Nature genetics 53, 1143–1155 (2021).
57. Pantazatos, S. P. et al. Whole-transcriptome brain expression and exon-usage profiling in major
depression and suicide: evidence for altered glial, endothelial and ATPase activity. Molecular
Psychiatry 22, 760–773 (2017).
58. Ramaker, R. C. et al. Post-mortem molecular profiling of three psychiatric disorders. Genome
Medicine 9, 72 (2017).
59. Lu, Y., Zhou, X. & Nardini, C. Dissection of the Module Networks Implementation “LemonTree”:
Enhancements towards Applications in Metagenomics and Translation in Autoimmune Maladies.
Mol. BioSyst. 13, (2017).
60. The UniProt Consortium. UniProt: the universal protein knowledgebase in 2021. Nucleic Acids
Research 49, D480–D489 (2021).
61. Schwartzentruber, J. et al. Genome-wide meta-analysis, fine-mapping and integrative
prioritization implicate new Alzheimer’s disease risk genes. Nature Genetics 53, 392–402 (2021).
62. Zeng, Q. et al. IRF-8 is Involved in Amyloid-β1-40 (Aβ1-40)-induced Microglial Activation: a New
Implication in Alzheimer’s Disease. Journal of Molecular Neuroscience 63, 159–164 (2017).
63. Samaan, Z. et al. Obesity genes and risk of major depressive disorder in a multiethnic
population: a cross-sectional study. The Journal of Clinical Psychiatry 76, e1611-1618 (2015).
64. Rasheed, M. et al. A Systematic Review of Circulatory microRNAs in Major Depressive
Disorder: Potential Biomarkers for Disease Prognosis. International Journal of Molecular Sciences
23, 1294 (2022).
65. Piñero, J. et al. The DisGeNET knowledge platform for disease genomics: 2019 update. Nucleic
Acids Research 48, D845–D855 (2020).
66. Katayama, H. Anti-interleukin-17A and anti-interleukin-23 antibodies may be effective against
Alzheimer’s disease: Role of neutrophils in the pathogenesis. Brain Behav 10, e01504 (2020).
67. Saelens, W., Cannoodt, R. & Saeys, Y. A comprehensive evaluation of module detection
methods for gene expression data. Nat Commun 9, 1090 (2018).
68. Rosenthal, S. L., Barmada, M. M., Wang, X., Demirci, F. Y. & Kamboh, M. I. Connecting the
Dots: Potential of Data Integration to Identify Regulatory SNPs in Late-Onset Alzheimer’s Disease
GWAS Findings. PLOS ONE 9, e95152 (2014).
69. Manocha, G. D. et al. NFATc2 Modulates Microglial Activation in the AβPP/PS1 Mouse Model
of Alzheimer’s Disease. J Alzheimers Dis 58, 775–787 (2017).
70. Kimura, R. et al. The DYRK1A gene, encoded in chromosome 21 Down syndrome critical
region, bridges between β-amyloid production and tau phosphorylation in Alzheimer disease. Human
Molecular Genetics 16, 15–23 (2007).
71. Lobo-Silva, D., Carriche, G. M., Castro, A. G., Roque, S. & Saraiva, M. Balancing the immune
response in the brain: IL-10 and its regulation. Journal of Neuroinflammation 13, 297 (2016).
72. Nagy, C. et al. Single-nucleus transcriptomics of the prefrontal cortex in major depressive
disorder implicates oligodendrocyte precursor cells and excitatory neurons. Nat Neurosci 23, 771–
781 (2020).
73. Ma, J., Jiang, T., Tan, L. & Yu, J.-T. TYROBP in Alzheimer’s disease. Mol Neurobiol 51, 820–
826 (2015).
74. Minelli, A. et al. Proteasome system dysregulation and treatment resistance mechanisms in
major depressive disorder. Transl Psychiatry 5, e687 (2015).
75. Lutz, M. W., Sprague, D., Barrera, J. & Chiba-Falek, O. Shared genetic etiology underlying
Alzheimer’s disease and major depressive disorder. Translational Psychiatry 10, (2020).
76. Bellenguez, C. et al. New insights into the genetic etiology of Alzheimer’s disease and related
dementias. Nat Genet 1–25 (2022) doi:10.1038/s41588-022-01024-z.
77. Jin, T. et al. scGRNom: a computational pipeline of integrative multi-omics analyses for
predicting cell-type disease genes and regulatory networks. Genome Med 13, 95 (2021).
78. Dong, X., Liu, C. & Dozmorov, M. Review of multi-omics data resources and integrative analysis
for human brain disorders. Brief Funct Genomics 20, 223–234 (2021).









